
论文题目:InstructProtein: Aligning Human and Protein Language via Knowledge Instruction
本文作者:王泽元(浙江大学)、张强(浙江大学)、丁科炎(浙江大学)、秦铭(浙江大学)、庄祥(浙江大学)、李晓彤(浙江大学)、陈华钧(浙江大学)
发表会议:ACL 2024
论文链接:https://arxiv.org/abs/2310.03269
代码链接:https://github.com/HICAI-ZJU/InstructProtein
欢迎转载,转载请注明出处

摘要
方法
方法
因此,在构建指令数据集时,我们不能简单地依赖现有的大型语言模型。我们需要采用其他策略或方法来确保数据集中各类子细胞器位置的合理代表性,从而让模型能够更加公平和准确地学习和预测这些位置。这可能包括人为介入以平衡数据集、使用合成数据来增强稀有类别的表示,或者开发新的模型架构和训练方法来更好地处理此类不均衡数据。
知识指令
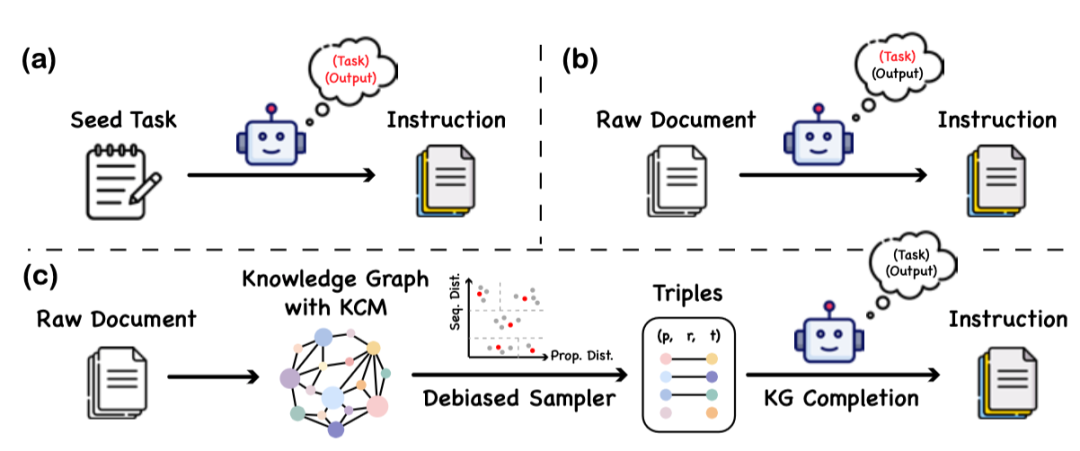
本文提出了一种名为“知识指令”的数据构建方法,这一方法通过利用知识图谱和大语言模型的协同工作,以构建一个平衡且多样化的指令数据集。该方法的核心在于,它不依赖于大语言模型对蛋白质语言的理解能力,从而避免了因模型偏误或幻觉而引入的虚假信息。
具体的构建过程分为三个主要阶段:
知识图谱的构建: 文章选用了蛋白质信息库UniProtKB作为主要数据源。由于UniProtKB中的数据本身具有很强的结构性,所以可以较为容易地将其转换为知识图谱的格式。 为了在指令数据中能有效反映不同蛋白质之间的逻辑关系,文章在知识图谱中添加了三元组之间的因果关系。这种设计有助于增强数据的逻辑性和实用性。 从知识图谱中抽取蛋白质-文本对: 为了保证数据集的多样性和平衡性,文章采用了一种基于蛋白质聚类的方法。在此过程中,首先计算蛋白质之间的序列和性质相似度,并依据这些相似度将蛋白质分组。 序列相似性是通过比较两个序列的一致性来评估的。性质相似度则是根据知识图谱中的嵌入计算得出,具体方法是比较两个蛋白质嵌入向量的距离。 指令数据的生成: 最后一步是将选择的蛋白质-文本三元组转换成实用的指令数据。这一转换过程中,文章利用了现有的大语言模型以及知识图谱中的信息。 利用大型语言模型根据知识图谱补全任务提供的模板,将抽取的信息转化为具体的指令,确保生成的指令既准确又具有指导意义。
通过这种方法,可以在不依赖预设模型理解蛋白质语言的前提下,有效地创建出一个既丰富又平衡的蛋白质函数和位置的指令数据集,为后续的蛋白质功能研究和应用提供更可靠的数据支持。
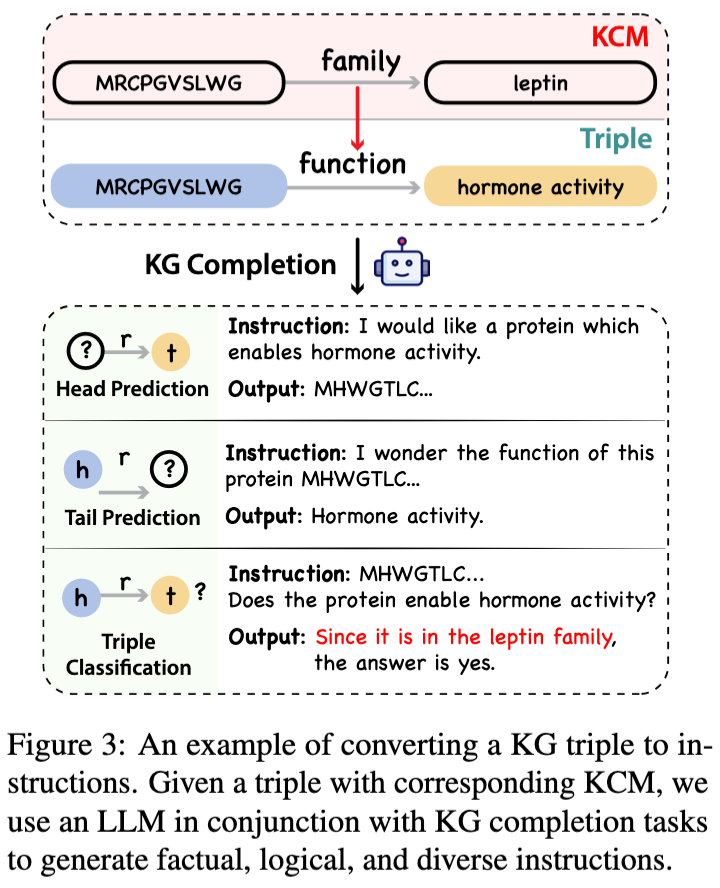
在本文提出的知识指令数据集构建方法中,训练过程分为两个阶段:多语言预训练和指令微调。在多语言预训练阶段,本文利用了生物相关的大型文本数据库来增强模型在生物领域的语言理解和知识背景。多语言指的是处理自然语言(如英文摘要)和生物序列语言(如蛋白质序列)的能力。文章使用了使用了UniRef100和PubMed中的摘要作为语料。指令微调阶段的目标是优化模型以理解和遵循针对特定生物学任务的指令。这个阶段利用从UniProt/Swiss-Prot数据库中构建的指令数据集对模型进行微调训练。通过预训练和微调的组合,得到的模型——称为InstructProtein——能够更好地执行涉及蛋白质序列的各种预测和注释任务,比如准确预测蛋白质的功能或者定位到特定的亚细胞位置。这对蛋白质工程、药物发现以及更广泛的生物医学研究可能具有重要意义。
实验
实验
本文通过全面的实验评估了大语言模型InstructProtein在蛋白质序列理解和设计方面的能力。研究覆盖了多个方面,包括蛋白质分类任务、蛋白质设计任务,以及消融实验,旨在了解不同因素对模型性能的影响。
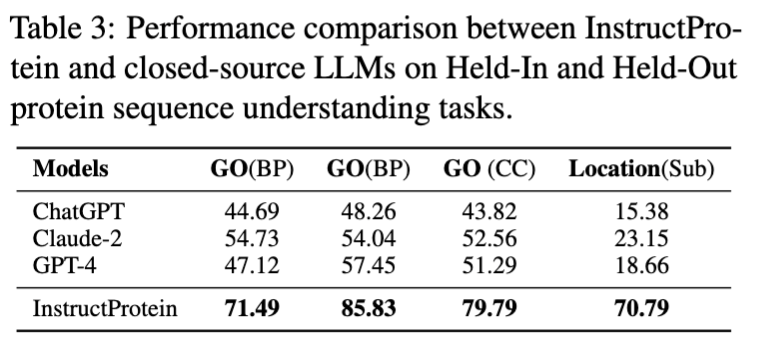
在蛋白质理解方面,本文评估了模型在以下三个分类任务上的性能:蛋白质位置预测、蛋白质功能预测 (基于Gene Ontology分类)、蛋白质金属离子结合能力预测。这些任务被设计成类似于自然语言中的阅读理解问题,其中每条数据包含一条蛋白质序列及一个问题,模型需回答是/否类型的问题。
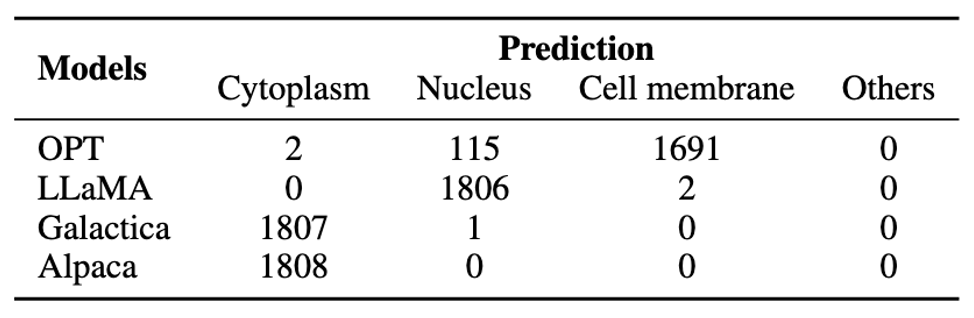
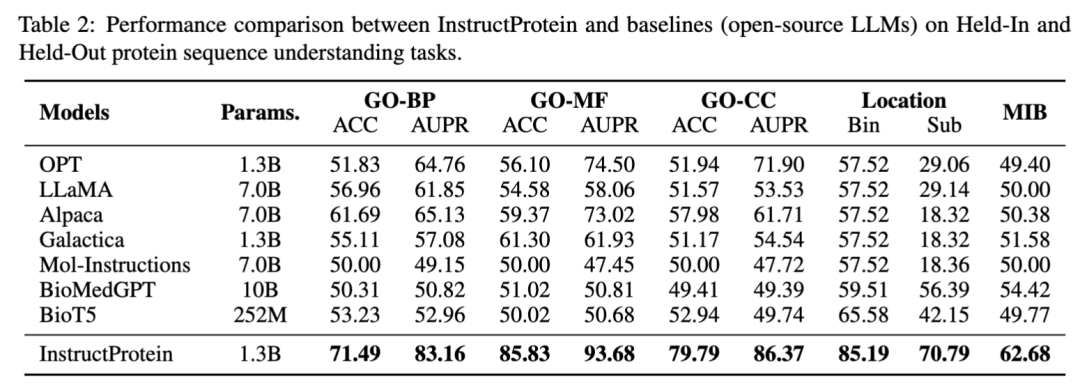
在蛋白质序列理解和分类任务上,InstructProtein展示了优异的表现,超越了所有基线模型。研究指出,将蛋白质和自然语言结合的训练语料库有利于提高语言模型在蛋白质语言理解方面的能力。特别值得注意的是,InstructProtein在多种任务(包括指令数据集中见过的和没见过的任务)上显示出较强的泛化能力,而其他模型如Mol-Instructions因模板不足而未能有效理解任务需求。此外,封闭源模型(如ChatGPT等)也显示出在蛋白质位置预测任务中受到注释不平衡的影响,这进一步强调了优质指令数据集的重要性。值得注意的是,在蛋白质位置预测(Bin)任务中的奇怪结果是因为现存的大语言模型受到注释不平衡的影响将所有蛋白质分类为单一组
蛋白质设计
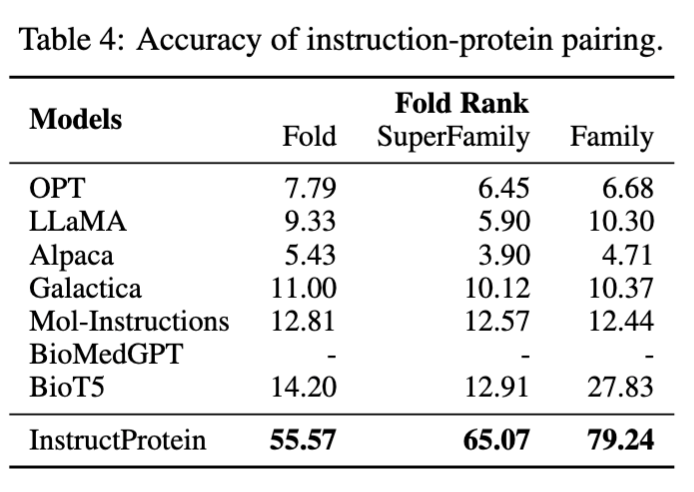
在蛋白质设计方面,文章设计了一个“指令蛋白配对”任务:给定一个蛋白质及其描述,模型需要从其对应描述及九个不对应的描述中选择最合适的一项。在指令-蛋白质配对任务中,InstructProtein显著超越了所有基线模型,展现出其在指令跟随和蛋白质设计方面的优越性。BioMedGPT因只专注于将蛋白质转换为文本而缺乏蛋白质设计能力。Galactica由于其训练数据集是以叙述性蛋白质语料为主,因此在零样本任务中与指令对齐的性能有限。Mol-Instructions没有在蛋白质语料上进行预训练,难以分辨蛋白质的细微差异,导致性能不佳。这些结果验证了我们模型在遵循设计指令方面的出色能力,强调了针对特定任务进行预训练和数据集优化的重要性,以提升模型在蛋白质设计方面的性能。
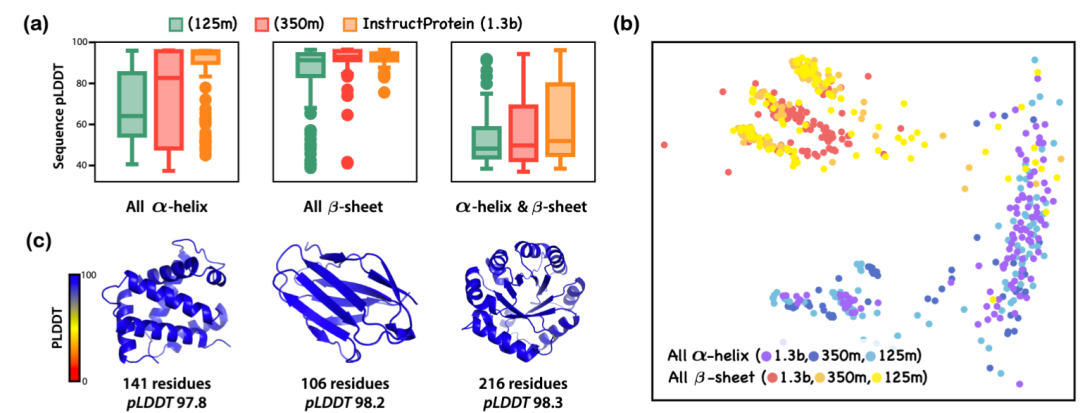
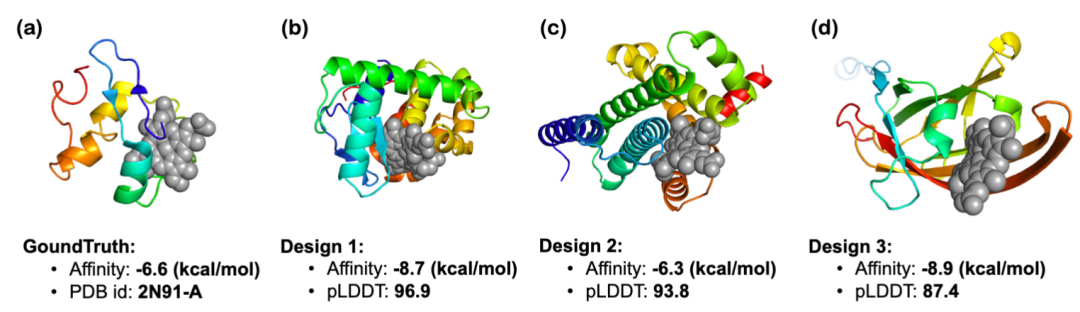
文章还探讨了结构相关指令和功能相关指令的蛋白质生成:InstructProtein 的研究展示了其在依据结构相关指令生成新蛋白质序列方面的有效性。通过使用 SCOPe 分类和 ColabFold 预测每个蛋白质序列的可折叠性,结果显示模型规模的增加能够产生更少内在无序区域的蛋白质。此外,通过 ESM2 和多维缩放算法对生成的全-螺旋和全-折叠蛋白质进行可视化,证明了模型遵循指令的能力。在功能相关的设计中,InstructProtein 成功设计了与血红素结合的蛋白质,其结合亲和力和结构预测评分均表明了良好的设计性能。结合 HHblits 对同源性的检查也显示了序列的新颖性。这些结果表明 InstructProtein 在基于自然语言的蛋白质设计中具有高效且创新的潜力。
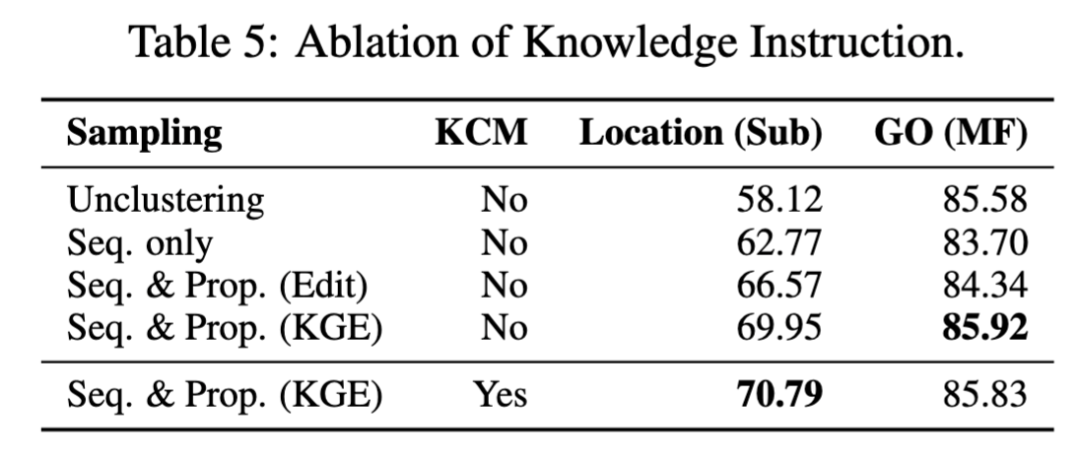
通过对数据集构建策略和知识因果建模进行的消融研究显示:在蛋白质位置预测等注释不平衡明显的任务中,相似蛋白质的聚类采样可以显著提高模型性能。在注释平衡的情况下(如GO任务),单纯基于序列的聚类可能降低了模型表现。通过同时考虑序列和性质的相似性可以避免这种下降。对于聚类方法,基于知识图谱嵌入距离的方法比编辑距离更能有效地捕捉性质相似性。引入的知识因果关系能够进一步提升模型性能。
总结
总结
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢