导读


分子的有效表示是影响人工智能模型性能的最关键因素之一。近日,湖南大学俞汝勤院士吴海龙教授团队在分子表征的研究中取得新进展。该研究介绍了一种可扩展的、基于片段的多尺度分子表示框架,称为t-SMILES(基于树的 SMILES),包括三种编码算法:TSSA、TSDY和TSID。t-SMILES系统仅引入了两个不需要配对的新符号,给1988年发表的最经典分子线性表示法SMILES引入了片段结构信息,同时缩短了SMILES描述符中由于需要配对的括号和数字所带来的长期依赖问题,并因其语法的简单性使得t-SMILES非常易于学习。该研究关注自然语言处理模型协助化学家进行分子空间探索时面临的最核心也是最基础的如何编码分子的问题,为AI辅助的分子建模提供了新的研究思路,助力分子“智造”。
t-SMILES是SMILES的超集,它使用SMILES而不是字典ID表示分子片段,通过广度优先算法遍历分子树,从而形成与经典SMILES相似的线性字符串表示。

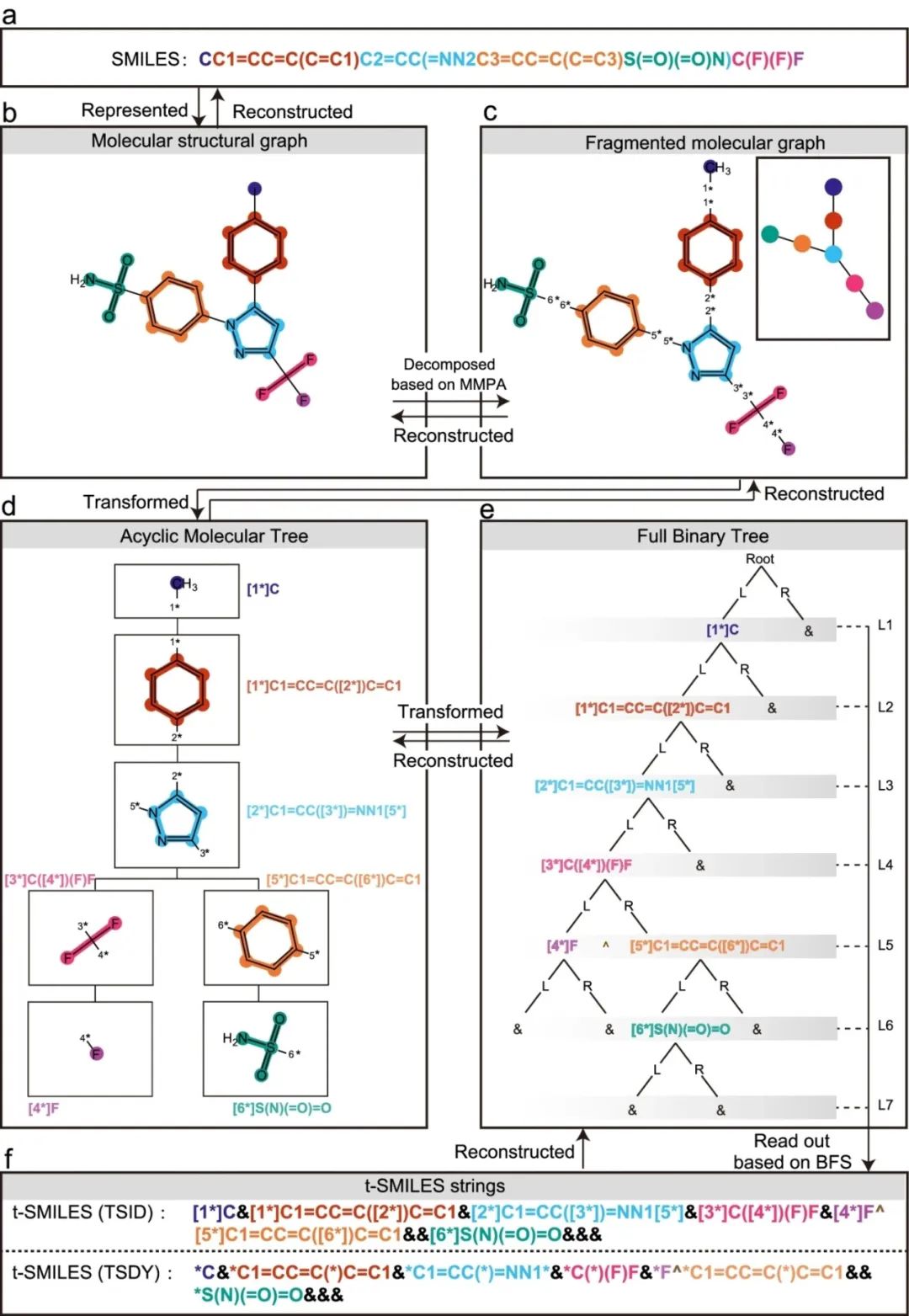
分子的t-SMILES获取路线图
(a. 塞来昔布分子的SMILES;b. 塞来昔布分子的结构式,不同颜色表明了其片段;c. 将分子打碎为碎片,不同碎片标记为不同颜色。附图表示碎片分子的整体拓扑结构;d.碎片分子的无环分子树(AMT),每个片段用SMILES编码;e.碎片分子的满二叉树(FBT)。树节点用片段或新引入的符号“&”表示。“L”和“R”指左子树或右子树。“L1”-“L7”指FBT的层数。在L5中,新符号“^”用于分隔t-SMILES字符串中的两部分。f.塞来昔布的TSID编码(带ID和虚拟原子的t-SMILES)和TSDY编码(带虚拟原子但没有ID的t-SMILES)。t-SMILES字符串中的颜色用于匹配结构式中的相应片段。首先使用选定的分子碎片算法分解分子图以构建AMT,然后将其转换为FBT。最后,使用BFS(广度优先搜索)算法遍历FBT并获取其t-SMILES字符串。要重构分子,需将t-SMILES字符串转为FBT,再将其转换为AMT,最后将AMT组装回原始分子。在TSID中,[n*]用于表示连接点。从TSID编码中删除ID后,可得到TSDY编码。新符号“&”用于标记空树节点。TSSA(具有共享原子的t-SMILES)使用不同的方式获取碎片。本图使用MMPA碎片化方法。)
据估计,类药化学空间中可探索的分子数高达1023-1060个,在如此巨大的化学空间中进行分子结构的智能生成和快速优化是分子设计面临的巨大挑战。系统评估表明,t-SMILES可以构建多编码混合的分子表示系统以探索更广泛的化学空间,其中各种编码相互补充,从而提高系统的整体性能。并且,基于t-SMILES的模型可以规避资源有限数据集上的过拟合问题,在保证生成的分子与训练集合理相似性的情况下具有更高的新颖性。
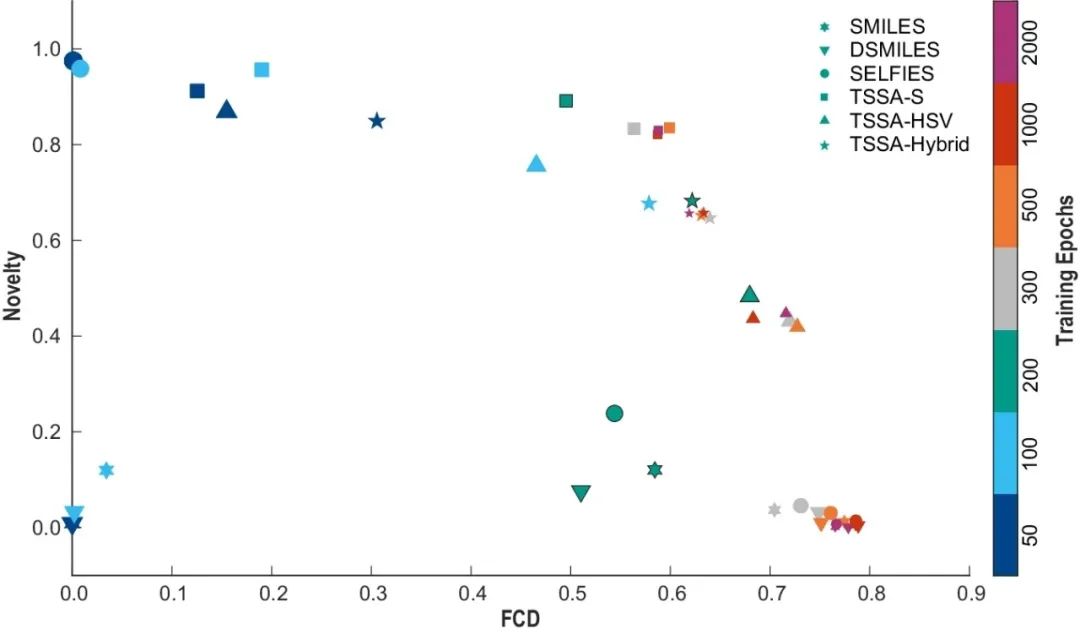
基于SMILES, DSMILES, SELFIES, TSSA_S, TSSA_HSV和 TSSA_Hybrid的模型分别在不同训练epochs的新颖性得分和FCD得分
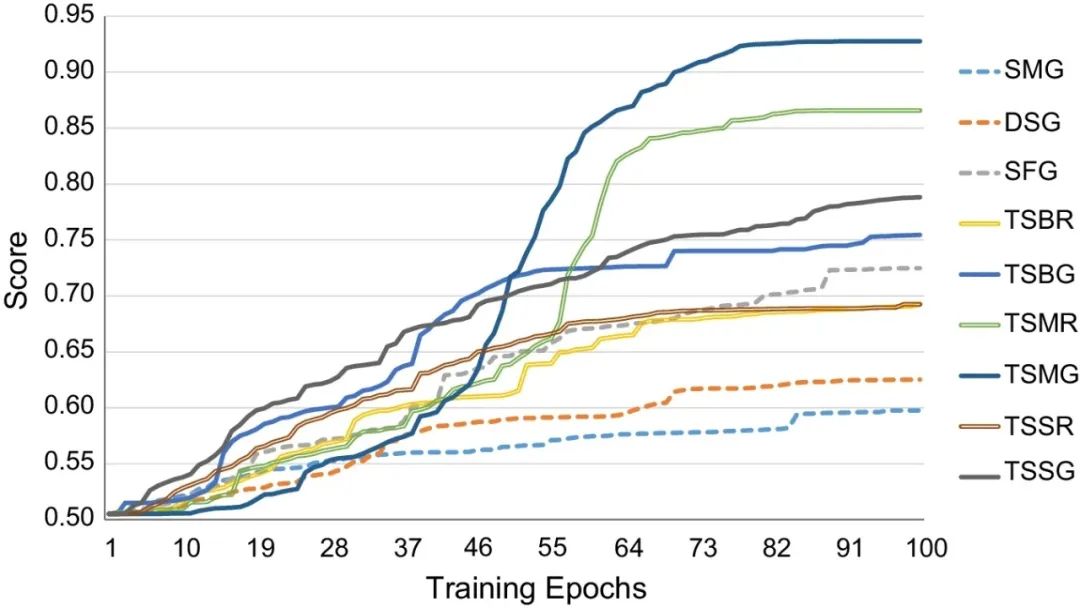
不同训练epochs下各种模型在目标导向分子生成任务(T16.SMPO)中的性能
结果与讨论

在使用先进的NLP方法解决化学问题时,会出现两个基本问题:(1)什么是“化学词汇”?(2)如何将它们编码为“化学句子”?
该研究引入了一个名为t-SMILES的框架来解决第二个问题。t-SMILES算法利用广度优先搜索(BFS)将碎片分子编码为字符串。不使用字典ID,而是使用经典的SMILES来描述碎片。这意味着,t-SMILES可以构建一个基于多尺度字符串的分子表示系统,简化计算复杂性并利用化学背景知识,因为分子结构本质上是局部相关的。
与经典SMILES相比t-SMILES仅引入了两个额外的符号“&”和“^”,以编码多尺度和层次化的分子拓扑结构。由于其相似性,优化t-SMILES模型以超越经典SMILES(尤其是TSDY和TSID)相对简单。
作者提出了三种编码算法:TSSA、TSDY和TSID。TSSA被推荐作为目标导向任务的首选,尤其是对于低资源任务,以避免与训练数据集“惊人相似”并实现“在合理的相似性下获得更好的新颖性”。TSID被推荐用于分布再现任务,因为它与训练数据的物理化学性质精确匹配。TSDY是目标导向和分布式再现任务的平衡选择。此外,还评估了随机和目标导向重建算法。后者在目标导向任务中的表现明显优于SOTA基线模型。此外,t-SMILES提供了更广泛的优势,尤其是对于低资源数据集,在平衡新颖性和FCD方面获得了更高的分数。
同时,t-SMILES框架能够将经典的SMILES统一为TS_Vanilla,使t-SMILES成为SMILES的超集,从而构建多种编码的分子描述系统。在现实世界的分子建模任务中,药物化学家通常面临具有挑战性的多目标优化问题,潜在的选择远远多于可以系统探索的范围。因此,建议使用混合t-SMILES方法探索尽可能多的化学空间,或针对特定任务使用单一t-SMILES模型。此外,t-SMILES使得在生成分子时可以生成有效且化学性质多样的片段,这可以作为其他基于片段的研究的基础。

理论上,t-SMILES算法能够支持任何有效的子结构类型和模式,只要它们能够用于获得化学上有效的分子片段。借助经验丰富的化学家对药物片段的宝贵积累,t-SMILES算法可以将这些经验与强大的基于序列的AI模型相结合,帮助化学家更好地探索所需化学空间。
此外,t-SMILES是一个开放且灵活的框架,使用基于树的算法对片段分子进行编码。如果使用SMILES对片段进行编码,会得到t-SMILES。如果使用DSMILES或SELFIES对片段进行编码,会得到t-DSMILES或t-SELFIES。
在某些特殊情况下,如果只需要探索特定的片段空间并且不需要扩展,t-SMILES算法可以用字典ID而不是SMILES对树节点进行编码。或者,可以根据给定的一组规则用来自训练数据的片段替换新生成的片段。在这些情况下,t-SMILES的编码逻辑和算法流程将保持不变。
6月11日,该研究成果以“t-SMILES: A Fragment-based Molecular Representation Framework for De Novo Ligand Design” 为题发表在《Nature Communications》上,湖南大学为独立完成单位,博士研究生伍娟妮为第一作者,俞汝勤院士和吴海龙教授为通讯作者。王童、陈悦、唐丽娟参与了本工作的研究。该工作得到了国家自然科学基金委、湖南大学化学化工学院、化学生物传感与计量学国家重点实验室支持。
论文链接:
https://www.nature.com/articles/s41467-024-49388-6


关注 点赞 收藏

内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢