DRUGAI
今天为大家介绍的是来自中国科学院大学Haicang Zhang团队的一篇论文。蛋白质序列设计对蛋白质工程至关重要。尽管深度学习方法取得了进展,但实现准确且稳定的序列设计仍然是一个挑战。在此,作者介绍了CarbonDesign,这是一种受AlphaFold成功元素启发并专门为蛋白质序列设计开发的方法。CarbonDesign的核心是引入了Inverseformer,它从主链结构中学习表示,并使用摊销马尔可夫随机场(amortized Markov random fields)模型进行序列解码。此外,CarbonDesign还整合了AlphaFold的其他关键概念:一种端到端的网络循环(recycling)技术,用于利用蛋白质语言模型中的进化约束,以及一种多任务学习技术,用于生成侧链结构和设计序列。CarbonDesign在独立测试集上的表现优于其他方法,比如CASP15数据集、CAMEO数据集以及来自RFDiffusion的de novo蛋白质。此外,它还支持序列变体功能效果的零样本预测,使其成为生物工程应用的有前途的工具。

蛋白质序列设计,也称为逆蛋白质折叠,是指确定能够折叠成给定蛋白质主链结构并具有所需功能的氨基酸序列。这是计算蛋白质设计中的关键步骤,最近在治疗剂、酶及其他应用的工程中取得了显著进展。通常在de novo蛋白质设计中,一旦通过基于能量的方法或最近的扩散生成模型获得了主链结构,确定最佳序列就变得至关重要。基于深度学习的序列设计方法在生成高度准确的候选序列方面展示了令人鼓舞的结果。这些方法在编码蛋白质结构和解码相关序列的策略上各不相同。通常,ProteinMPNN和ESM-IF利用神经网络对整个主链结构进行编码,然后以端到端的自回归方式解码序列。另一方面,3DCNN、ABACUS-R和ProDESIGN-LE等方法单独编码每个残基的结构背景,并从随机初始化的序列开始,逐步优化设计的序列。
蛋白质结构预测和蛋白质序列设计密切相关,一个领域的进步往往能促进另一个领域的发展。受到AlphaFold和RoseTTAFold在解决蛋白质折叠问题上取得显著成功的启发,作者将其关键概念应用于逆折叠,并提出了CarbonDesign,旨在通过改进编码器和解码器结构、利用更高效的特征以及优化训练策略来提升序列设计。
模型架构
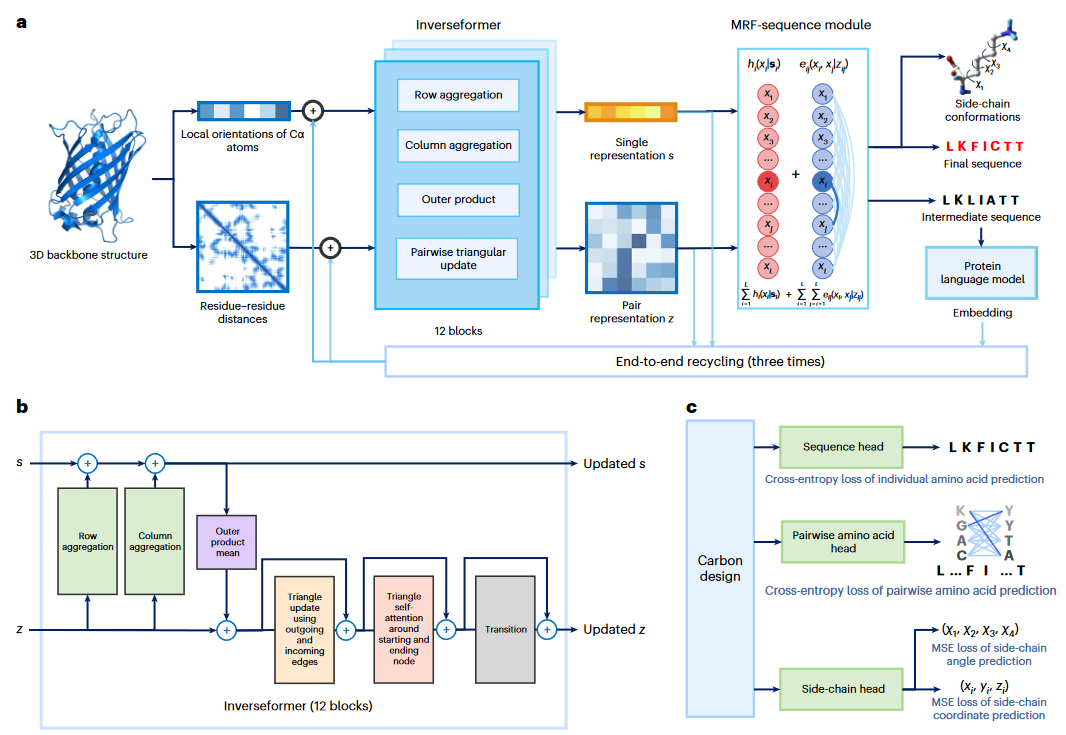
图1
CarbonDesign通过结合Inverseformer神经网络架构和基于进化及结构约束的训练程序来改进蛋白质序列设计。为了将蛋白质三维结构转换为一维序列,作者对AlphaFold中用于从一维序列预测三维结构的网络架构进行了逆转和调整(图1和表1)。
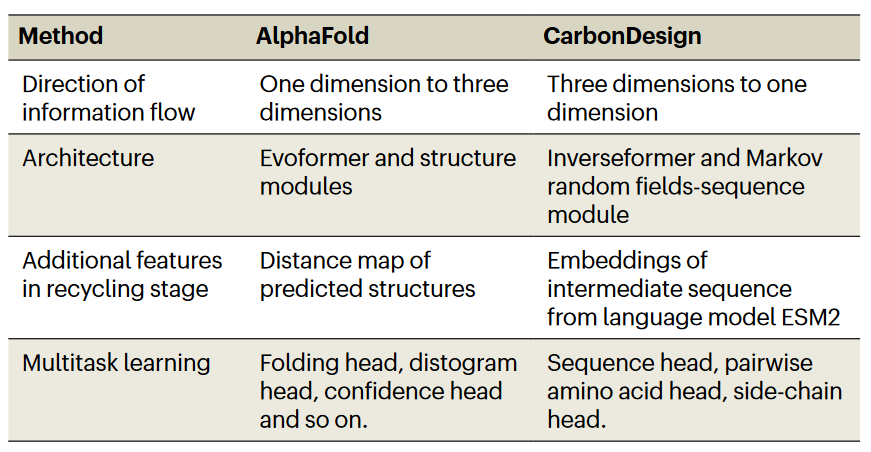
表1
这个网络包括两个主要阶段。首先,使用Inverseformer模块渐进式更新单一表征(single representation,后称single)和成对表征(pair representation,后称pair),它们是用局部方向和残基-残基距离初始化的。其次,使用马尔可夫随机场-序列模块来解码序列,其配对耦合项和位点偏置项分别基于学习到的配对和单一表征进行参数化。
Inverseformer旨在学习能够解码的单点和配对氨基酸的single和pair表示(图1b)。single和pair表示通过一系列模块进行交互和精炼。具体来说,single通过行聚合和列聚合层更新,以配对表示作为输入,实现信息从二维到一维的流动。随后,通过外积层和四个三角注意力层修正pair。
在蛋白质结构预测中,三角边更新的直观动机是为了满足残基-残基距离上的三角不等式约束。另一方面,对于序列设计,作者建立了Inverseformer的三角更新和信念传播(Belief Propagation,BP)算法中边信息更新之间的直观联系,BP算法常用于概率图模型如MRF和贝叶斯网络的学习和推理。在BP算法中,节点和边信息交替更新以聚合来自邻近变量节点的概率质量。每个边信息ij通过三角边更新操作进行更新,涉及与变量节点j相关的所有其他边信息jk。基于这种直觉,作者假设三角边更新鼓励在后续MRF-序列模块中生成在MRF模型下具有更高概率的序列表示。
MRF-序列模块为基于学习到的单一和配对表示的序列构建一个概率模型。MRF广泛用于直接耦合分析以建模序列概率。在CarbonDesign的上下文中,学习到的单一和配对表示自然地参数化了MRF中的耦合和位点偏置项。随后,使用一个简单的临时算法从MRF模型中采样候选氨基酸序列。
作者使用交叉熵损失来直接指导single和pair表示的学习,分别用于单个氨基酸和成对氨基酸的身份。此外,为了在MRF模型中近似序列的确切似然,作者在训练过程中使用了复合似然。作者还在训练中加入了侧链扭转角损失和侧链结构损失,使CarbonDesign能够预测序列及相应的侧链结构(图1c)。
CarbonDesign在独立测试集上的性能评估
作者在两个主要数据集上对CarbonDesign进行了广泛评估:CAMEO测试集和CASP15测试集。将作者的方法与蛋白质序列设计中的代表性方法进行了比较,包括ProteinMPNN、ESM-IF、ABACUS-R、Rosetta软件和ProDESIGN-LE。作者使用两个关键指标评估了CarbonDesign的性能:序列恢复率(sequence recovery)和BLOcks SUbstitution Matrix (BLOSUM)得分。序列恢复率评估模型设计的序列与目标结构的匹配程度,而BLOSUM得分衡量设计的序列与天然序列之间的相似性。
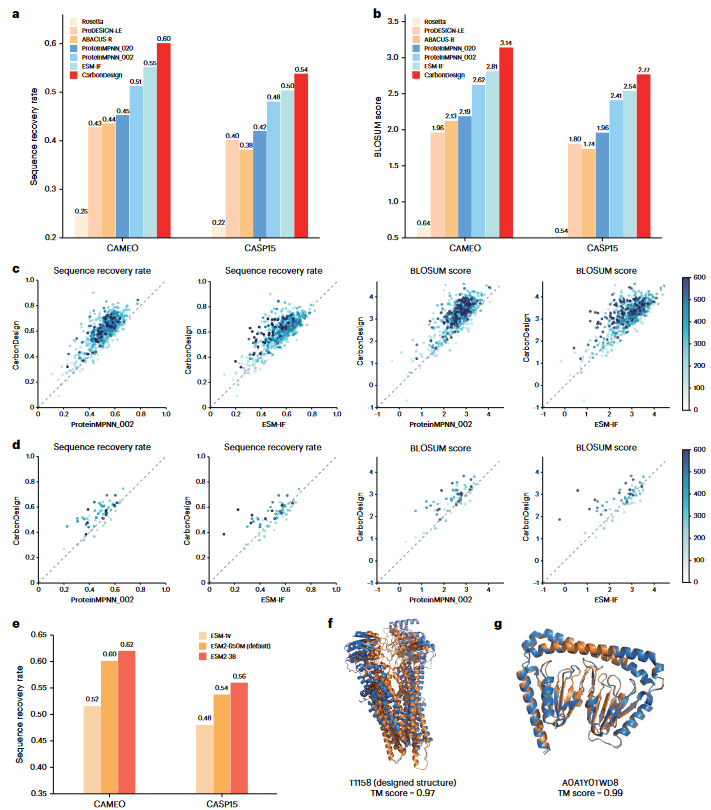
图2
在CAMEO和CASP数据集上,CarbonDesign的序列恢复率和BLOSUM得分指标均优于其他比较方法(图2a,b)。值得注意的是,使用更大的语言模型ESM-3B,序列设计的准确性进一步提高(图2e)。
作者进一步使用一组孤儿蛋白的数据集对CarbonDesign进行了评估。这些蛋白质由于缺乏同源序列和结构模板,对于现有的结构预测方法来说构成了重大挑战,因为它们缺乏进化信息。同时,这些蛋白质也为蛋白质序列设计提供了严格的测试集,因为它们在现有的序列和结构数据库中缺乏同源信息。在对CASP15中的孤儿蛋白进行评估时,CarbonDesign仍表现出强大的性能,达到49.1%的序列恢复率,优于所有其他代表性方法。
最近基于扩散的方法取得了进展,使得设计长的主链结构成为可能,这对蛋白质序列设计构成了挑战。作者从CASP15和CAMEO测试集中整理了一组长度超过800个氨基酸的长蛋白质数据集来评估CarbonDesign的性能。值得注意的是,CarbonDesign达到了55.1%的序列恢复率,超过了比较方法。作为一个例子,作者评估了CarbonDesign在多药耐药蛋白T1158(Bos taurus MRP4)上的表现,该蛋白长度为1,340个氨基酸(图2f)。在使用ESMFold预测的结构与天然结构进行比较时,CarbonDesign展示了58.1%的序列恢复率和0.97的模板建模(TM)得分。
作为一个案例研究,作者考察了双持NTPase(dwNTPase)(图2g),这是一种通过对AlphaFold数据库中预测结构的数据挖掘发现的高度新颖的架构。CarbonDesign成功生成了一个序列,具有70.2%的高序列恢复率。这个案例突显了CarbonDesign在预测主链结构上的稳健性及其强大的模型泛化能力,使其能够对新型折叠类型进行准确设计。
使用 CarbonDesign 改进de novo蛋白质设计
最近基于扩散的方法,如RFdiffusion,通过生成多种从未在自然界中观察到的新型主链结构,彻底改变了de novo蛋白质设计。鉴于这些进展,作者评估了CarbonDesign在通过为这些主链结构生成更准确的序列以增强de novo蛋白质设计的效果。由于没有天然序列来评估序列恢复率和BLOSUM相似性得分,作者采用了自一致TM(scTM)得分作为替代指标。具体来说,首先使用ESMFold预测与RFdiffusion生成的主链结构相对应的设计序列的结构,然后使用TM得分来衡量预测结构和原始结构之间的一致性。作者在训练过程中引入噪声到晶体结构中,类似于ProteinMPNN和ESM-IF的方法。这种方法考虑到在实际应用中,de novo生成的结构或预测结构可能不如用于训练的晶体结构精确。作者使用RFdiffusion生成了2,560个不同长度(从200到600个氨基酸)的主链结构,并在不同噪声水平下评估了CarbonDesign和ProteinMPNN的性能。
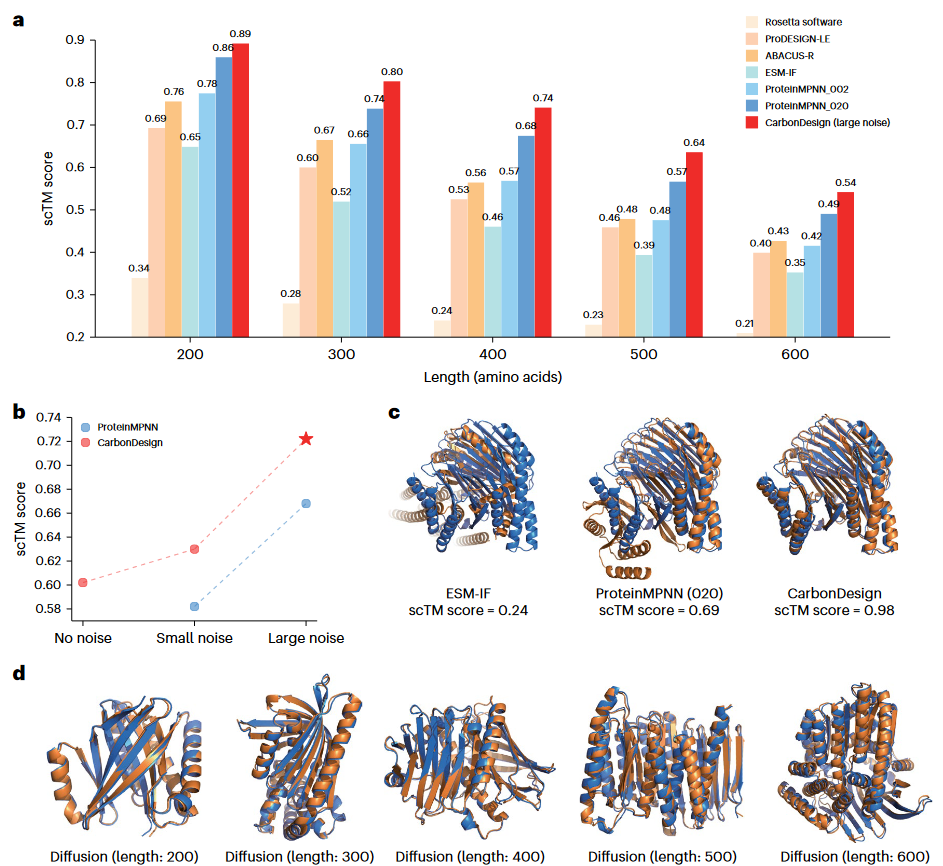
图3
作者的结果突显了两个主要发现。首先,在每个噪声水平下,CarbonDesign的scTM得分始终优于ProteinMPNN(图3b)。其次,作者观察到较高的噪声水平提高了CarbonDesign和ProteinMPNN的性能,这表明噪声在生成de novo结构序列中具有有益作用。更具体地说,CarbonDesign在所有不同长度上都优于现有的代表性方法,包括ProteinMPNN和ESM-IF(图3a)。
此外,作者展示了一个由500个残基组成的生成主链结构的成功例子。CarbonDesign的scTM得分为0.98,显著高于ESM-IF(scTM = 0.24)和ProteinMPNN(scTM = 0.69)(图3c)。作者还展示了其他不同长度的成功设计序列示例(图3d)。
编译 | 黄海涛
审稿 | 曾全晨
参考资料
Ren, M., Yu, C., Bu, D., & Zhang, H. (2024). Accurate and robust protein sequence design with CarbonDesign. Nature Machine Intelligence, 6(5), 536-547.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢