DRUGAI
今天为大家介绍的是来自Wei Pan团队的一篇论文。孟德尔随机化(MR)使用遗传变异作为工具变量(IV)来研究性状之间的因果关系。与传统的MR不同,顺式MR仅使用顺式SNP去关注单个基因组区域。例如,使用一种蛋白质的顺式pQTL作为疾病的暴露因子,为药物靶点发现开辟了一条成本有效的路径。然而,极少有方法能够有效处理顺式SNP的多效性和连锁不平衡(LD)。在此,作者提出了基于约束最大似然法的cisMR-cML方法,该方法对工具变量假设的违背具有很强的理论支持。作者进一步阐明了当前建模边际遗传效应而非条件遗传效应,以及仅在顺式MR分析中使用暴露相关SNP的做法所带来的严重但被广泛忽视的后果。数值研究表明,作者的方法优于现有的其他方法。在对冠状动脉疾病(CAD)进行的药物靶点分析中,包括一个蛋白质组范围的应用,作者确定了三个潜在的CAD药物靶点,分别是PCSK9、COLEC11和FGFR1。

孟德尔随机化(MR)是一种广泛使用的方法,通过遗传变异作为工具变量(IV)来推断两个性状之间的因果关系,一个性状称为暴露,另一个称为结果。由于遗传变异在受孕时是随机分配并固定的,这减少了混杂因素和反向因果关系的风险。在IV回归框架内,MR要求三个有效的IV假设以获得有效推论:IV必须(1)与暴露相关;(2)与暴露-结果关系的任何混杂因素无关;(3)在控制暴露和混杂因素后,与结果无关。基于这些假设,MR可以提供暴露与结果之间(假定的)因果关系的证据,并可应用逆方差加权(IVW)方法。然而,只有第一个IV假设可以被检验,并且在实践中通过使用与暴露相关的全基因组显著SNP相对容易满足;相对而言,第二和第三个假设无法实证检验,并且由于广泛存在的(水平)多效性,可能被违反。在存在水平多效性的情况下,已经提出了许多MR方法,但大多数方法要求使用独立的IV,这在大多数MR分析中是常见的。
与此同时,MR研究越来越关注使用一些局部和相关的顺式SNP作为IV的小基因组区域,即顺式MR。顺式MR最有前途的应用之一是药物靶点发现,包括药物靶点的优先排序、验证或重新定位。药物靶点MR分析使用一种蛋白质(作为潜在的药物靶点)或其下游生物标志物作为暴露,使用编码该蛋白质的基因的顺式SNP作为IV。尽管这种分析具有重要意义,但它仍然关键依赖于三个有效的IV假设。使用蛋白质作为暴露使违反无水平多效性假设的可能性降低,因为蛋白质在许多传统/多基因MR使用的常见风险因素上游,然而,即使在同一个基因/蛋白质区域的顺式SNP中,不同的生物机制仍可能存在。事实上,仅有一个或两个SNP时,许多稳健MR方法无法应用。最后,更重要的是,正如后续数值研究所示,仅使用独立SNP很可能导致分析中缺乏有效的IV,因为它们与区域内其他SNP相关。作为替代,作者更愿意在顺式MR中使用多个相关的IV。然而,只有少数顺式MR方法对IV假设的违反具有稳健性。
模型部分
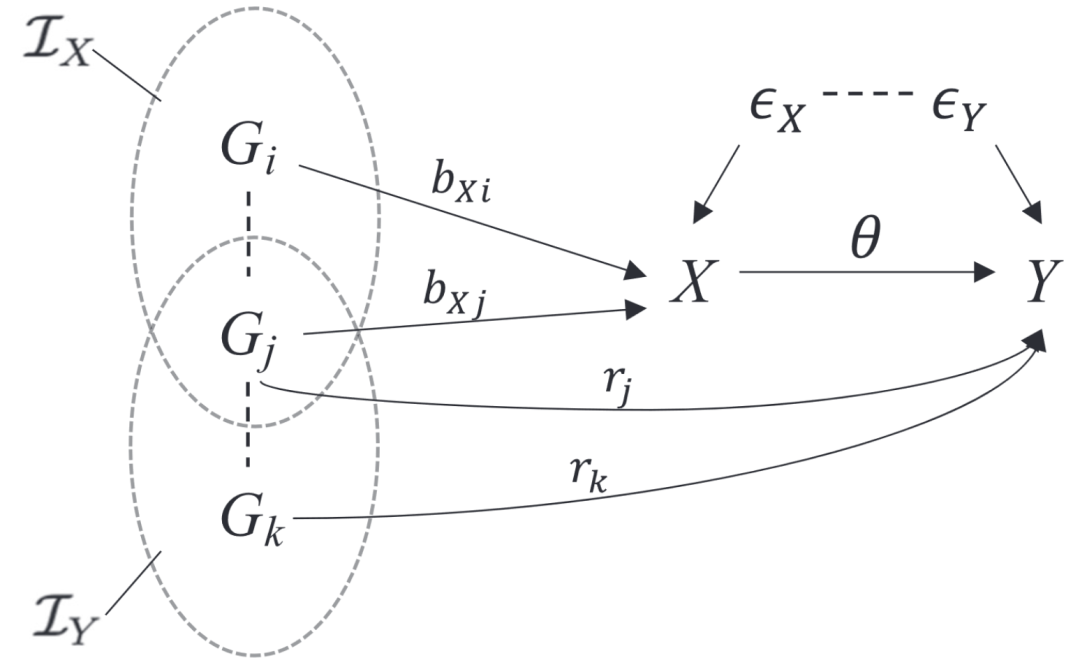
图 1
作者提出了cisMR-cML方法,用于估计暴露(如基因或蛋白质)对结果的因果效应,即使在存在无效IV的情况下,也能使用公开的GWAS总结数据。这是对MR-cML的重要扩展,允许将相关SNP作为分子暴露(如基因或蛋白质)的IV。通过允许使用相关IV,cisMR-cML适用于顺式MR分析,而在顺式区域内使用MR-cML可能无法获得至少三个独立的IV。此外,cisMR-cML相比MR-cML有两个关键区别,增强了其对无效IV的稳健性。首先,如一般因果模型图1所示,cisMR-cML建模了SNP与暴露之间的条件效应,以及SNP与结果之间的条件效应,这不同于MR-cML和其他MR方法中建模边际GWAS估计的传统方法。这一区别显著减轻了在顺式MR中处理相关SNP时引入额外(且不必要)水平多效性的风险。其次,不同于通常只使用与暴露相关的SNP,cisMR-cML使用与暴露或结果共同相关的变异作为IV,即中的变异。作者使用一种称为GCTA-COJO的条件和联合关联分析来选择这些变异。正确考虑与结果相关的SNP进一步有助于避免额外的水平多效性。尽管cisMR-cML中提出的这两个基本考虑在统计上看似简单,但在当前的顺式MR应用中却常被忽视。例如,两种最广泛使用的顺式MR方法——广义IVW和广义Egger,直接建模边际GWAS估计;最近的两个药物靶点应用(如Zhao等人和Zheng等人)在选择条件独立的pQTL后也应用了这些方法。
一旦选择了作为候选工具变量(IV)的遗传变异,就可以使用公开的参考面板估计这些变异之间的连锁不平衡(LD)矩阵。然后将GWAS总结数据中的边际估计转换为条件GWAS估计。接着,在双样本MR框架下,在无效IV数量有限制的情况下,使用最大似然框架实现cisMR-cML,该限制考虑了水平(相关和/或无关)多效性。无效IV的数量通过贝叶斯信息准则(BIC)一致性选择。简而言之,cisMR-cML从候选集中选择有效的IV,以推断X到Y的因果关系。作者建立了统计理论,以证明cisMR-cML的一些理想属性,例如在存在相关或无关多效性的无效IV时的估计一致性和渐近正态性。最后,作者还实施了一种数据扰动(DP)方法,以考虑模型选择的不确定性。给定K值下,作者提出的的cisMR-cML算法如图2所示。

图 2
实验设置
作者进行了两组模拟研究,比较了作者提出的方法(除非另有说明,均使用数据扰动)与其他现有的顺式MR方法,包括广义IVW和Egger(GIVW和GEgger)、考虑LD的Egger(LEgger),以及这些方法的不同实现。在第一组模拟研究中,作者从自回归LD模式中直接生成了10个SNP的GWAS总结统计数据,分别对应弱相关(ρ = 0.2)、中等相关(ρ = 0.6)和强相关(ρ = 0.8),并考虑了两种情景:(1)所有10个SNP都对暴露有影响,即;(2)只有一半的SNP对暴露有影响,即,且 = 2。在这两种情景中,作者改变了中无效IV的数量,记为。作者使用了几种方法:cisMR-cML和LEgger,基于所有10个SNP计算条件估计;GIVW和GEgger,基于边际GWAS估计。在情景(1)中,作者还选择了独立的IV()并应用了IVW、Egger和MR-cML的独立版本,基于边际GWAS估计。作者将这些实现称为IVW-IND、Egger-IND和cML-IND。作者进一步应用了四种可以考虑LD的多基因MR方法,但这些方法并未专门为顺式MR分析提出,包括MR.LDP、MR.Corr2、MR.CUE和MRAID。在情景(2)中,作者还研究了仅使用中的SNP的不同顺式MR方法的表现。具体来说,作者应用了cisMR-cML和LEgger,基于中的5个SNP计算条件估计;应用了GIVW和GEgger,使用中5个SNP的GWAS总结数据。作者将这些实现分别称为cisMR-cML-X、LEgger-X、GIVW-X和GEgger-X。
情景1的实验结果
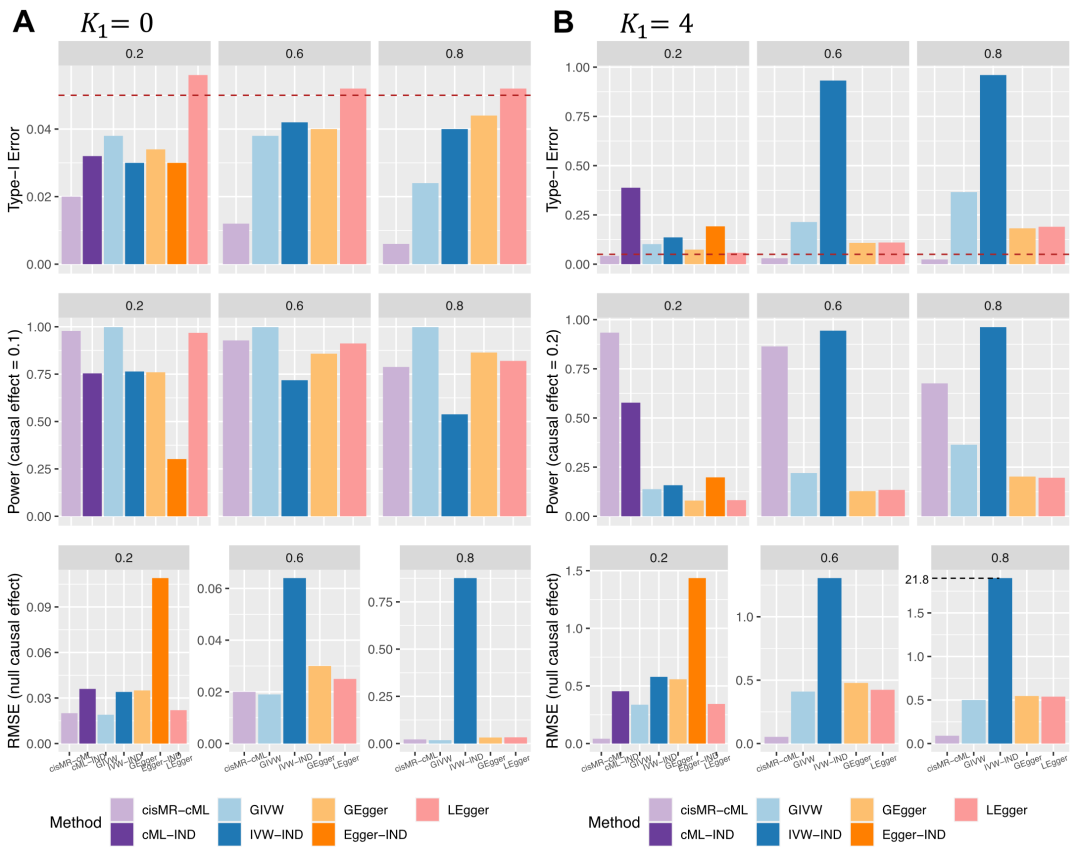
图 3
在第一个情景中,所有10个IV都对暴露有影响(),代表性结果展示在图3中。在整个模拟过程中,作者在显著性水平5%评估了I型错误率。首先,当所有10个IV都有效时(图3A),所有方法的I型错误率都控制得很好。在这种理想情景下,没有无效IV的情况下,cisMR-cML(采用数据扰动实现)比其他方法更保守,这与之前在MR-cML和MVMR-cML中的观察结果相似。需要注意的是,即使在这样的理想情景中,GEgger的均方根误差(RMSE)相对较大(估计值不够精确),这可能是由于方法中实施的等位基因方向步骤。IVW-IND、Egger-IND和cML-IND的效能都低于它们的相关版本对应方法,即GIVW、GEgger和cisMR-cML。
在存在4个无效IV的情况下(图3B),只有cisMR-cML能够控制I型错误率,同时保持高效能。此外,它的RMSE远低于其他三种方法。另一方面,随着SNP之间相关性的增加,GIVW、GEgger和LEgger的I型错误率也逐渐增加。使用独立IV的三种方法的I型错误率也显著增高,因为无效IV的直接效应被吸收到分析中使用的边际GWAS效应中。四种多基因MR方法表现不稳定,表现为极低的效能、增高的I型错误率或未成功收敛。最后,作者进一步研究了在cisMR-cML中使用边际GWAS估计,cisMR-cML-Marg由于边际模型中多重性假设的违反,导致I型错误率显著增加。
情景2的实验结果
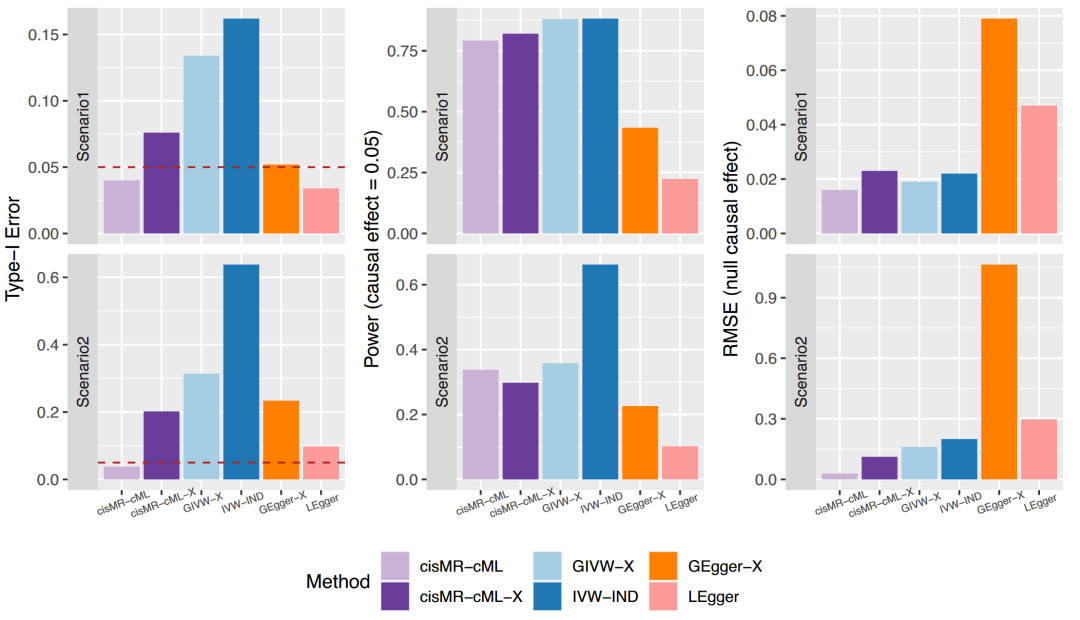
图 4
在第二种情景下,只有5个SNP对暴露有影响,作者进一步研究了仅使用这5个SNP数据的四种方法的表现,如图4中的“-X”后缀所示。当K1 = 0时(图4A),似乎中的所有IV都是有效的。然而,由于它们与中的SNP相关,如果没有包括中的SNP,它们吸收了中SNP对结果的直接效应。因此,中的所有IV都变得无效,并且在cisMR-cML-X中违反了多重性条件,导致I型错误率显著增加。同样,仅使用与暴露条件相关的SNP的GIVW-X和GEgger-X也导致了I型错误率增加。另一方面,使用所有10个SNP的cisMR-cML在所有情景中都能很好地控制I型错误率,保持高效能并具有最小的RMSE。通过这个例子,可以看出在计算条件估计时,包括中的SNP的重要性,因为否则,cisMR-cML所需的多重性条件可能会被违反(除非和中的SNP之间没有或很少有LD)。此外,值得一提的是,当K1 = 1时(图4B),在cisMR-cML使用的10个IV中,有些仅违反了“相关性”假设,有些仅违反了“无水平多效性”假设,但也有些同时违反了这两个假设。值得注意的是,该方法在存在不同类型的无效IV情况下表现稳健,产生了无偏估计并很好地控制了I型错误率。
编译 | 黄海涛
审稿 | 曾全晨
参考资料
Lin, Z., & Pan, W. (2024). A robust cis-Mendelian randomization method with application to drug target discovery. Nature Communications, 15(1), 6072.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢