DRUGAI
理解生物分子的三维结构对于研究它们的功能和相互作用至关重要。这种理解又是设计针对生命细胞机制的治疗分子的基础。在过去的几年中,使用深度学习方法来预测蛋白质和核酸折叠结构方面取得了显著进展。最近,诸如RoseTTAFold All-Atom和AlphaFold3 等方法引入了能够预测广泛蛋白质和核酸结构、它们的共价修饰及小分子配体与这些复合物相互作用的模型。
Chai Discovery发布了Chai-1,一款用于分子结构预测的多模态基础模型,适用于药物发现等任务。Chai-1具备先进的预测能力,能够对蛋白质、小分子、DNA、RNA等进行统一预测,并在PoseBusters和CASP15等多个基准测试中表现出色。与许多需要多序列比对(MSA)的工具不同,Chai-1可以在单序列模式下运行,同时保持高性能。

Chai-1模型在PoseBusters基准测试中达到了77%的成功率(相比之下,谷歌的AlphaFold 3为76%),并在CASP15蛋白质单体结构预测中实现了0.849的Cα LDDT预测集(相比之下,ESM3-98B为0.801)。不同于许多依赖MSA的模型,Chai-1可以在不使用MSA的情况下运行,并仍保持高精度。对于多聚体结构,Chai-1甚至能够超越AlphaFold-Multimer。
Chai-1可用于商业应用,并为支持非商业用途提供免费网络界面和开源代码库。其发布旨在通过与研究界和工业界的合作,促进整个生态系统的发展。
Chai-1的主要功能
生物分子结构预测
Chai-1可以直接从原始分子序列和化学信息中预测蛋白质、核酸等生物分子的三维结构。这对于研究分子的折叠方式、相互作用及其在细胞中的功能具有重要意义。
蛋白质-配体结构预测
Chai-1擅长预测**蛋白质与药物分子(配体)**的相互作用结构,帮助研究人员理解药物如何与蛋白质结合,并为药物设计提供参考。
蛋白质复合物预测
该模型可以预测蛋白质-蛋白质复合物的三维结构,尤其是蛋白质多聚体之间的相互作用。这对于研究蛋白质功能和设计蛋白质药物至关重要。
单序列结构预测
Chai-1可以从单序列输入中进行高精度的结构预测,而无需多序列比对(MSA),即使在数据不足或无相关序列信息的情况下,仍能保持优异性能。
基于实验数据的精准预测
Chai-1能够利用实验数据(如质谱数据或表位图谱)提供的约束信息,进一步提高结构预测的准确性,特别是在复杂分子相互作用的预测中。
抗体-抗原相互作用预测
Chai-1在抗体-抗原相互作用的预测方面具有极高的准确性,能够帮助研究人员准确预测抗体与抗原的结合模式,促进抗体药物的设计和开发。
多模态输入支持
Chai-1支持多种输入形式,包括蛋白质序列、化学配体信息、实验数据等,使其具备预测复杂分子结构的能力,适用于广泛的生物和药物开发任务。
Chai-1 模型的架构
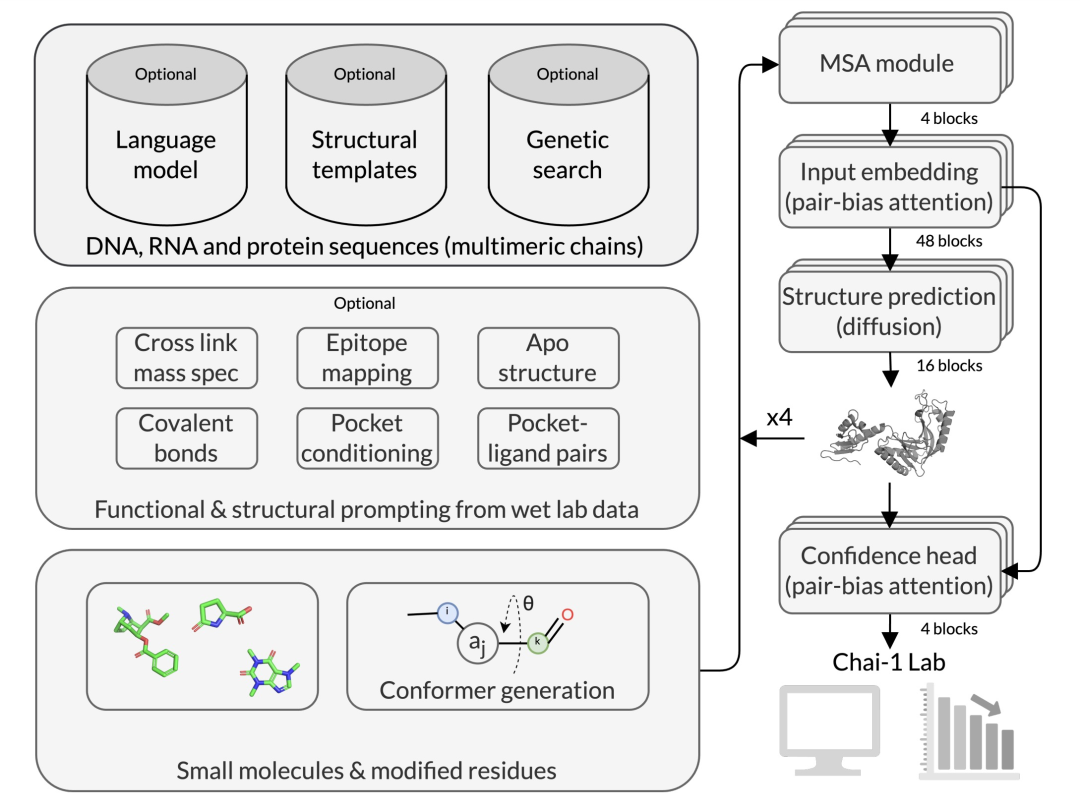
整体架构
Chai-1的模型架构主要基于深度学习神经网络,类似于传统的生物分子结构预测模型,但在多个关键方面进行了改进。该模型的设计允许多种输入形式,包括蛋白质序列、语言模型嵌入和实验约束数据,从而增强了预测的灵活性和准确性。
语言模型嵌入
Chai-1在架构中引入了蛋白质语言模型嵌入,这是一种基于蛋白质序列为每个残基生成嵌入表示的方法。该嵌入由一个拥有30亿参数的蛋白质语言模型生成,旨在捕捉序列中的语法和结构信息。这种设计使得Chai-1在单序列模式下实现高精度预测,尤其是在没有多序列比对(MSA)信息的情况下,模型依然能够表现出色。
约束特征
Chai-1支持实验约束输入,如通过质谱实验获得的结构数据或表位图谱信息。模型的约束特征包括以下几种:
口袋约束:通过提供分子结合口袋的距离约束,模型能够更好地预测分子间相互作用的位置。
接触约束:通过指定分子残基之间的接触距离,模型可以预测多分子系统中残基的相对位置。
对接约束:模型基于不同链或分子组之间的距离约束预测分子系统的对接模式。
这些约束特征在训练过程中通过dropout机制进行随机化,确保模型不过度依赖特定的约束,从而在推理过程中保持泛化能力。
多模态输入和可选结构模板
除了语言模型嵌入和实验约束,Chai-1还支持多模态输入,例如多序列比对(MSA)中的共进化信号和结构模板。MSA信息通常用于捕捉蛋白质序列中的进化信号,而结构模板则提供了额外的空间约束信息,有助于提高复杂结构的预测精度。
这些多模态输入的综合使用使Chai-1在实验数据或结构信息稀缺的情况下,仍能保持高预测精度和灵活性。
改进的训练和推理策略
Chai-1的训练策略基于大量蛋白质和生物分子结构数据,使用大规模GPU并行计算。截止到2021年,该模型使用了蛋白质数据库(PDB)和AlphaFold数据库(AFDB)的数据进行训练,并从PDB70数据库中使用了结构模板。
在推理过程中,模型可以通过随机采样和扩展搜索策略生成多个预测结构,并基于置信度选择最佳预测。模型可以在推理时禁用dropout机制,以提高结果的一致性和可重复性。
模块化设计
Chai-1的架构设计采用模块化方法,能够根据任务需求在推理过程中选择性启用或禁用某些输入特征。例如,当没有MSA数据时,用户可以选择依赖语言模型嵌入,或者通过实验约束信息提高特定分子系统的预测精度。
Chai-1实验结果
蛋白质-配体预测:在PoseBusters基准测试中,Chai-1的预测成功率达到77%,与AF3相当。当结合对接约束时,成功率提升至81%。
肽聚合物预测:Chai-1在单序列模式下(不使用MSA)表现与使用MSA的AF2.3模型相当,在一些评估中甚至超越了AF2.3。
抗体-蛋白质预测:Chai-1在预测抗体-抗原相互作用方面表现出色,使用约束时精度显著提高,DockQ成功率明显高于AF2.3。
蛋白质单体预测:在不使用MSA的情况下,Chai-1的预测精度略低于AF2.3,但在使用MSA时,Chai-1的表现优于AF2.3。
Chai-1在多种生物分子预测任务中表现出优异的性能,以下是重点实验结果总结:
蛋白质-配体预测
测试集:使用PoseBusters基准测试集进行评估,该测试集包含427个蛋白质-配体结构。
评估指标:基于配体的均方根偏差(RMSD),当RMSD小于2Å时视为成功预测。
结果:
Chai-1的预测成功率为77.05%,与AlphaFold3(AF3)的76.34%相当。
当使用对接约束时,Chai-1的成功率提高到81.20%,优于无约束情况下的表现。
在某些情况下,Chai-1有时预测出比真实结构更深的配体结合口袋,表明该模型能够捕捉潜在的结合位点。
肽聚合物(蛋白质复合物)预测
测试集:基于来自蛋白质数据库(PDB)的929个蛋白质-蛋白质界面和1054个蛋白质复合物结构,所有条目均在模型训练数据集截止日期之后。
评估指标:通过DockQ得分(DockQ > 0.23)评估成功率。
结果:
Chai-1在预测蛋白质-蛋白质界面方面的成功率为75.1%,显著高于AlphaFold 2.3的67.7%。
即使在没有多序列比对(MSA)的单序列模式下,Chai-1的成功率也达到了69.8%,与使用MSA的AF2.3相当,表明其单序列模式的预测能力非常强。
在抗体-蛋白质相互作用的预测中,Chai-1的成功率为52.9%,明显高于AF2.3的38%。即使在没有MSA的情况下,Chai-1的表现也非常出色。
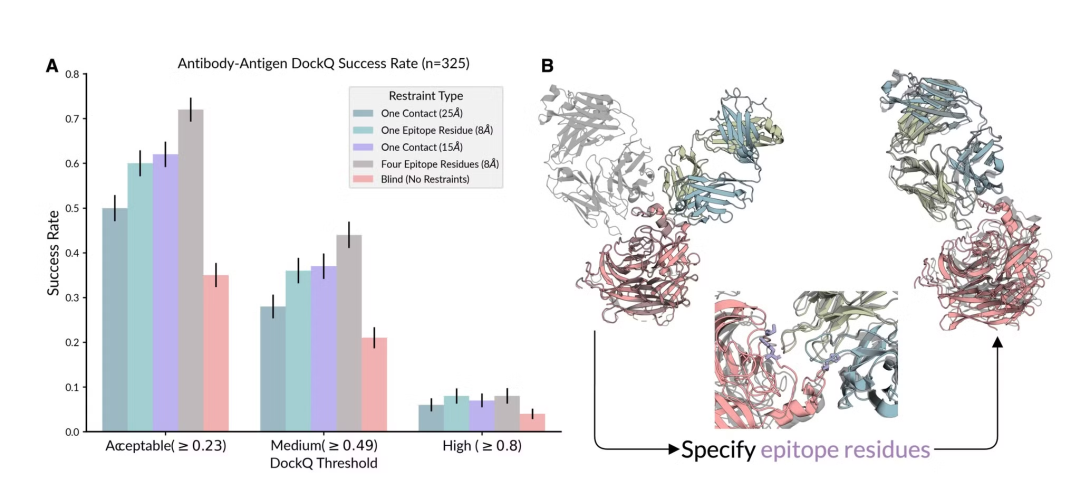
抗体-抗原相互作用预测
测试集:包含268个抗体-抗原界面,并评估模型在约束条件下的预测性能。
评估方法:通过实验数据模拟口袋和接触约束,并使用DockQ成功率进行评估。
结果:
当模型在无约束(盲)模式下运行时,预测的DockQ成功率为35%。
提供抗体-抗原距离约束(θ ≤ 15Å)后,成功率提高到57%。
当提供四个抗体-抗原表位约束时,预测成功率翻倍,但高质量预测仍相对罕见(约4-8%),表明高质量抗体-抗原结构预测仍具挑战性。
蛋白质单体预测
测试集:基于447个蛋白质单体,数据集经过严格同源性筛选,以确保与训练数据的同源性较低。
评估指标:使用Cα-LDDT评估结构预测的准确性。
结果:
使用MSA时,Chai-1的平均LDDT得分为0.915,略高于AF2.3的0.903。
Chai-1在无MSA的单序列模式下的LDDT得分为0.852,虽然略低于AF2.3,但仍然具有较高的准确性。
核酸结构预测
测试集:包含低同源性的蛋白质-核酸复合物测试集和CASP15 RNA目标,以评估模型预测RNA及其与蛋白质相互作用的能力。
评估指标:使用Cα-LDDT和C1′-LDDT评估结构准确性。
结果:
在蛋白质-核酸复合物预测中,Chai-1的表现与RoseTTAFold2NA模型相当,尽管Chai-1未使用核酸的多序列比对信息。
在CASP15 RNA目标中,Chai-1的平均LDDT为0.849,高于AF2.3的0.843。
置信度评估
Chai-1通过预测的TM得分(ipTM)有效评估模型的预测置信度。结果表明,ipTM得分与预测质量有良好的相关性,能够有效区分高质量和低质量的预测结果。
编译:王建民
参考资料
模型地址:
https://www.chaidiscovery.com/blog/introducing-chai-1
GitHub:
https://github.com/chaidiscovery/chai-lab
技术报告:
https://chaiassets.com/chai-1/paper/technical_report_v1.pdf
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢