DRUGAI
今天为大家介绍的是来自福州大学的翁祖铨团队的一篇论文。非预期的药物–药物相互作用(DDIs)是药物研究和临床应用中的重要问题,因为它们具有引发严重不良药物反应或导致药物撤市的高风险。许多深度学习模型在DDI预测方面已经取得了很高的性能,但这些模型在解释DDI的潜在原因方面的可解释性并未得到广泛研究。本文提出了一种名为MeTDDI的深度学习框架,该框架通过局部-全局自注意机制和协同注意机制来学习基于模体的图用于DDI预测。MeTDDI在性能上与最先进的模型相比具有竞争力。在可解释性方面,作者对73种药物的13,786个DDI进行了广泛评估,MeTDDI能够准确解释涉及58种药物的5,602个DDI的结构机制。此外,MeTDDI显示出解释复杂DDI机制和减少DDI风险的潜力。总的来说,MeTDDI为探索DDI机制提供了新的视角,将有助于药物发现和多药治疗中更安全的治疗方法。

随着人口老龄化和多病共存,药物组合(即多药治疗)广泛使用,可能带来公共卫生和经济后果。尽管多药治疗具有治疗效果,但也伴随意外药物–药物相互作用的风险,可能导致严重不良反应甚至药物撤市。因此,提前预测DDI对提高药物安全性和保护患者健康有显著益处。传统的体外和体内实验虽然有用,但耗时、昂贵且劳动力密集,不适合大规模筛选。近年来,深度学习模型为高通量准确预测DDI提供了有前景的替代方案。
目前最先进的模型主要分为基于生物医学图谱和分子结构的模型。生物医学图谱利用图神经网络等方法,虽然提升了DDI预测性能,但受限于数据的质量与可用性。而分子结构模型则基于药物分子的结构预测DDI,假设DDI源自分子中的子结构或官能团。尽管表现优异,但仍面临三大挑战:准确学习分子内外的子结构互动,预测药物代谢变化,以及模型可解释性。
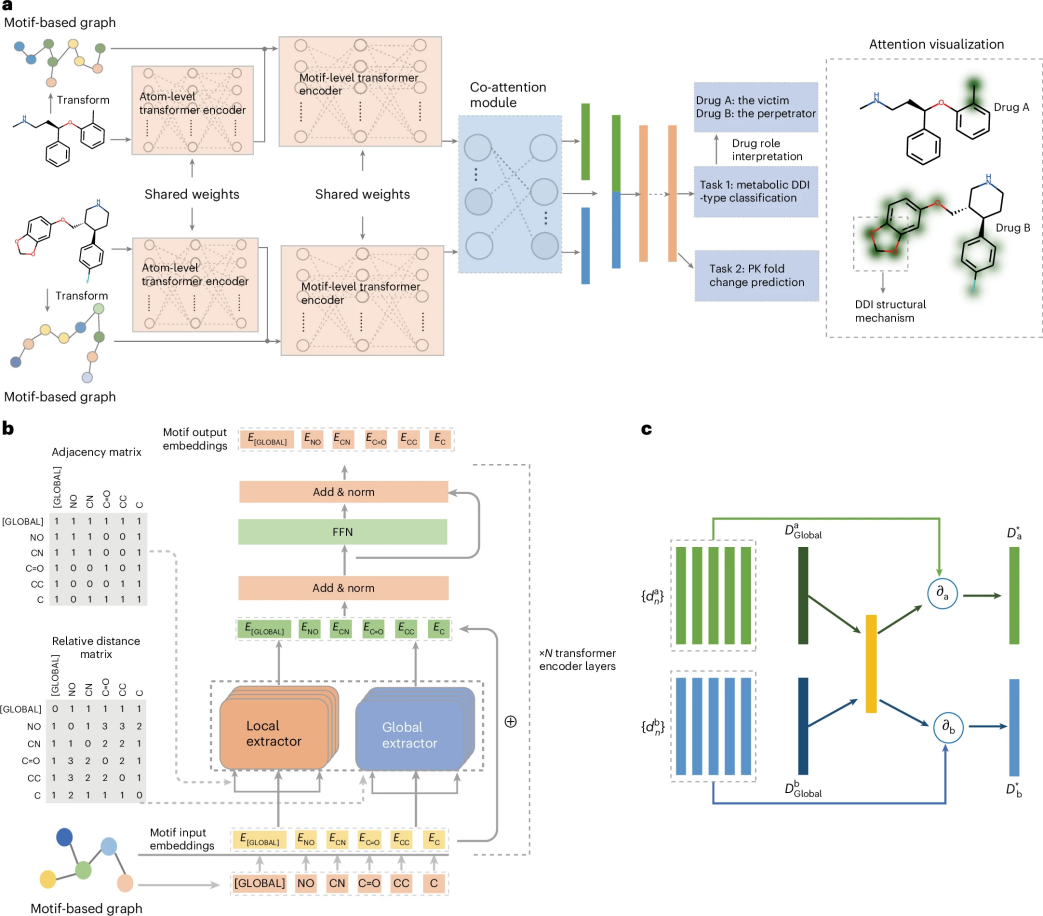
图 1
在图1中,作者提出了一种基于分子结构的深度学习框架——MeTDDI,用于预测代谢介导的药物–药物相互作用。实验表明,MeTDDI在性能上与最先进模型相媲美,并提供了关于DDI风险缓解和机制解释的新见解。
问题表述
给定一对药物,通过分析药物对在同一代谢酶下的作用机制,可以预测潜在的代谢介导的药物–药物相互作用(MMDDIs),并利用开放数据库(如DrugBank和SuperCYP)进行辅助。与MMDDI分类相比,更具挑战性的是识别药物对中的致效药(perpetrator)和受害药(victim),以及量化致效药对受害药药代动力学(PK)的影响。致效药是在药物相互作用中改变另一药物代谢或药效的药物。它通过影响代谢酶或其他机制,改变受害药的代谢或药代动力学,导致药物浓度增加或减少,进而影响疗效或引发不良反应。
因此,本研究关注两个与MMDDI相关的预测任务:(1)预测作用机制类型(如酶抑制或酶诱导)并识别致效药和受害药(分类任务);(2)量化受害药的PK倍数变化(回归任务)。作者基于MMDDI描述的语义定义了四种分类标签:Label1和Label3表示酶抑制,Label2和Label4表示酶诱导。Label1和Label2反映了DDI的真实语义,而Label3和Label4则表示错误的DDI描述。
在设置中,一个药物分子由两个图表示:分子图Ga = (Va, Ea),其中Va表示原子,Ea表示键;模体图Gm = (Vm, Em),其中Vm和Em分别表示模体的集合及其连接。给定任意药物对(Da, Db),预测任务可以表示为一个函数F:F(Da, Db)→labeli∈[1,2,3,4]或c。其中labeli∈[1,2,3,4]表示目标DDI标签,c表示PK倍数变化的值。
任务1:分类任务
在该任务中,作者在四种场景下测试了模型:未见的DDIs(S1)、未见的单药物(S2)、未见的双药物(S3)以及一个外部数据集(DDInter)。
表 1

在S1场景中,除了CGIB和SA-DDI外,MeTDDI在所有评估指标上显著优于所有基线模型。在S2、S3和DDInter场景中,所有模型的预测性能都显著下降,但MeTDDI依然保持了较强的预测能力。
任务2:回归任务
为了量化致效药对受害药药代动力学的影响,作者进一步预测了受害药的血浆浓度时间曲线下面积倍数变化(AUC FC)值。数据集来源于美国食品药品监督管理局(FDA)的药品标签,包含684种药物和3,987个DDIs。
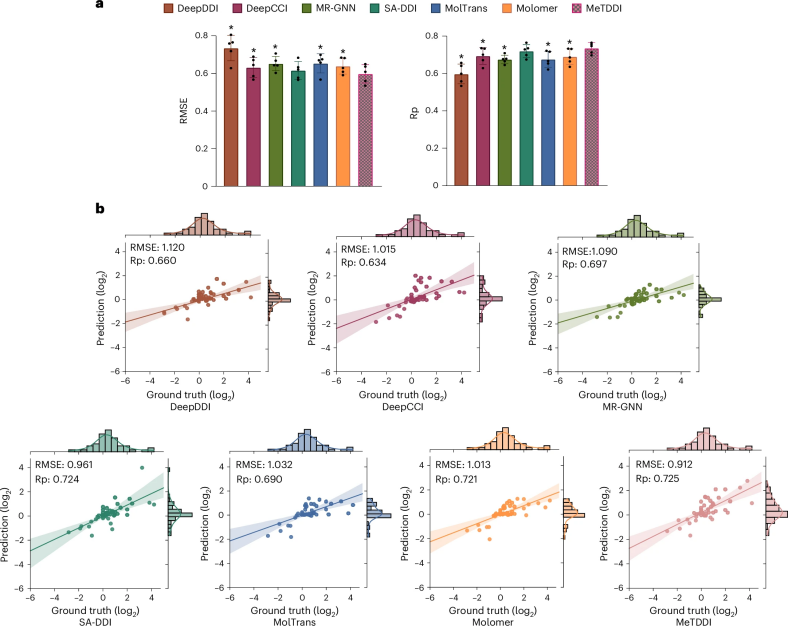
图 2
图2a展示了MeTDDI和基线模型通过五折交叉验证的均方根误差(RMSE)和皮尔逊相关系数(Rp)。MeTDDI显著优于大多数基线模型,并与SA-DDI相当,其平均RMSE为0.596,Rp为0.736。
作者还使用包含FDA在2023年批准的9种新药的47个DDIs的额外数据集验证了MeTDDI和基线模型。图2b显示,MeTDDI表现最佳,其RMSE为0.912,Rp为0.725,突显了MeTDDI在量化PK变化方面的出色预测和泛化能力。
DDI机制的结构解释
在MMDDIs中,致效药通过诱导或抑制代谢酶来影响受害药的药代动力学。因此,开发能够提供致效药关键结构见解的模型对MMDDIs的研究具有重要意义。本部分中,MeTDDI的可解释性通过文献报道的酶抑制致效药关键子结构进行了测试。手动整理的数据集包含73种通过特定子结构或官能团抑制代谢酶引发MMDDIs的化学物质,以及这些化学物质作为致效药引发的13,786个DDIs。
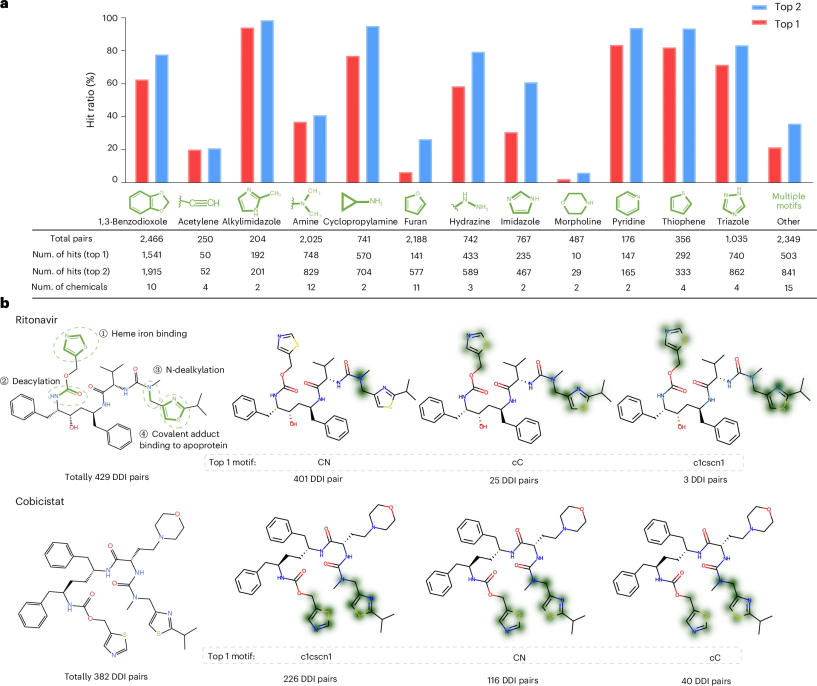
图 3
MeTDDI的协同注意模块识别了致效药中得分最高的两个模体。对每个致效药,其前两个模体与文献报道的官能团进行了比较。在13,786个DDIs中,5,602个与文献中的首个模体匹配,7,564个与前两个模体匹配(图3a)。此外,MeTDDI还识别出了七个可能作为酶抑制主要机制的官能团。这些官能团占据了超过50%的首个模体匹配率,超过75%的前两个模体匹配率。
MeTDDI解释DDI的争议性结构机制
MMDDIs的潜在机制复杂多样,尤其是在代谢酶抑制方面,一些化学物质可能根据其结构特征表现出多种机制。幸运的是,MeTDDI提供了一种统计方法来探索化学物质酶抑制的主要机制。作者利用MeTDDI进行了一项案例研究,确认了利托那韦对CYP3A酶抑制的主要机制。通过分析利托那韦的结构特征,可以识别出其作为致效药如何影响受害药的关键机制。
作者收集了429个MMDDI药物对,其中预测利托那韦作为致效药,受害药通过CYP3A酶(CYP3A4和CYP3A5)代谢。在图3b中,作者使用MeTDDI可视化了利托那韦结构中每个DDI药物对的最高权重模体,发现其中三个子结构与CYP3A抑制有关。其中两个子结构“CN”和“c1cscn1”已在之前的文献中报告,通过N-脱烷基化和血红素铁结合或与载体蛋白形成共价加合物失活CYP3A。
根据MeTDDI的可视化结果,CN 模体是大多数药物对(429个中的401个)的主要子结构。此外,MeTDDI还可视化了“cC”模体,该模体是25个DDI药物对的主要子结构。尽管作者缺乏证据证明该模体如何参与CYP3A抑制机制,但推测它可能与“c1cscn1”模体的抑制机制有关,因为它与“c1cscn1”模体直接相连。利托那韦含有两个“c1cscn1”模体(也称为噻唑环),MeTDDI倾向于在所有可视化中为这两个噻唑环分配高权重,这与利托那韦需要两个噻唑环才能发挥CYP3A抑制作用的证据相符。
作者还使用相同的程序测试了利托那韦的类似物——考比司他,发现相同的模体类型,但排名不同。“c1cscn1”模体被确认为考比司他在大多数DDI药物对中的主要机制(382个中的226个),并且MeTDDI对这两个噻唑环的权重相同。
使用MeTDDI进行DDI缓解的结构优化
通过对抑制相关官能团的药物候选物进行结构优化,可以减少或缓解DDIs,消除不良抑制作用并尽量减少受害药物的暴露。然而,传统的优化过程需要大量时间和成本来列举可能的修改并进行验证。作为解决方案,MeTDDI提供了一种快速有效的方法,通过计算测试修改后的分子结构是否能减少或缓解DDIs。作者以帕罗西汀和伊曲康唑为例进行了演示。
帕罗西汀是一种广泛使用的抗抑郁药,它通过使CYP2D6失活,可能增加其受害药物的血浆浓度以造成危险。帕罗西汀分子中的1,3-苯二氧杂环基团负责CYP2D6的准不可逆失活。最近的研究发现,通过将该基团的两个氢取代为氘,氘代帕罗西汀(CTP-347)可以缓解人体内的CYP2D6失活。
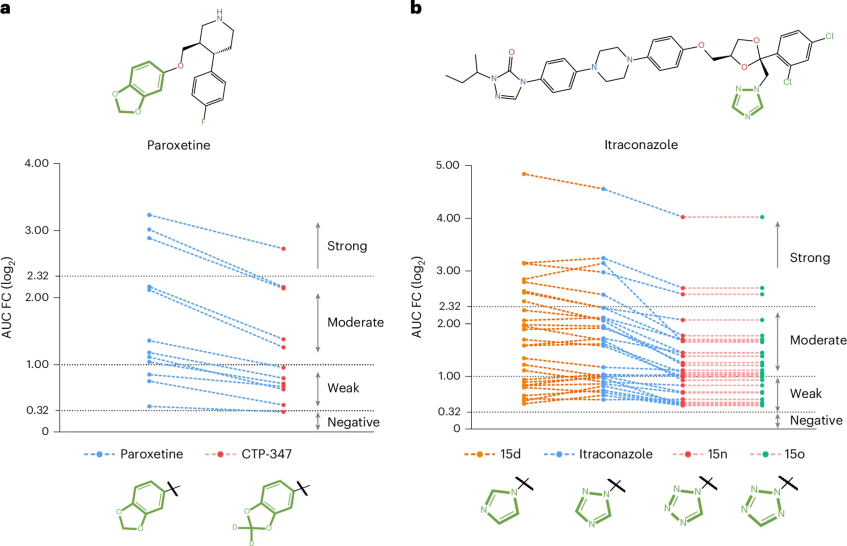
图 4
为验证这一点,作者从任务2的数据集中收集了12对帕罗西汀作为致效药的DDI药物对,并用CTP-347替换帕罗西汀。然后,作者使用MeTDDI预测每个受害药物的AUC FC值。图4a显示,CTP-347降低了所有12个受害药的AUC FC值,特别是阿托莫西汀和匹莫齐特的log2(AUC FC)值分别从2.89和3.02(强效)降至2.14和2.16(中等)。尽管CTP-347仍会对CYP2D6代谢的药物产生中度影响,但其抑制机制已从准不可逆变为可逆且具有竞争性,不再完全失活CYP2D6,也不会引发持续的DDIs。
除了帕罗西汀的原子替换外,伊曲康唑的修改是子结构替换。伊曲康唑是一种抗真菌药,最近研究显示它具有抗肿瘤活性与抗血管生成活性。然而,因其强效抑制CYP3A4而引发的DDIs限制了其用于癌症治疗。现有研究表明,1,2,4-三唑环是主要原因,将其替换为四唑环可以大大减少CYP3A4的抑制作用。
为测试MeTDDI是否能够预测这一情况,作者从任务2的数据集中收集了28对伊曲康唑作为致效药的DDI药物对,并分别用伊曲康唑类似物(15d、15n和15o)替换伊曲康唑,这些类似物分别用咪唑、1-四唑基和2-四唑基替代了1,2,4-三唑环。作者预测了所有受害药物的AUC FC值。如图4b所示,与伊曲康唑相比,15n和15o主要降低了受害药物的AUC FC值,而15d则增加了受害药物的AUC FC值。这些结果与体外实验一致,15n和15o减轻了CYP3A4的抑制作用,而15d增强了这种抑制作用。
讨论
本研究提出了MeTDDI模型,解决了DDI预测中的三个挑战:学习分子内外的子结构相互作用、预测药物代谢以及提供模型可解释性。通过局部-全局自注意机制和协同注意机制,MeTDDI有效捕捉了基于模体图的相互作用,在分类和回归任务中表现优异。MeTDDI能够识别关键的结构子机制,验证结果与文献匹配,并成功预测药物的代谢位点。此外,MeTDDI还展示了在化学物质结构优化方面的潜力,有助于缓解MMDDI风险。然而,MeTDDI在处理复杂DDI机制时仍面临挑战,并且在多药物相互作用预测中也存在一定局限。未来可结合分子对接技术来增强模型的可信度。
编译 | 于洲
审稿 | 曾全晨
参考资料
Zhong Y, Li G, Yang J, et al. Learning motif-based graphs for drug–drug interaction prediction via local–global self-attention[J]. Nature Machine Intelligence, 2024: 1-12.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢