DRUGAI
今天为大家介绍的是来自浙江大学王毅团队的一篇论文。大规模药物发现与再利用面临诸多挑战。识别药物的作用机制(MOA)至关重要,但现有方法成本高且通量低。本文提出了一种通过分析线粒体表型变化来识别MOA的方法。通过对线粒体形态和膜电位的时间序列成像,作者建立了一个用于监测时间分辨线粒体图像的流程,生成了一个包含570,096张单细胞图像的数据集,这些细胞暴露于1,068种美国食品药品监督管理局批准的药物中。作者开发了一种名为MitoReID的深度学习模型,该模型基于重识别(ReID)框架,并采用了膨胀的3D ResNet骨干网络。在测试集中,该模型实现了76.32%的Rank-1精度和65.92%的平均精度,并成功基于线粒体表型识别了六种未训练药物的MOA。此外,MitoReID还识别出茶中天然化合物表儿茶素的MOA为环氧合酶-2抑制,并通过体外实验成功验证。作者的方法因此提供了一种自动化且成本效益高的靶标识别替代方案,能够加速大规模药物发现与再利用。

药物发现与再利用因多种因素而复杂且具挑战性。研究表明,新药上市的成本超过20亿美元,时间超过十年。高成本和长周期对大规模药物发现构成重大挑战,尤其是药物靶点的识别。生物系统的复杂性、广阔的化学空间以及对高质量数据的需求进一步增加了难度。虽然现有的MOA识别方法如生化实验和基因干扰提供了有价值的见解,但它们在处理复杂生物系统中的新化合物时仍有局限性。为克服这些限制,基于图像的分析方法已成为预测MOA的有前途手段。
基于图像的分析策略旨在将生物图像中的信息提炼为多维特征,以便同时分析各种细胞特征和表型,为细胞对药物反应提供全面视图。这种方法已经成功用于疾病相关表型的识别以及小分子生物活性或毒性的预测。线粒体在细胞生物学中具有多种功能,包括代谢控制,其形态和功能受到多种药物的影响。基于显微镜的高内涵成像技术使得线粒体结构和功能在复杂细胞环境中的可视化成为可能。
然而,传统方法仅提取了有限的形态特征,导致信息丢失,不利于靶点识别。为了解决这一问题,作者引入了时间维度,并开发了一个名为MitoReID的深度学习模型,通过分析线粒体表型来识别MOA。该模型成功将477种FDA批准的药物分类为38种已知MOA,并正确识别了六种未见过的药物及数十种天然化合物的MOA。这项研究通过结合大规模线粒体动态监测与深度学习方法,建立了一种快速、自动且无偏的化学物质MOA预测方法。
基于深度学习的MOA识别框架
如图1所示,该研究框架包含四个基本组成部分。
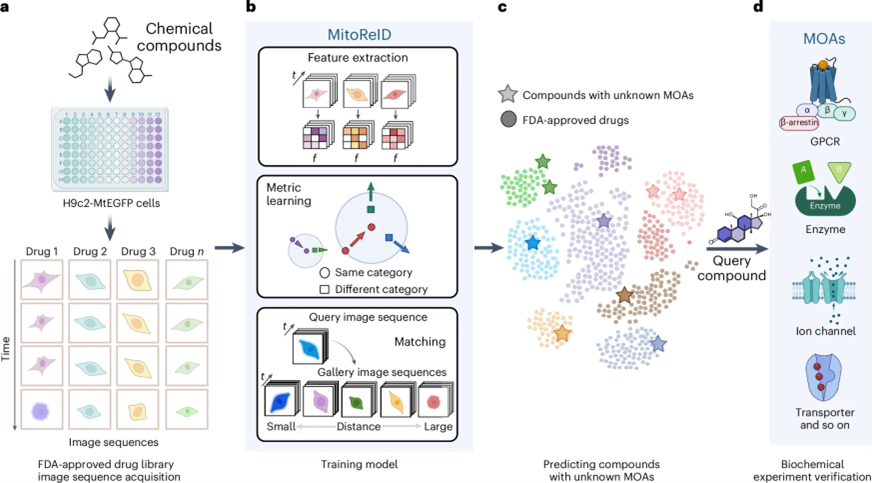
图 1
首先,作者建立了一个稳健的细胞损伤模型,通过1,068种FDA批准的药物来观察细胞的应激反应。通过严格的高通量方法,特别关注线粒体形态和膜电位,得到了时间序列图像,这些图像构成了原始数据集的基础。随后,在重识别(ReID)框架下,作者利用这些图像序列训练了一个创新的深度学习模型MitoReID。
在下一阶段,作者使用训练好的模型提取未见过的药物和天然化合物的特征,并将这些特征与FDA批准药物的特征进行余弦距离匹配,根据与最近药物的MOA关联推断预测结果。最后,进行体外生化实验,以验证未识别天然化合物的假设MOA。
通过活细胞成像监测线粒体动态
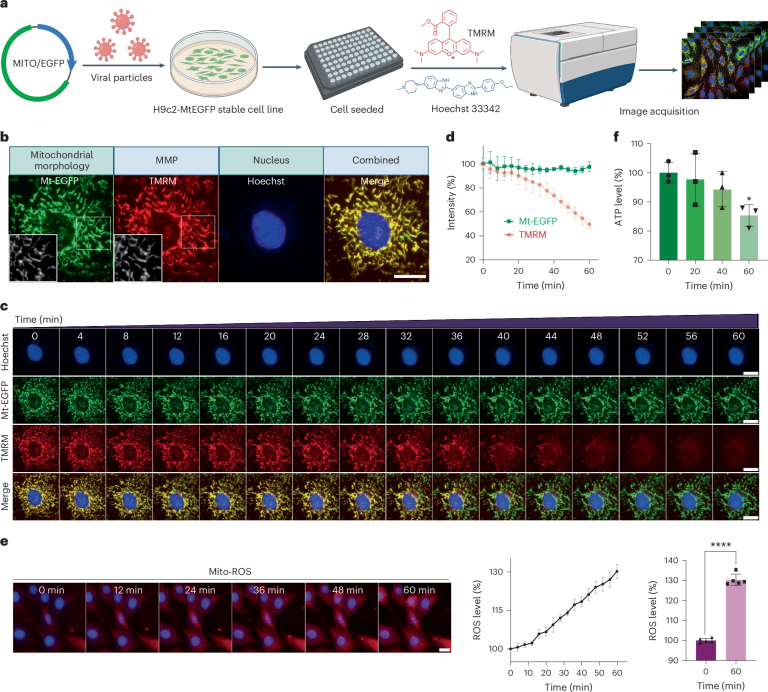
图 2
如图2a所示,作者结合了TMRM成像和MtEGFP,通过基于慢病毒的方法将其稳定地转导到细胞中,从而能够同时监测线粒体形态和线粒体膜电位(MMP)。作者随后将细胞接种到96孔微孔板中,孵育24小时后进行染色和成像,以同时监测线粒体形态、膜电位和细胞核(图2b)。
为了研究暴露于多种药物的细胞中线粒体表型的时间变化,作者开发了一个氧化应激的细胞模型,用于高通量获取线粒体形态和膜电位。图2c观察到了线粒体形状、长度、质量和MMP的随时间变化。定量分析显示,MtEGFP的荧光强度随时间保持稳定,而TMRM的荧光强度逐渐下降至初始值的49.7%(图2d)。与MMP相比,H9c2-MtEGFP细胞中的线粒体活性氧(ROS)水平随着时间的推移逐渐增加了30.4%(图2e),并且在经过16次激光刺激后,ATP水平略有下降(图2f)。
因此,作者建立了一种基于光损伤的策略来诱导细胞损伤,同时在此过程中实现线粒体动态的纵向分析。
具有不同MOA的药物表现出多样化的线粒体表型
作为筛选平台概念验证的一部分,作者记录了暴露于carvedilol(一种具有保护线粒体免受毒性作用的肾上腺素能受体拮抗剂)和cisplatin(一种已知会引起线粒体功能障碍的DNA烷基化剂)后线粒体动态的图像。
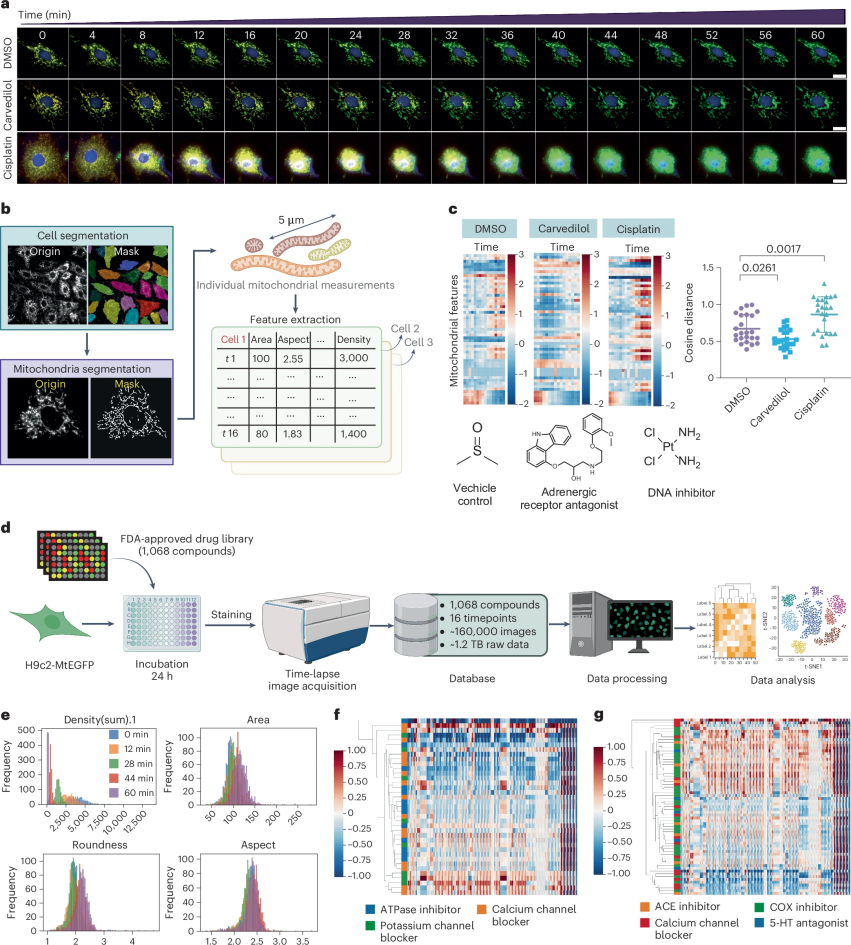
图 3
如图3a所示,这两种药物对线粒体表型的影响截然不同。cisplatin处理的心肌细胞显示出线粒体内向绿色荧光的更快转换,并伴有细胞萎缩,表明细胞凋亡;而carvedilol处理的细胞中,线粒体动态总体上与对照组相似,在初始时间点线粒体网络连接性增加。
为了定量评估线粒体图像的变化,传统方法通常涉及线粒体的分割和相关测量特征的提取。在图3b中,作者通过Cellpose进行细胞分割,然后使用Image Pro Plus v.6.0(IPP)对单细胞图像进行批量线粒体分割。图3c中的特征图谱揭示了对照组、carvedilol和cisplatin暴露之间的显著差异。
为了进一步研究各种药物对线粒体表型的时间影响,作者分析了一个包含1,068种FDA批准药物的商业化合物库。图3d-e显示,关键线粒体特征在时间上存在变化,其中线粒体膜电位表现出最明显的时间变化。
为了区分暴露于不同药物后线粒体反应之间的关联性,作者进行了分层聚类分析,将药物分类为三类可能对线粒体功能产生重大影响的药物。图3f-g显示,钙通道阻滞剂和钾通道阻滞剂在与ATP合酶抑制剂相比时,表现出一定的接近性和差异性,但没有明确的界限。此外,四类MOA进行特征分析后也难以区分。这些观察结果表明,传统技术难以区分具有不同MOA的药物。
MitoReID识别相似的线粒体表型
作者开发了一个受行人重识别(ReID)启发的ReID框架,通过特征相似性在多个视野下匹配未识别的对象与已知的身份。这里,已知的身份是具有相同MOA的FDA批准药物,而未识别的对象是MOA未知的新药和天然化合物。对于每个查询样本,识别出图库集中与之最相似的前k个样本,并将这些样本的MOA作为查询样本的潜在候选MOA。
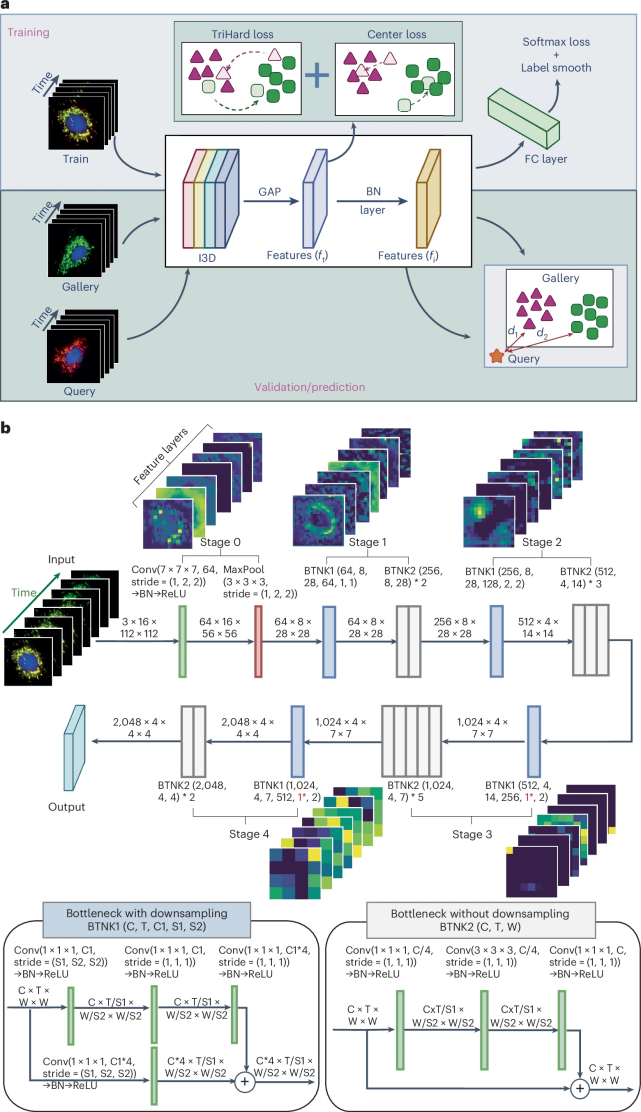
图 4
作者的MitoReID框架采用了Hao Luo及其同事的方法,其通过各种策略增强了行人ReID基准的性能。作者使用了I3D ResNet作为骨干网络。MitoReID流程在训练阶段(图4a上)和验证/预测阶段(图4a下)整合了表示学习和度量学习。
在训练阶段,使用在线batch-hard样本挖掘策略输入图像序列,从每个批次中选择具有挑战性的样本。经过数据增强后,I3D ResNet从输入数据中提取特征。图4b展示了I3D ResNet-50架构,该架构设计用于三维数据处理。作者对其进行了调整,排除了第3和第4阶段的空间下采样组件,以保留更多的空间特征。作者使用全局平均池化(GAP)层来聚合空间信息,生成用于计算TriHard和center损失的特征ft。
TriHard损失专注于选择具有挑战性的正样本和负样本对,从而增强类内和类间的可分离性。同时,center损失旨在最小化特征与其各自类别中心的距离,从而促进更好的聚类。接下来,作者应用批量归一化来规范化特征,通过全连接(FC)层后计算softmax损失。为了应对标签错误或低质量数据,作者引入了标签平滑,增加了一些不确定性以改善模型的泛化能力。
在验证/预测阶段,从查询集和图库集中提取的特征通过训练好的I3D网络进行计算。查询集样本根据与图库样本的特征距离进行排序,识别出距离最短的图库样本作为匹配身份。最后,关联MOA被推断为查询样本的预测MOA。
MitoReID模型在MOA识别中的性能表现
MitoReID模型被用于基于线粒体表型的时间维度分类38种单一MOA类别。作者首先预训练了一个I3D ResNet模型,将每种药物/DMSO视为一个独立的类别,总共生成1,069个类别。然后作者基于预训练模型对MitoReID模型进行了微调,以提升其性能。
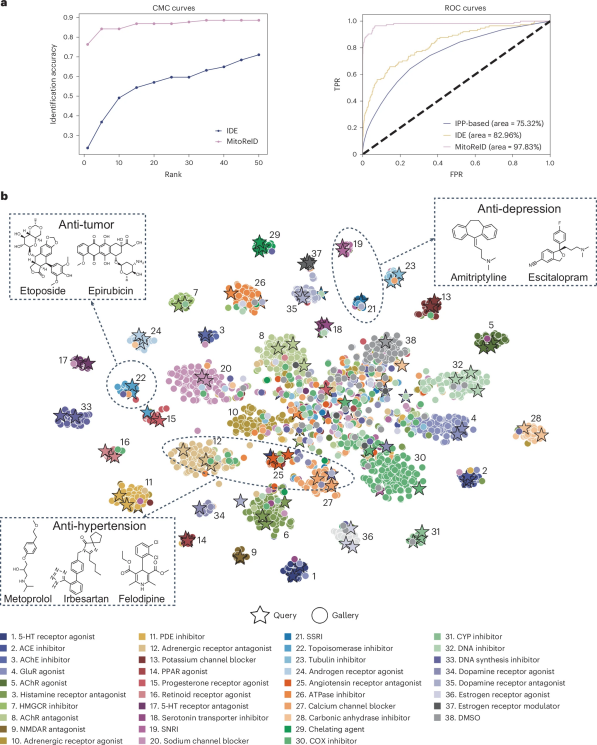
图 5
作者将MitoReID的性能与ReID(IDE)和基于IPP的模型(随机森林、SVM、MLP、LSTM、多通道CNN)进行了比较,利用线粒体表型的时间维度来识别38种MOA。如图5a所示,MitoReID在所有指标上均优于基于IPP的模型和IDE。基于IPP的模型的准确率低于16%,而MitoReID的准确率达到了82.46%。
为了进一步展示MitoReID的区分能力,作者使用t分布随机邻域嵌入(t-SNE)将查询集和图库集的特征向量投影到二维平面上进行可视化。图5b中每个点或星形表示一个图像序列的特征向量,不同的颜色表示不同的MOA类别。图中点或星形的接近程度反映了它们各自特征的相似性。例如,簇19和簇21显示出更近的距离,这与它们具有抗抑郁作用的共同药理特性一致。此外,簇12、簇25和簇27也表现出更紧密的关联,反映了它们在抗高血压方面的作用。
预测未知药物和天然化合物的MOA

图 6
为了评估MitoReID模型的适用性,作者在图6a中测试了八种未在训练集中使用的FDA批准药物。作者将排名前五的MOA视为潜在候选项,排名指标优先考虑Rank-1(越高越好)和余弦距离(越低越好)。如图6b所示,在这八种FDA批准药物中,有六种药物的候选MOA包含正确的标签。
讨论
本研究通过建立高通量线粒体动态成像的流程,在药物表型分析方面取得了显著进展。MitoReID模型展示了预测未知药物和天然化合物MOA的潜力,有望推动大规模药物发现和再利用。MitoReID能够捕捉全面的线粒体特征,超越了传统形态分析技术,并通过自动学习全局特征,减少了复杂的数据预处理和手动特征提取的需求。然而,MitoReID的性能受数据质量、实验条件和标签注释的影响。未来研究将整合药物靶点关系及图神经网络,进一步提升模型的预测能力,并探索新的药物分类框架,为药物发现提供更精准的工具。
编译 | 于洲
审稿 | 曾全晨
参考资料
Yu M, Li W, Yu Y, et al. Deep learning large-scale drug discovery and repurposing[J]. Nature Computational Science, 2024: 1-15.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢