DRUGAI
今天为大家介绍的是来自华盛顿大学的Frank DiMaio团队的一篇论文。基于结构的虚拟筛选是药物早期研发中的一项关键工具,对多达数十亿化学化合物库的筛选兴趣日益增长。然而,虚拟筛选的成功在很大程度上取决于计算对接所预测的结合构象和结合亲和力的准确性。在此,作者开发了一种名为RosettaVS的高精度结构虚拟筛选方法,用于预测对接构象和结合亲和力。作者的方法在广泛的基准测试中优于其他最先进的方法,部分原因在于作者能够对受体的灵活性进行建模。作者将其整合到一个新的开源人工智能加速虚拟筛选平台中,用于药物发现。利用该平台,作者对两种无关靶标——泛素连接酶靶标KLHDC2和人类电压门控钠通道NaV1.7——进行了数十亿化合物库的筛选。对于这两个靶标,作者都发现了命中化合物,包括对KLHDC2的7个命中(命中率为14%)和对NaV1.7的4个命中(命中率为44%),它们均具有个位数微摩尔级的结合亲和力。这两种情况下的筛选都在不到七天内完成。最后,高分辨率的X射线晶体结构验证了对KLHDC2配体复合物的预测对接构象,证明了作者的方法在先导化合物发现中的有效性。

基于结构的虚拟筛选(Structure-Based Virtual Screening, SBVS)是一种通过计算机模拟方法,在已知靶标蛋白质的三维结构的基础上,筛选大量化合物以寻找潜在活性分子的方法。该方法利用靶标蛋白的结构信息,对化合物库中的每个小分子进行对接预测,以评估它们与靶标蛋白的结合能力和亲和力。经过初步筛选发现的具有生物活性的小分子被称为先导化合物。虽然这些化合物还不能直接作为药物用于临床治疗,但它们为进一步的药物开发提供了良好的起点。
基于结构的虚拟筛选在药物发现中起关键作用,可用于识别潜在的化合物。随着包含数十亿化合物的化学库变得容易获取,人们对筛选广阔化学空间以发现先导化合物的兴趣增加。然而,筛选如此大规模的化合物库对于基于物理的对接方法来说非常耗时且成本高昂。近年来,为实现超大规模化合物库的虚拟筛选,引入了一些技术,如可扩展的虚拟筛选平台、高性能计算集群上的并行对接、深度学习引导的化学空间探索、主动学习技术以及GPU加速的配体对接等。
然而,虚拟筛选的成功在很大程度上取决于对蛋白质-配体复合物结构的准确预测以及区分真正的结合分子和非结合分子的能力。目前领先的基于物理的配体对接程序,如Schrödinger Glide和CCDC GOLD等,并不免费,而免费的Autodock Vina在虚拟筛选的准确性方面略低。此外,缺乏一种利用主动学习对超大化学库进行虚拟筛选的开源平台。虽然深度学习在预测蛋白质-配体复合物结构方面取得了一些进展,但更适合于未知结合位点的盲对接问题,对于已知结合位点,基于物理的对接方法仍然表现更好。
在这项工作中,作者开发了一种最先进的基于物理的虚拟筛选方法以及一个开源虚拟筛选平台,能够高效筛选数十亿化合物库。这是通过改进之前的Rosetta通用力场(RosettaGenFF)来实现的,得到了改进的力场RosettaGenFF-VS。基于这一新力场,作者开发了RosettaVS虚拟筛选方法。并且,作者创建了一个高度可扩展的开源AI加速虚拟筛选平台OpenVS,集成了药物发现所需的所有组件。
人工智能加速的虚拟筛选平台开发
作者之前开发的Rosetta GALigandDock是一种基于物理力场RosettaGenFF的配体对接方法,该方法在配体对接准确性方面表现优异。它能够精确地模拟蛋白质-配体复合物,允许受体侧链的完全灵活性和骨架的部分灵活性。然而,它并不适用于大规模虚拟筛选,原因包括:
其无法准确建模某些官能团(因为原始方法仅测试了数十万种化合物,而非本研究中的数十亿);
缺乏一个熵模型来准确排序与同一靶标结合的不同化合物。
此外,使用基于物理的虚拟筛选方法对数十亿化合物进行逐一对接的成本过高。
为了实现对超大规模化合物库的筛选,作者采取了两种策略。
首先,作者开发了一个修改后的对接方法RosettaVS,其中包含两种高速配体对接模式:虚拟筛选快速模式(VSX)用于初始快速筛选,而虚拟筛选高精度模式(VSH)则用于对初始筛选中的最佳命中物进行最终排序。
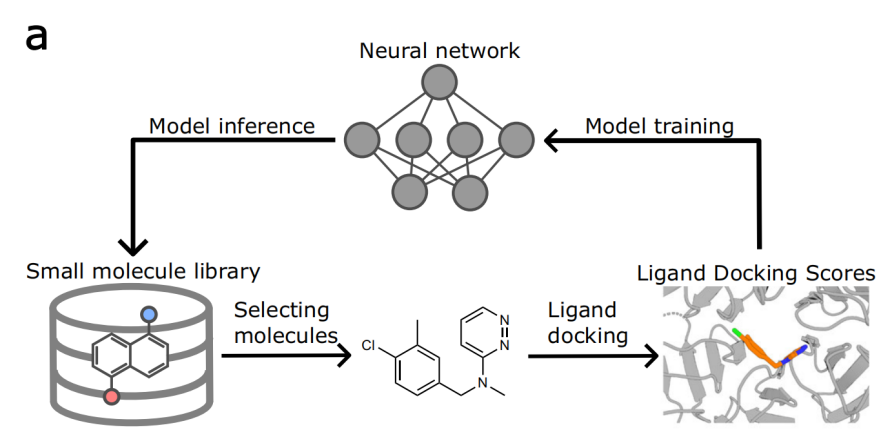
虚拟筛选模型框架
然而,即使经过这些加速处理,对数十亿化合物进行对接仍然成本高昂。基于最近的研究,作者开发了一个开源的虚拟筛选平台(OpenVS),它利用主动学习技术,在对接计算期间同时训练针对特定靶标的神经网络,以有效筛选和选择最有希望的化合物进行高成本的对接计算。该平台还被设计为高度可扩展和并行化,适用于大规模虚拟筛选。
RosettaVS在虚拟筛选基准测试中表现出最先进的性能
作者首先使用了2016年评分函数的比较评估(CASF2016)数据集来评估RosettaGenFF-VS的性能。CASF2016包含285个多样化的蛋白质-配体复合物,是专为评分函数评估设计的标准基准。它提供了所有小分子结构作为虚假结构,有效地将评分过程与分子对接中固有的构象采样过程分离开来。
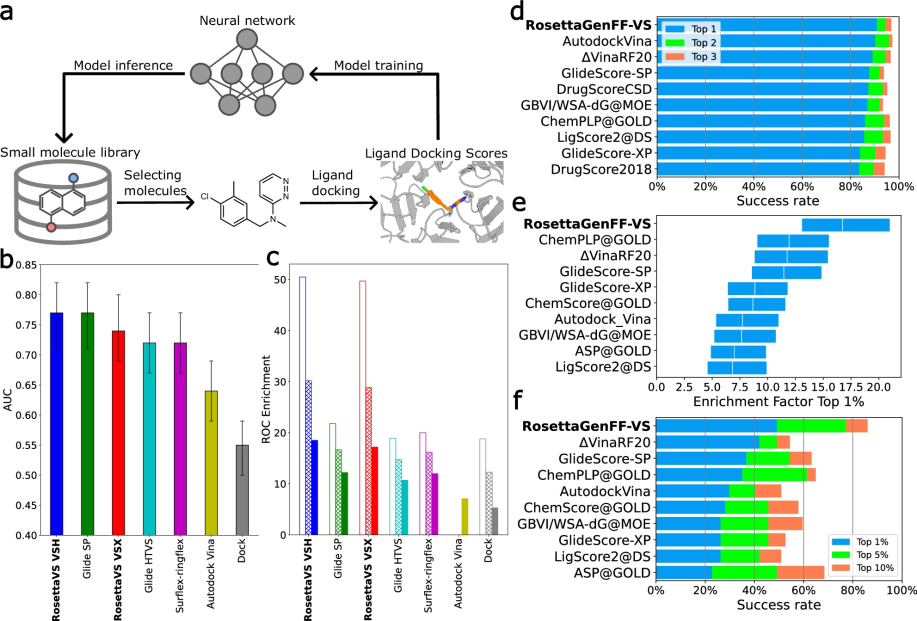
图 1
作者使用对接能力测试来评估对接准确性,使用筛选能力测试来评估筛选准确性。正如图1d所示,RosettaGenFF-VS在准确区分天然结合构象与虚假结构方面表现领先。
接下来,作者进行了筛选能力测试以评估评分函数在大量负向小分子中识别真正结合物的能力。筛选能力测试中使用了两个指标:富集因子(EF)和成功率。
EF衡量对接计算在给定X%恢复化合物的截止值下识别出真正阳性物的早期富集能力。成功率衡量在数据集中所有目标蛋白的1%、5%或10%排名中将最佳结合物放置在其中的能力。
在图1e中,RosettaGenFF-VS的1%富集因子(EF1% = 16.72)明显优于次优的方法(EF1% = 11.9)。同样,图1f展示了RosettaGenFF-VS在前1/5/10%排名分子中识别出最佳结合小分子的能力。
为了进一步评估RosettaVS方法中VSX和VSH模式在虚拟筛选中的表现,作者在实用虚假目录(DUD)数据集上进行了测试。DUD数据集包含40个与药物相关的蛋白质目标和超过10万个小分子。在图1b, c中,作者使用了两个常见指标:AUC(曲线下面积)和ROC(接收者操作特征)富集,来量化虚拟筛选性能。
ROC曲线广泛用于评估虚拟筛选性能,其目标是区分活性化合物和非活性化合物。AUC用于评估一种方法在区分活性化合物与非活性化合物方面的整体性能。ROC富集在给定X%假阳性率下测量真正阳性的富集,弥补了富集因子的一些不足。
从实验结果来看,RosettaVS成为最优的虚拟筛选方法。
针对KLHDC2泛素连接酶的小分子命中化合物发现
为了展示新开发方法的有效性,作者针对人类KLHDC2泛素连接酶进行了大规模虚拟筛选。KLHDC2目前尚未与任何已知的药物样小分子结合。作为CUL2-RBX1 E3复合物的底物受体亚基,KLHDC2具有KELCH重复螺旋桨结构域,能够以纳摩尔级亲和力识别其底物的二甘氨酸C端降解标记。
作者利用OpenVS平台和RosettaVS中的VSX模式对Enamine-REAL库进行了虚拟筛选,该库包含约55亿种可购买的小分子,合成成功率为80%。该方法在每次对接迭代后都会发现具有更高预测结合亲和力的化合物。当虚拟筛选达到第十次迭代的最大次数时,作者停止了筛选,这是因为在第八次迭代之后没有发现新的全局最低能量结构。随后,作者使用RosettaVS中的VSH模式对虚拟筛选中排名前50,000的小分子进行了重新对接。整个计算在一个配备3000个CPU和一个RTX2080 GPU的本地高性能计算集群上在一周内完成。约有600万个(占总数0.11%)来自Enamine REAL库的化合物进行了对接。
作者最终选择了37种化合物进行化学合成,其中29种化合物成功合成,并在AlphaLISA竞争试验中进行了表征,测试了每种化合物与含有二甘氨酸的SelK C端降解肽竞争结合KLHDC2的能力。其中,化合物29(C29)在置换降解肽方面表现突出,IC50约为3 μM(见图2a, c)。
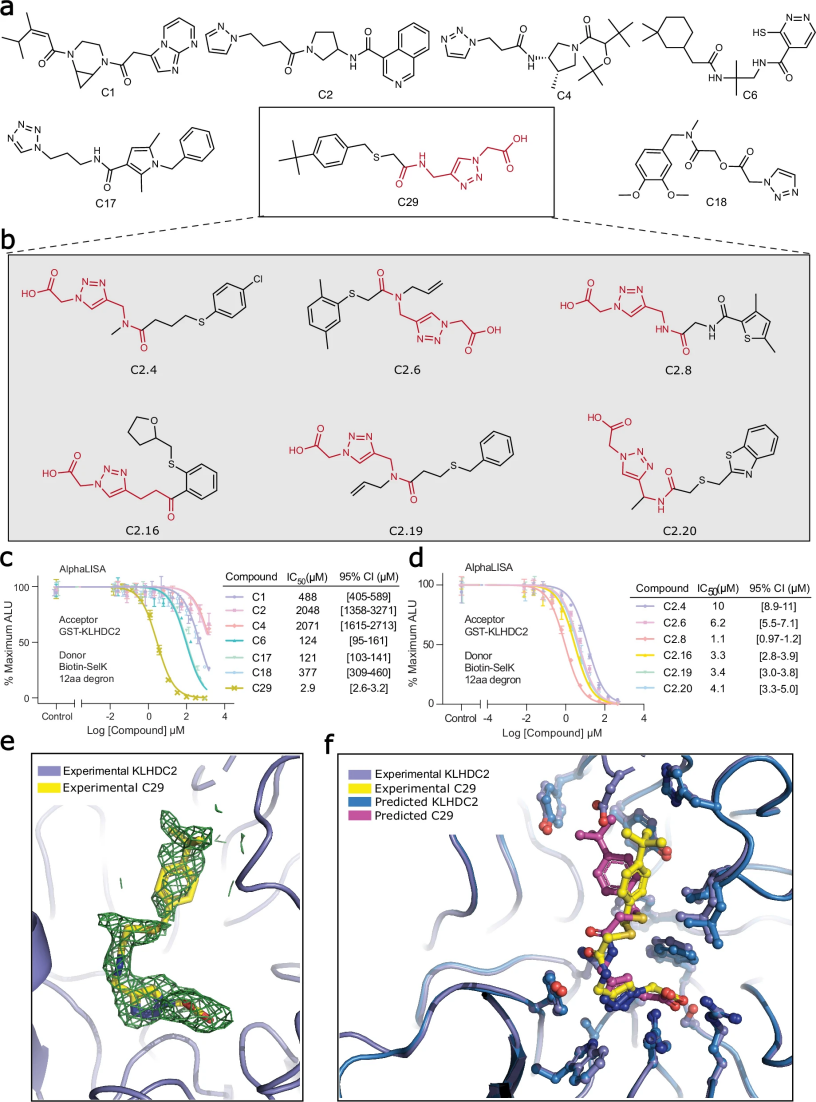
图 2
为了揭示C29的结合模式,作者将apo KLHDC2晶体与C29共浸泡,确定了KLHDC2-C29复合物的结构,分辨率为2.0 Å。C29结合在降解标记结合口袋中,其远端羧基与KLHDC2中两个关键的精氨酸残基(Arg236和Arg241)和一个丝氨酸残基(Ser269)相互作用,这些残基参与识别降解标记的C端。该化合物的羧基旁的三唑基团嵌在三个芳香残基(Tyr163、Trp191和Trp270)之间,并通过NH…N氢键稳定。通过在Lys147与小分子的中心羰基之间形成了一个氢键,并且叔丁基苯基基团直接堆积在降解标记结合口袋的辅助腔中,C29与E3的相互作用进一步增强了。
与C29的两端相比,中间的二甲基硫醚连接子显示出较差的电子密度,表明其具有较高的结构柔性(见图2e)。总体而言,如图2f所示,C29的结合模式与二甘氨酸C端降解标记的结合方式非常相似,其结合姿态与预测结果高度吻合。
在发现初始命中化合物后,作者将探索范围扩大到ZINC22库,该库中包含大约41亿个已准备好进行对接的三维格式小分子。作者在ZINC22库中对乙酰氨基甲基三唑乙酸骨架(图2a, b中的红色突出显示的二维结构)进行了子结构搜索,识别了约381,567个化合物。利用GALigandDock的柔性对接模式进行对接,从通过上述所有筛选的前100个结构中手动挑选了21个化合物。
这些化合物被合成并在基于AlphaLISA的竞争试验中以C29作为阳性对照测试了它们的活性,其中有6个化合物显示出个位数微摩尔级的IC50,进一步验证了该方法的有效性。
NaV1.7 VSD4的小分子拮抗剂的发现
为了评估该虚拟筛选方法的更广泛适用性,作者将其应用于人类电压门控钠通道hNaV1.7。作者专注于电压传感结构域IV(VSD4),该结构域与NaV通道的快速失活有关,并包含一个可稳定通道失活状态的小分子受体位点。作者使用相同的虚拟筛选方法对ZINC22库进行了筛选。与KLHDC2的筛选类似,每次迭代后都会发现具有更高预测结合亲和力的新化合物,前0.1%的化合物的预测结合亲和力从第一次迭代的−10.8 kcal/mol提高到最后一次迭代的−18.2 kcal/mol。在第七次迭代后,由于最高预测结合亲和力达到收敛,虚拟筛选停止。作者使用RosettaVS中的VSH模式重新对虚拟筛选中排名前100,000的小分子进行了对接,以考虑受体结构的灵活性。大约450万个(占总数0.11%)来自ZINC22库的化合物进行了对接。
首先,作者对排名前100,000的小分子进行了聚类,然后对前1000个聚类代表分子应用了筛选标准。共有160个通过聚类和筛选的分子进行了手动检查。为了确保选择的化合物在化学结构上的新颖性,作者特意排除了那些包含已知芳基磺酰胺活性基团或结构上类似于抗组胺药或β受体阻滞剂的分子。最终,选择了10种与ChEMBL数据库中已知的NaV1.7抑制剂的Tanimoto相似性小于0.33的分子进行合成。
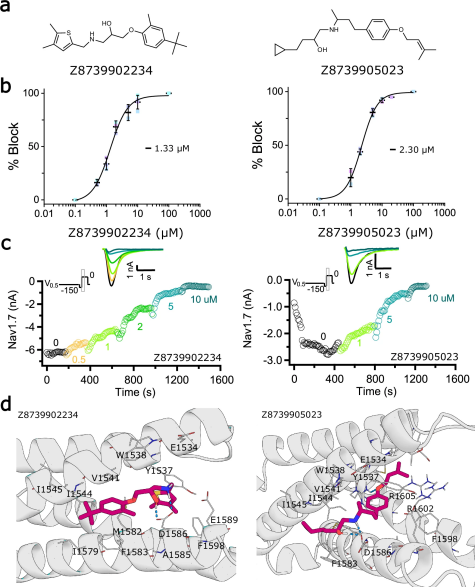
图 4
在这些化合物中,9种被成功合成并使用在HEK-293细胞中稳定表达的hNaV1.7通道进行了全细胞膜片钳电生理测试以测定其活性。化合物Z8739902234展示了最高效力,对NaV1.7的IC50 = 1.3 μM,表现出依赖失活状态的特性(见图4)。有四个化合物的IC50值优于10 μM,命中率为44.4%。
讨论
本文提出了一种最先进的基于物理的虚拟筛选方法,整合到一个综合且可扩展的平台中,用于大规模虚拟筛选和先导物发现。该方法成功发现了针对E3连接酶KLHDC2的7个结合物,以及针对电压门控钠通道NaV1.7 VSD4的4个结合物。RosettaGenFF-VS和RosettaVS在CASF2016和DUD基准测试中表现优异,归功于其高对接准确性和对多样化蛋白质口袋的适应性。尽管该方法已经领先,但未来可以结合GPU加速和深度学习,以提高筛选的准确性和效率。
编译|于洲
审稿|王梓旭
参考资料
Zhou G, Rusnac D V, Park H, et al. An artificial intelligence accelerated virtual screening platform for drug discovery[J]. Nature Communications, 2024, 15(1): 7761.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢