DRUGAI
今天为大家介绍的是来自北京大学张铭团队的一篇论文。化合物的生物活性在药物开发和发现中起着重要作用。现有的机器学习方法由于每个实验中的化合物数量较少且实验之间的测量不兼容,在生物活性预测方面表现出较差的泛化能力。本文中,作者提出了ActFound,一种基于ChEMBL数据库中160万实验测得的生物活性数据和35,644个实验训练的生物活性基础模型。ActFound的核心理念是使用成对学习来学习同一实验中两个化合物之间的相对生物活性差异,从而绕过实验之间的不兼容性。ActFound还利用元学习来共同优化所有实验的模型。在六个真实世界的生物活性数据集上,ActFound展示了精确的域内预测能力,并在不同的实验类型和分子骨架上表现出很强的泛化能力。作者还证明了ActFound可以作为领先的基于物理计算工具FEP+(OPLS4)的精确替代方案,只需使用少量数据点进行微调就能实现相当的性能。该有希望的结果表明,ActFound可以成为一种有效的化合物生物活性预测基础模型,为基于机器学习的药物开发和发现铺平道路。

化合物的生物活性评估在药物开发中至关重要,涵盖了化合物的多种性质,如与靶点的相互作用、对生物系统的影响和治疗效果。生物活性预测可以帮助科学家快速从大量候选化合物中筛选出理想的化合物,并减少昂贵且耗时的实验需求。传统的基于物理的计算方法依赖于难以获得的三维靶蛋白结构,并且在精度与计算成本之间存在权衡。尽管自由能微扰(FEP)等方法可以提供准确的预测,但其计算资源消耗巨大,难以大规模应用。
近十年来,深度学习在生物活性预测中显示了巨大潜力。然而,现有方法存在两个主要局限性:每个实验中的化合物数量有限,无法训练出针对特定实验的模型;不同实验的单位、数值范围和测量标准不一致,导致模型泛化能力差。
为了解决这些问题,作者提出了ActFound,一种生物活性基础模型。该模型结合了元学习和成对学习两种技术,能够处理实验之间的不兼容性,形成一个通用的生物活性预测模型。作者通过多个生物活性预测任务验证了ActFound的性能,包括域内和跨域的生物活性预测、与FEP工具的比较以及癌症药物反应预测。实验结果表明,ActFound不仅在多种实验类型中表现优异,还能广泛应用于药物开发的各个阶段。
模型部分
如图1所示,ActFound是一种针对生物活性的基础模型。作者开发ActFound以应对生物活性预测中的两个主要挑战:每个实验中标记数据的有限性以及不同实验之间的不兼容性。
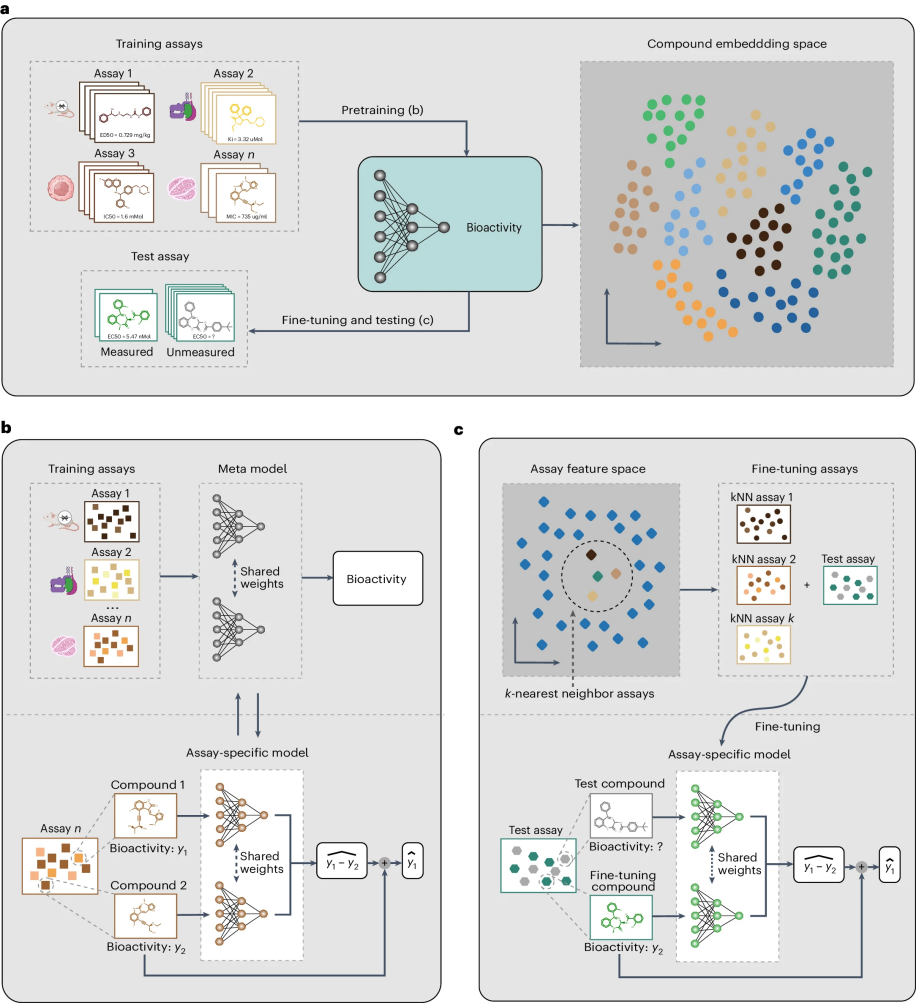
图 1
ActFound有效利用了大量可用的实验和化合物,通过元学习和成对学习(图1a)提供了一个预训练的生物活性模型。在预训练阶段,ActFound的参数在多种实验中通过双层优化过程进行交互更新,该过程包括用于微调特定实验的内循环和基于微调模型表现更新模型参数的外循环。因此,ActFound对各种实验的更新更加敏感,并且可以快速适应新实验。
ActFound的骨干是一个孪生网络,它计算两个化合物之间生物活性值的相对差异。通过关注相对活性,可以绕过不同实验之间生物活性值不兼容的问题。最终的生物活性值可以基于预测的相对生物活性值进行重构(图1b)。
最后,ActFound使用少量具有实验生物活性的化合物进行微调,然后可以预测未测量化合物的生物活性(图1c)。作者证明了ActFound可以精确执行域内生物活性预测,并在跨域中表现出强大的泛化能力。
精确的域内生物活性预测
作者首先评估了ActFound在ChEMBL和BindingDB的不同类型实验中的生物活性预测性能。这些实验涵盖了药物发现的各个方面,评估了化合物的多种性质(例如,结合、功能,以及吸收、分布、代谢、排泄和毒性)。根据之前的研究,作者在16-shot设置中评估了模型性能,即每个实验中使用16种化合物微调模型,剩余化合物用于评估。
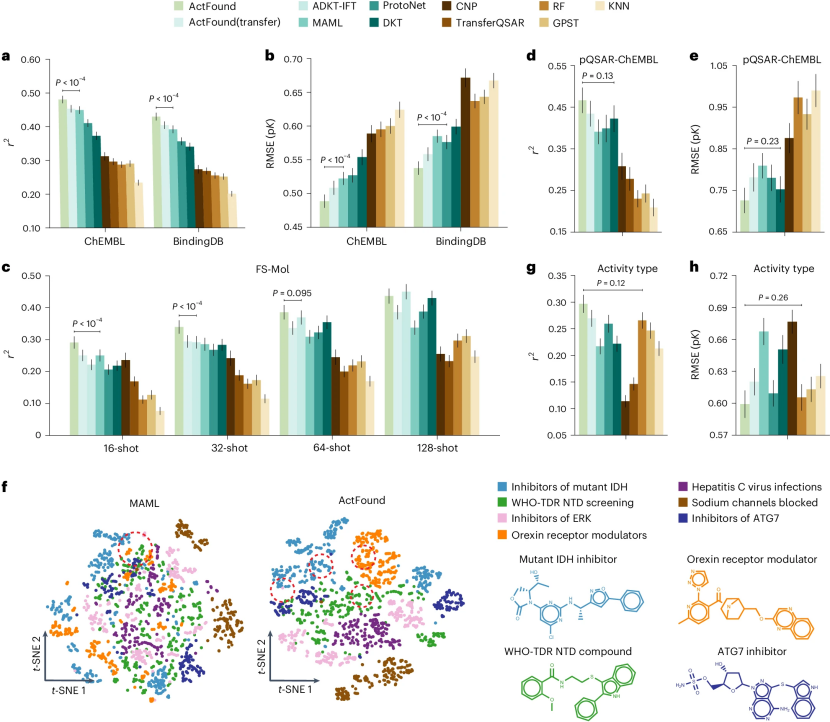
图 2
作者发现,ActFound在两个数据集中都优于所有竞争方法,且r2值(图2a)和RMSE(图2b)表现出色。ActFound明显优于传统的元学习方法MAML和ProtoNet,这表明成对学习在学习相对生物活性值方面的有效性。其次,作者发现ActFound优于其使用迁移学习而非元学习的变体,这表明元学习在多样化实验中训练生物活性基础模型的优势。
随后,作者在FS-Mol上评估了该方法,该数据集是从ChEMBL中筛选出的仅包含摩尔浓度单位的实验。FS-Mol数据集规模较小,仅包含ChEMBL中13.8%的实验和30.8%的独特化合物。作者使用从16到128个微调化合物评估了性能(图2c),发现ActFound在低资源设置(16-shot和32-shot)中表现最佳,并在128-shot设置中表现相当。
在使用pQSAR-ChEMBL的更具挑战性和现实性的数据划分设置时,微调化合物与测试化合物在结构上不相似,ActFound表现最佳(图2d, e),展示了其对未见过的分子骨架的泛化能力。
为了更好地理解ActFound在学习相对生物活性差异方面的优越性能,作者比较了ActFound和MAML获得的化合物嵌入(图2f)。与MAML相比,ActFound的嵌入展示了更明显的模式,证明了其高质量的嵌入。在所有实验中,作者发现ActFound在未包含百分比(%)的实验中表现最差(图2g, h)。尽管如此,ActFound仍然超越了其他迁移学习和元学习方法,再次表明其在不同单位下的优越泛化能力。
跨领域的改进泛化能力
作者系统地评估了它在跨领域设置中的泛化能力:模型在一个领域的实验中进行训练,并在另一个领域的实验中进行微调。结果总结如图3所示:
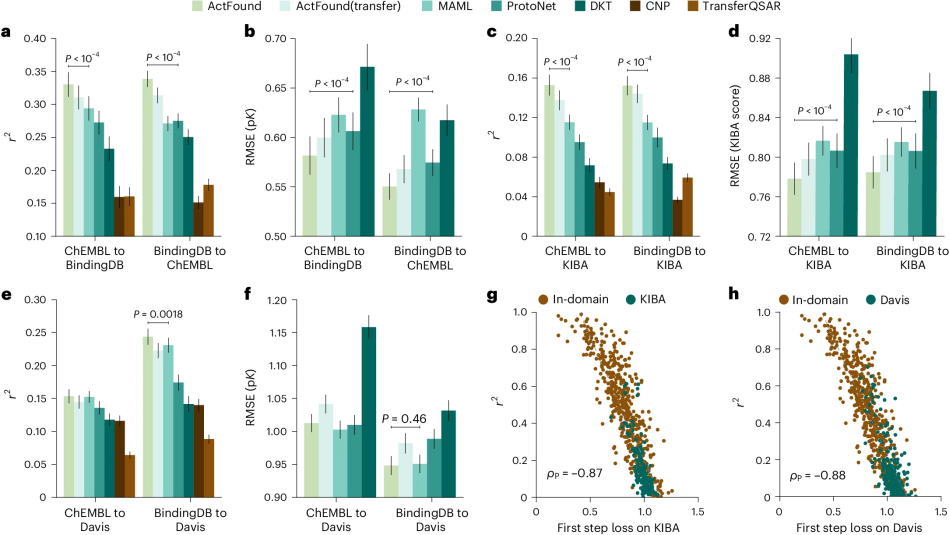
图 3
与域内设置相比,ActFound在跨领域设置中性能略有下降,表明这是一个更具挑战性的环境。然而,ActFound在所有数据集的所有指标上都优于所有比较方法。在ChEMBL到BindingDB的实验中,ActFound的性能达到了BindingDB域内实验的76.7%;而在BindingDB到ChEMBL的实验中,其性能达到了ChEMBL域内实验的70.44%。作者再次将其归因于ChEMBL中较多的实验数量,这为成对元学习框架提供了更多的任务。
作者还利用了两个独立的激酶抑制剂数据集——KIBA和Davis,来评估ActFound的跨领域预测性能。作者的方法在这两个数据集中都取得了总体最佳表现(图3c-f),证实了成对学习和元学习的优势。
作者发现测试实验的性能与第一次优化步骤的损失值(ρP = -0.88)强烈相关(图3g, h)。直观上,这种强相关性反映了该测试实验可能从元学习中获益的程度,因为较小的损失意味着更容易适应新实验,从而帮助最终用户识别出可以通过ActFound改进预测的实验。
FEP的机器学习替代方案
为了进一步展示ActFound在药物设计中的实用性,作者将其应用于两个FEP基准测试。FEP是一种重要的基于物理的计算方法,在药物发现中有许多成功的应用。它用于指导药物结构的优化,以提高生物活性。
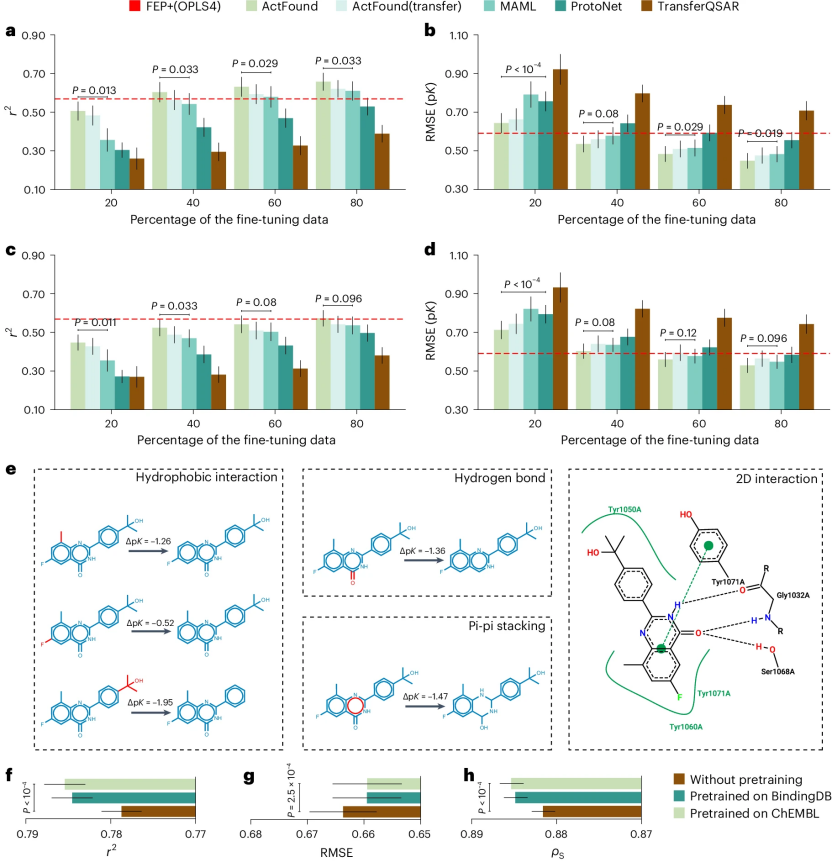
图 4
如图4a, b所示,作者使用不同比例的微调数据(从20%到80%)比较了所有方法的性能。ActFound在使用不同比例的微调数据时始终优于竞争方法。当使用较少数据时改进更大,这表明ActFound可以更有效地利用新实验中的标记数据。更重要的是,作者发现通过使用40%的数据进行微调,该模型可以超过FEP计算工具FEP+(OPLS4)的性能,这是一种由Schrödinger开发的领先商业工具。
如果没有实验测得的数据,ActFound还可以使用FEP+(OPLS4)计算的数据来微调模型。作者在图4c, d中对此进行了评估:作者使用FEP+(OPLS4)的结果进行微调,而没有结合任何实验测得的生物活性数据。ActFound仍然优于所有比较方法。在使用80%的化合物进行微调时,作者的方法的r²值几乎与FEP+(OPLS4)相当,且RMSE值甚至比FEP+(OPLS4)高出10.4%。
最后,为了进一步了解ActFound的良好性能,作者对一个TNKS2靶点实验进行了案例研究,展示了给定化合物的不同修饰导致的生物活性变化。作者首先使用ProteinsPlus确定了几个潜在的相互作用,然后对生物活性最佳的化合物进行了修饰,并预测了这些修饰引起的生物活性变化(图4e)。第一个和第二个化合物的实验和预测活性变化(ΔpK)之间的平均绝对误差为0.351 pK,表明预测的活性变化与体外实验结果接近。这个案例研究揭示了ActFound如何利用成对学习捕捉功能基团和骨架的结构差异,以指导药物优化。
讨论
作者提出了ActFound,这是一种几乎适用于所有实验的生物活性基础模型。ActFound结合了元学习和成对学习,充分利用多样化实验中的丰富信息,提升了少样本生物活性预测的性能。在域内和跨域预测中,ActFound表现出色,并在FEP基准测试和新细胞系的药物敏感性预测中展示了实用性。与现有方法相比,ActFound不依赖测量单位,能泛化到不同实验类型,并通过预测相对生物活性提升预测准确性。尽管ActFound存在未考虑实验元数据和简单分子指纹特征的限制,但其表现优异,未来可通过引入预训练模型进一步优化。
编译 | 于洲
审稿 | 曾全晨
参考资料
Feng B, Liu Z, Huang N, et al. A bioactivity foundation model using pairwise meta-learning[J]. Nature Machine Intelligence, 2024: 1-13.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢