论文题目:
TMO-Net: an explainable pretrained multi-omics model for multi-task learning in oncology
今天给大家介绍一篇发表在Genome Biology上的文章《TMO-Net: An Explainable Pretrained Multi-omics Model for Multi-task Learning in Oncology》。在肿瘤研究中,整合多组学数据来进行多任务学习越来越重要,因为它能够更全面地理解癌症的生物学复杂性。TMO-Net提供了一种全新的方法,通过一个预训练的多组学模型来同时完成多种任务,如癌症分型、预后预测、药物响应预测。作者强调了模型的可解释性,揭示了不同组学特征对模型决策的贡献。本文在多组学数据的整合与分析上进行了深入探讨,并通过在多个公共肿瘤数据集上的实验,验证了TMO-Net在多个任务上的优越表现。
随着肿瘤学研究的深入和高通量技术的发展,研究人员能够同时获得不同组学层面的海量数据,如基因组、转录组、表观基因组和代谢组数据。这些多组学数据为肿瘤的诊断、分型和治疗提供了丰富的信息,但如何有效整合和利用这些异构数据仍是重大挑战。 TMO-Net提出了一种新的多任务学习框架,通过整合多组学数据来实现癌症分型、预后预测、药物响应预测等任务。TMO-Net另一个显著特点是引入了可解释性技术,利用Integrated Gradients(IG)方法来评估各个组学特征对模型决策的贡献,这使得研究人员能够更好地理解模型背后的生物学意义。 本研究的重要性在于通过一个统一的框架将多组学数据整合到一起,并且提供了可解释性方法来揭示不同组学层面的影响。这不仅提高了预测精度,也促进了癌症生物标志物的发现,有助于个性化治疗的发展。
TMO-Net模型专为利用大规模泛癌症多组学数据集进行预训练而设计,使其适用于广泛的癌症相关深度学习任务(图1a)。TMO-Net模型的架构包括自模态变分自编码器、跨模态变分自编码器和交叉融合模块(图1b、c)。 TMO-Net模型使用处理过的大规模泛癌症多组学数据集进行预训练,包括基因突变、mRNA表达、拷贝数变异(CNV)和DNA甲基化模态。TMO-Net采用了多个变分自编码器(VAEs)来捕捉自模态和跨模态特征的关联,并集成了“交叉融合模块”,促进缺失模态的推断。这些编码器协同工作,为每种组学类型生成联合嵌入,然后将其融合生成综合的多组学样本联合嵌入。 此外,TMO-Net通过多模态编码器学习的联合嵌入被进一步应用于适应各种下游癌症任务和模型,尤其是用于独立且规模有限的癌症患者队列(图1d)。同时,作者还整合了模型解释方法,揭示特定组学分子特征与预测结果的相关性,帮助识别与癌症相关的多组学机制(图1e)。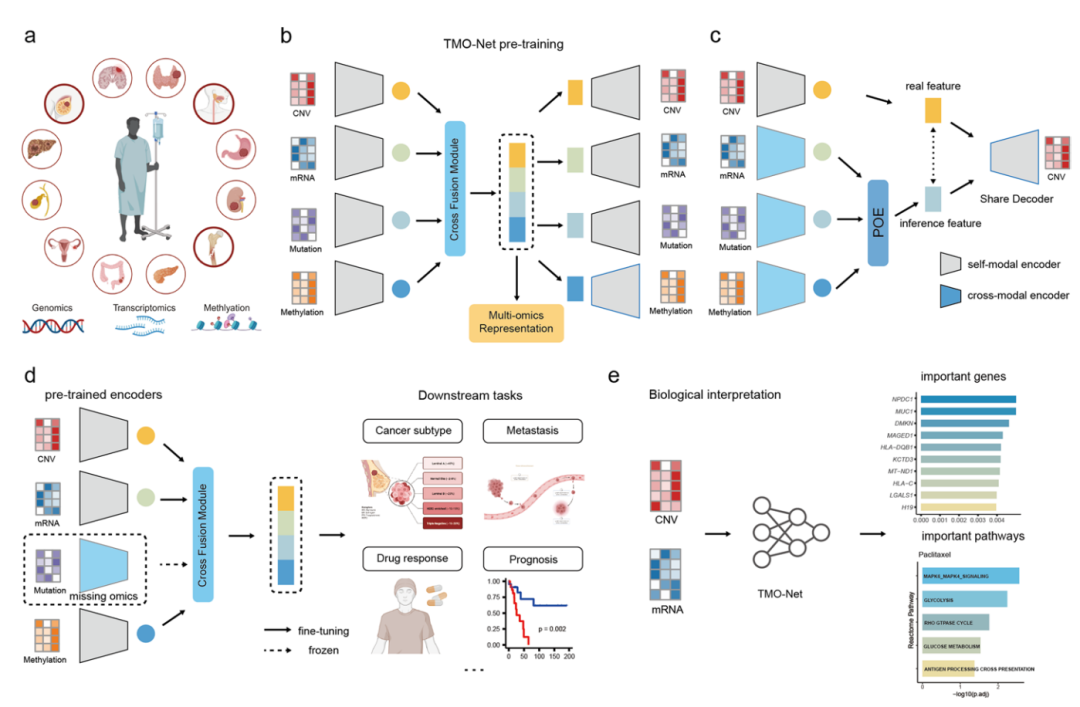
图1. TMO-Net模型概述
TMO-Net模型的架构包括自模态变分自编码器、跨模态变分自编码器和交叉融合模块:
1.自模态变分自编码器(self-VAE):通过捕捉组学特异性表示并重构目标组学数据,采用重参数化技巧优化变分证据下界(ELBO),以降低高维数据维度并生成嵌入。self-VAE损失定义如下:
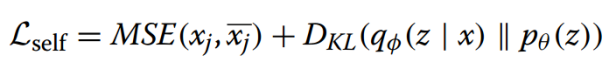
2.跨模态变分自动编码器(cross-VAE):学习不同组学数据间的关联,使用其他组学数据来预测目标组学。输入数据经过降维处理,生成隐变量的高斯分布,用于推断目标组学的表示。
3.交叉融合模块:融合不同模态的嵌入表示,生成联合后验分布,解决缺失模态问题。使用专家乘积模型(PoE)来重建缺失数据,并对齐自模态与交叉模态分布。跨模态损失可以计算为:4.该模型采用了多目标的自监督学习框架进行预训练。其综合损失函数包括以下几个组成部分:(1)self-ELBO loss:学习潜在的方差分布,同时准确重构输入数据。
(2)cross-modal ELBO Loss:这一部分侧重于捕捉不同组学层次之间的关联,使模型能够从其他模态重构特定模态。
(3)判别器损失:该损失迫使模型区分原始数据和重构数据,并对齐不同模态的潜在表示。
(4)对比损失:该损失使同一肿瘤亚型样本嵌入对齐,同时区分其他亚型。
1.TMO-Net的训练分为两个阶段:
(1)自监督预训练:使用处理过的大规模泛癌症多组学数据集进行预训练,涵盖32种癌症类型的8174个样本,包括基因突变、mRNA表达、拷贝数变异(CNV)和DNA甲基化模态。
(2)下游任务模型微调:在微调阶段,设定了多项肿瘤学和临床目标,包括癌症亚型分类和转移预测等。采用前馈神经网络作为分类器,保持参数不变,仅更新下游分类器。使用五折交叉验证,并利用Adam优化器进行训练,学习率为1e-5,权重衰减为1e-4,防止过拟合,忽略预训练阶段的其他损失。
2.评估指标包括:
(1)跨模态推断任务的评估:Pearson相关系数、R²
(2)分类任务:Accuracy、Precision、Recall、F1-score
(3)生存分析任务的评估:C-index、Kaplan-Meier生存曲线、Log-rank test
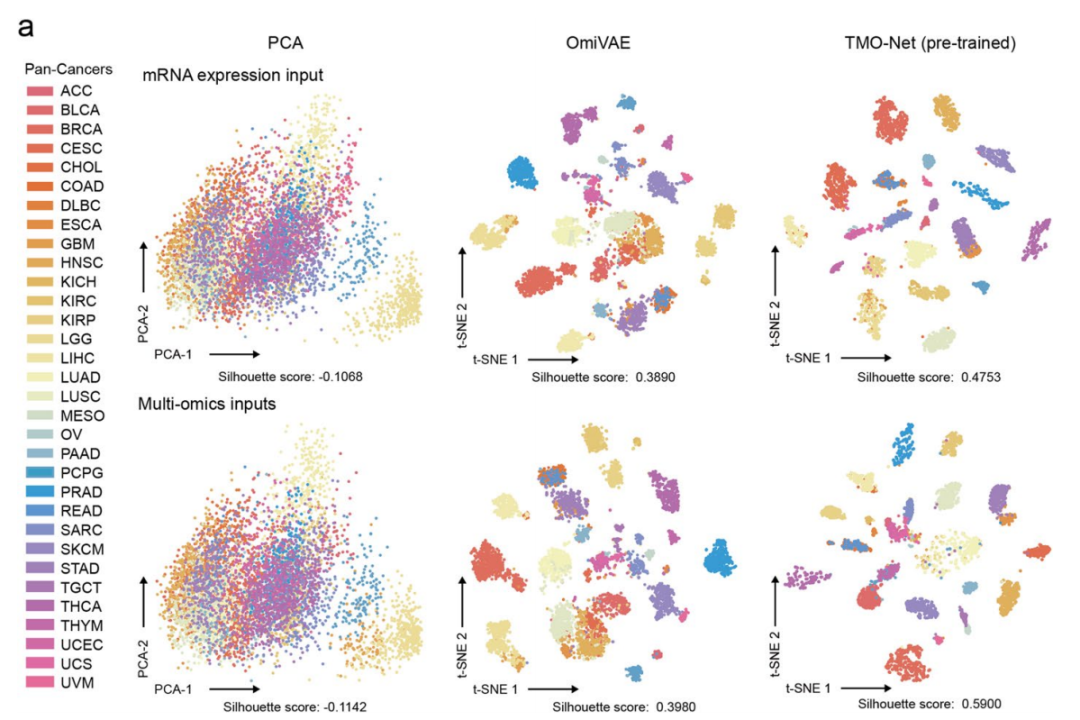
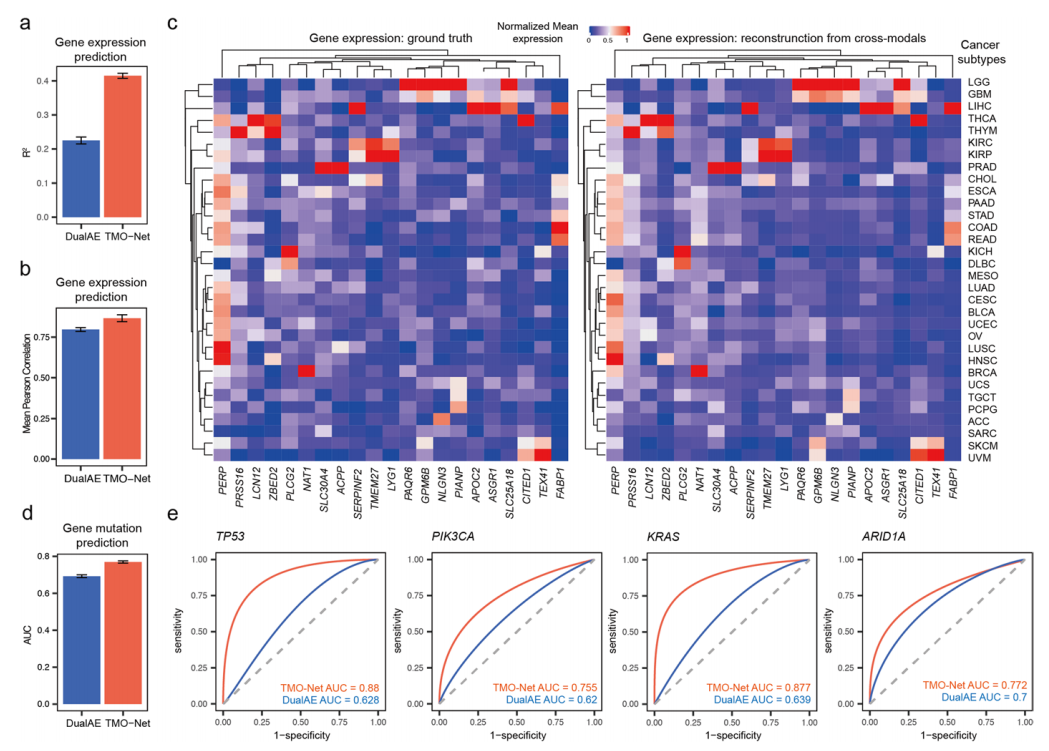
1.TMO-Net模型能够提升癌症表示学习:TMO-Net在区分不同癌症亚型方面优于PCA和OmiVAE,并在分类任务中取得了最高的F1分数0.751。其中基因表达和甲基化数据对癌症分类贡献最大。2.TMO-Net模型实现了跨模态推理:TMO-Net的跨模态学习模块通过跨模态数据再生促进不同模态的交互学习,重构基因表达和突变的能力优于基线双自编码器模型,并在基因突变预测中表现出更高的AUC和ACC。图3. TMO-Net模型在跨组学生成中的能力
3.TMO-Net通过模型预训练实现了乳腺癌亚型预测:利用TCGA-BRCA和METABRIC数据集进行微调和测试。结果显示,TMO-Net在多个分类任务中表现优异,平均F1得分为0.921,显著提升了模型性能。使用IG算法识别了关键基因特征,如TM4SF5和RERG,进一步支持模型的解释性。
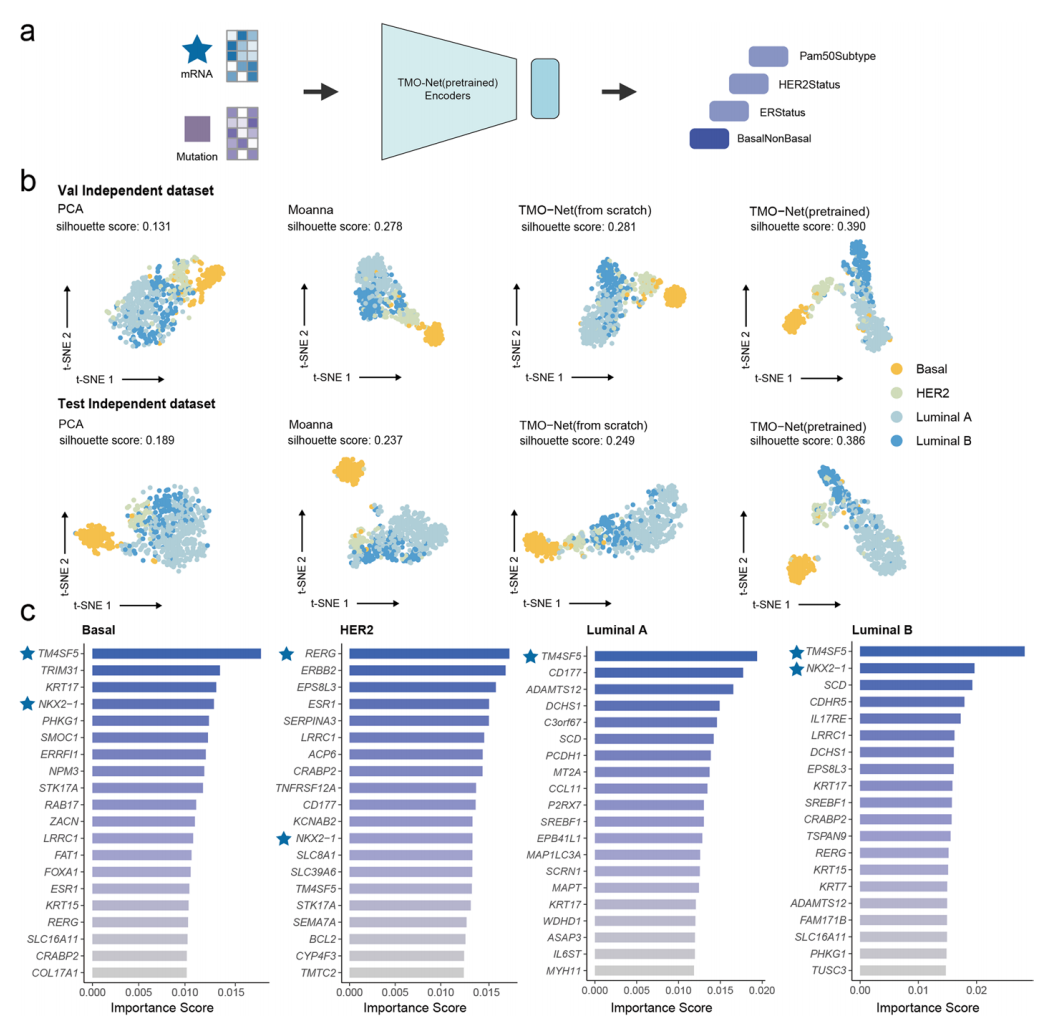
图4. TMO-Net模型可以提高乳腺癌亚型预测的准确
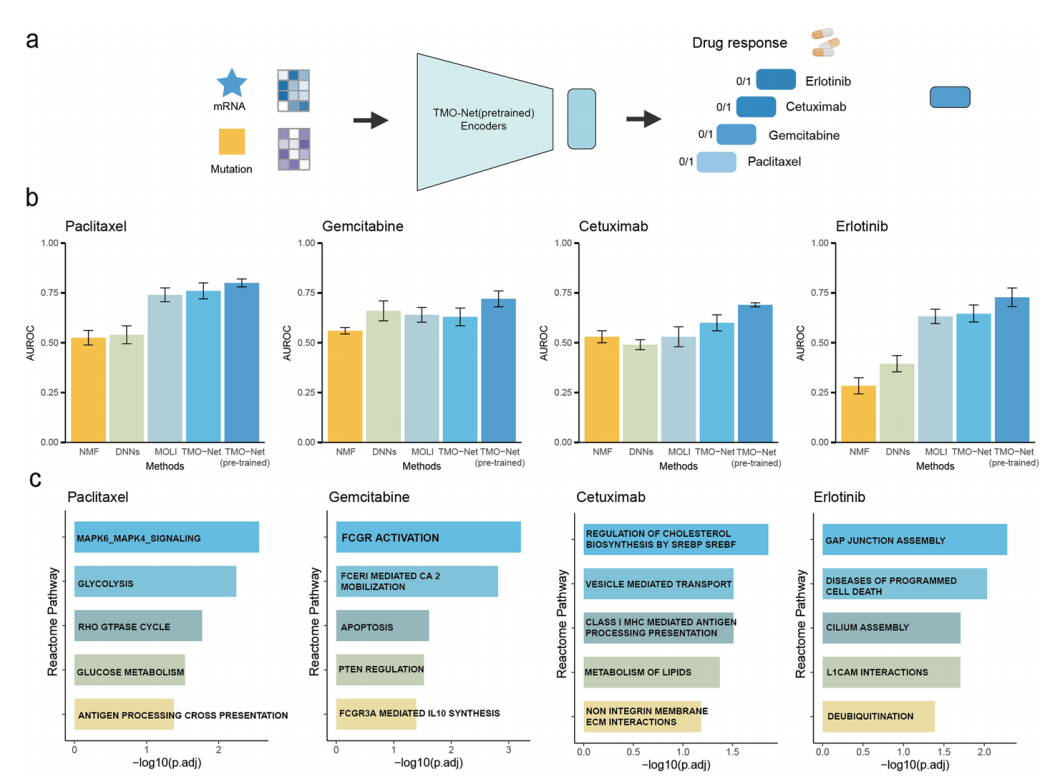
4.TMO-Net增强了对原发癌或转移癌样本的预测能力:通过使用基因表达和甲基化数据,TMO-Net在预测转移肿瘤样本时取得了0.8980的F1分数,优于其他方法。这些结果突显了模型预训练的优势和TMO-Net在多组学癌症数据集中的适应性。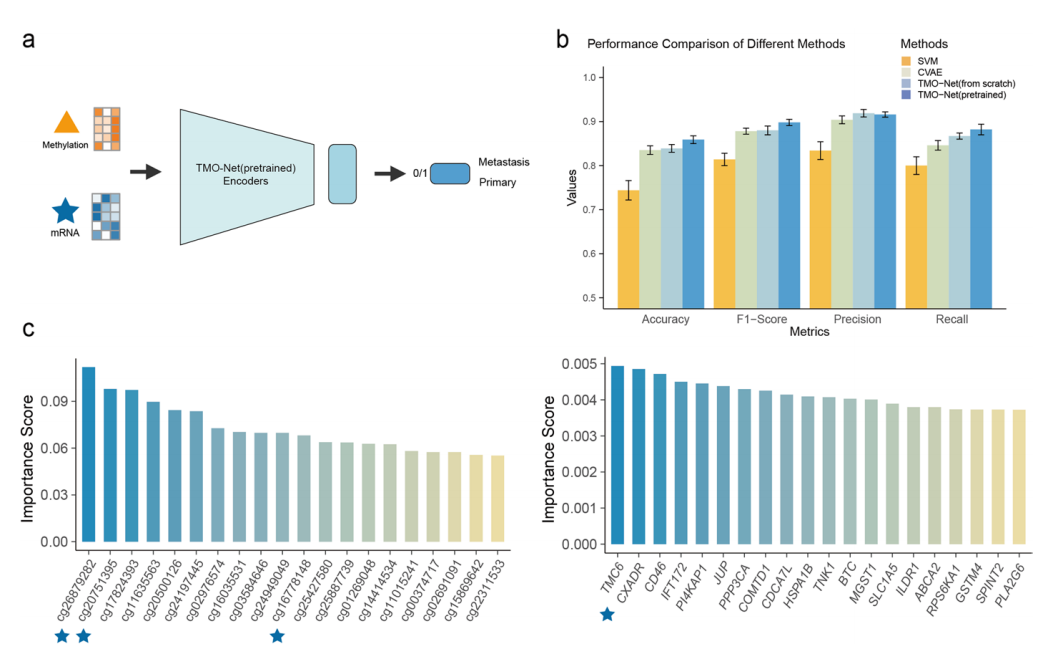 图5. TMO-Net模型能够区分原发癌和转移癌样本5.TMO-Net能够准确预测药物反应:使用TMO-Net模型预测癌症患者的药物反应,基于GDSC项目中的多组学数据进行训练,并使用PDX数据集进行验证。与NMF、DNN等基线模型对比,TMO-Net在药物反应任务中的ROC-AUC评分最高。
图5. TMO-Net模型能够区分原发癌和转移癌样本5.TMO-Net能够准确预测药物反应:使用TMO-Net模型预测癌症患者的药物反应,基于GDSC项目中的多组学数据进行训练,并使用PDX数据集进行验证。与NMF、DNN等基线模型对比,TMO-Net在药物反应任务中的ROC-AUC评分最高。图6. TMO-Net模型在药物反应预测中的应用
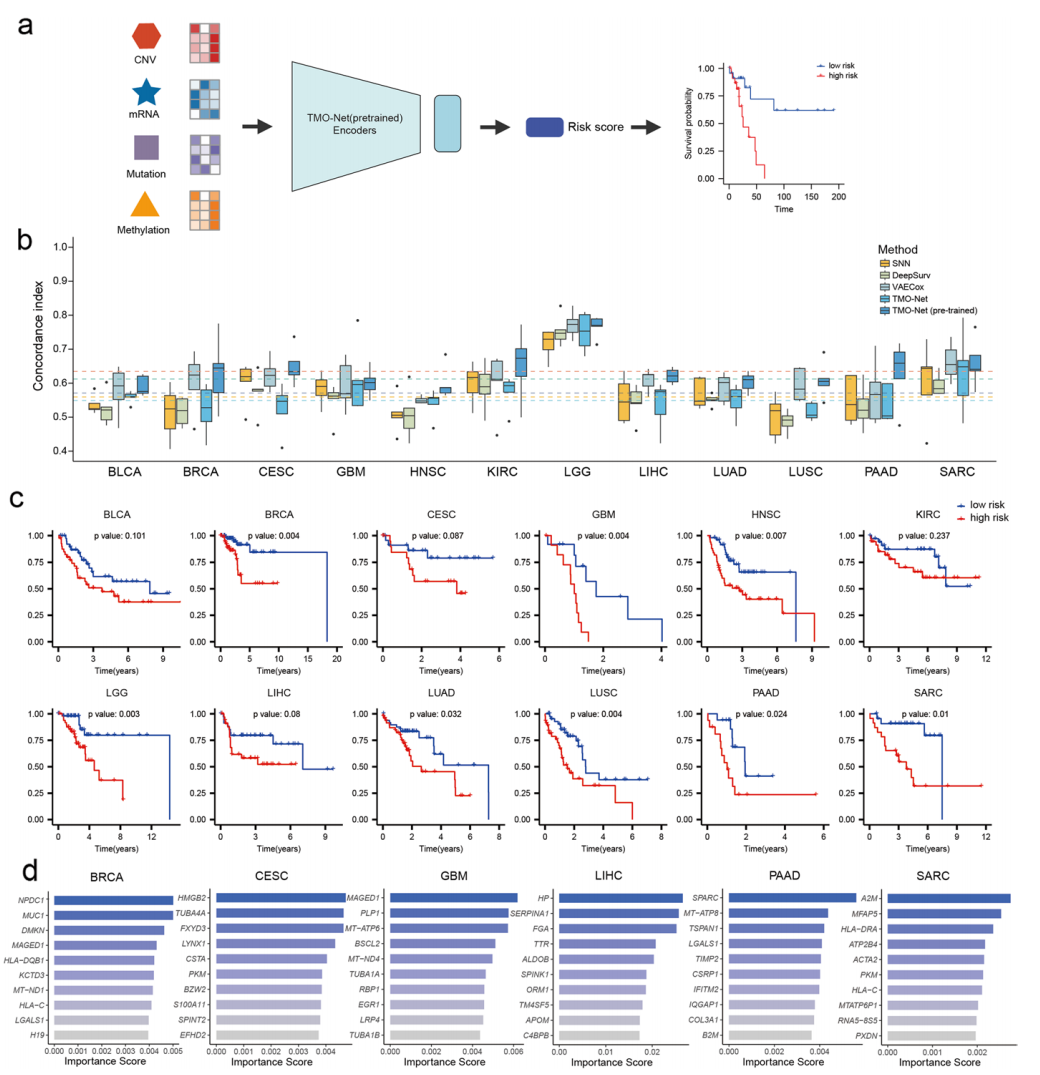
6.TMO-Net通过肿瘤多组学数据提升了预后预测的准确性:使用TMO-Net模型预测了12种不同亚型癌症患者的整体生存状态,建立了基于Cox生存回归网络的预后预测模型。TMO-Net在C-index评分上表现优异。模型有效区分高低风险患者,并通过Kaplan-Meier生存曲线可视化。重要的基因表达特征包括线粒体基因和HLA相关基因,验证了TMO-Net在提取生物学相关基因特征方面的有效性。图7. TMO-Net模型可以预测全癌种的预后情况
TMO-Net模型突出了大规模数据预训练作为未来多组学癌症研究的关键方法,并促进了通用多组学癌症基础模型的发展。通过在多种肿瘤数据集和下游任务中的有效性展示,TMO-Net证明了在多组学数据集上进行预训练可以显著增强表示学习,尤其对于多组学和不完整组学数据。此外,该模型易于迁移到其他基于组学的肿瘤研究应用中,这进一步提升了其在肿瘤学中的价值。这项研究为未来多组学基础模型的开发奠定了基础,这将加速癌症研究进程。
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-024-03293-9
内容中包含的图片若涉及版权问题,请及时与我们联系删除


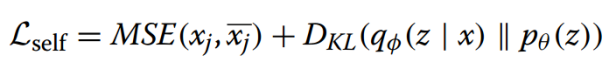
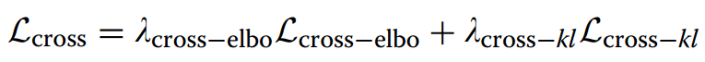





评论
沙发等你来抢