DRUGAI
今天为大家介绍的是来自佐治亚理工学院的Jeffrey Skolnick团队的一篇论文。识别能够中和特定抗原的抗体对于开发有效的免疫疗法至关重要,但这一任务在许多目标抗原上仍然具有挑战性。深度学习为基础的计算方法的兴起为解决这一挑战提供了一条有希望的途径。本文通过两个基准测试评估了深度学习方法在预测针对SARS-CoV-2刺突蛋白受体结合域(RBD)的抗体方面的表现。为了预测抗原–抗体复合物的结构模型,作者采用了三种不同的输入序列比对策略。在最初的测试集中,这些策略在61%的案例中成功生成了排名靠前的显著预测,并取得了47%的成功率。作者的研究结果强调了将深度学习方法与单B细胞测序技术相结合,以提高抗原–抗体相互作用预测精度的潜力。

解析蛋白质在原子水平上如何相互作用形成功能复合物,是理解生物过程的关键。AlphaFold 2(AF2)的成功推动了利用深度学习预测蛋白质复合物的新发展。作者推出了AF2Complex,基于AF2模型预测蛋白质-蛋白质相互作用,并引入了专门的置信度指标来评估多个蛋白质相互作用的可能性。AF2Complex在实际应用中已被验证。例如,它成功预测了大肠杆菌CcmI复合物,并揭示了细胞膜内的意外高置信度相互作用。此外,作者还用AF2Complex检测了T细胞调节相关的蛋白质相互作用。
尽管取得了显著进展,但在预测抗体-抗原相互作用方面仍存在挑战。AF-Multimer虽提升了抗体-抗原复合物的预测精度,但成功率仍低于其他多进化同源序列的蛋白质复合物。抗体-抗原靶标缺乏同源序列,限制了深度学习方法的预测能力。作者评估了AF2Complex在抗体-抗原相互作用预测中的表现,集中研究了两个问题:其一,AF2Complex在预测抗体-抗原复合物结构的准确性如何;其二,AF2Complex能否在混合了已知和随机抗体的库中识别结合特定抗原的抗体。为此,作者使用SARS-CoV-2刺突蛋白的受体结合域(RBD)进行基准测试,依赖于Cov-AbDab数据库中的抗体序列,并设计了一个MSA策略,显著提升了模型的预测能力。
作者展示了针对上述两个问题设计的两项基准测试的结果。第一个测试评估了AF2Complex在预测36种IgG抗体与RBD复合物结构的准确性,并将其与实验确定的真实结构进行比较。第二个测试在混合了471种已知RBD结合抗体和500种从健康个体的B细胞测序研究中随机选择的抗体的抗体池中,评估了模型的敏感性和特异性。
预测靶向刺突RBD不同表位的IgG抗体结构
作者从CoV-AbDab数据库中选取了36对已在实验中确定与SARS-CoV-2 RBD结合的IgG抗体(称为PDB36),并验证这些抗体的重链序列是独特的。作者测试了三种输入序列策略:UniProt序列库、RBD结合抗体序列库和随机选取的健康个体抗体序列库。最后,作者还考虑了第四种组合策略,即从三种MSA策略中排名最高的预测中选择接口得分(iScore)最高的模型。每种策略预测50个结构,并根据iScore评估模型质量。
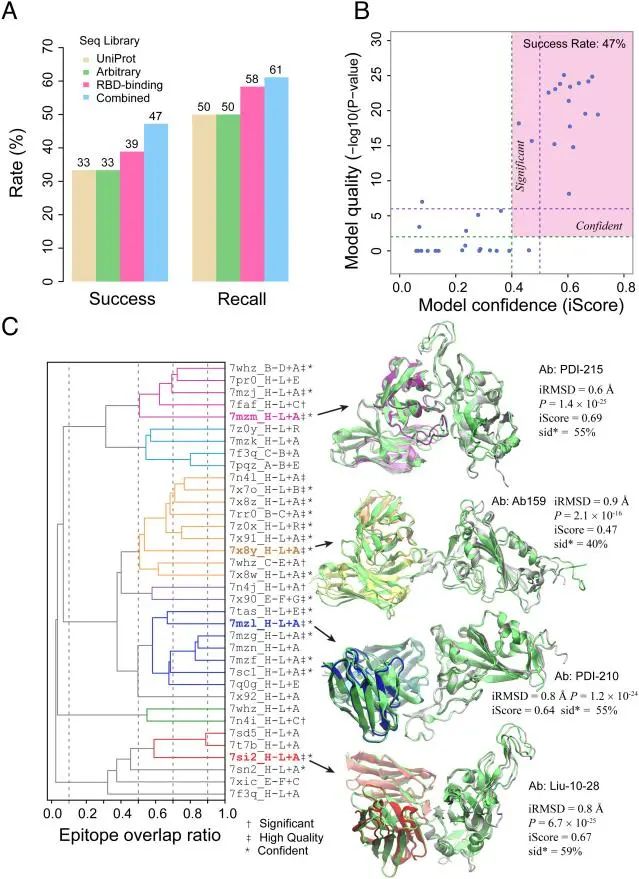
图 1
图1A显示了这四种策略的召回率和成功率。在这些标准下,AF2Complex使用UniProt和随机MSA策略时的召回率为50%(18/36),使用RBD结合策略时提高到58%(21/36),当三种策略结合时,召回率达到最高的61%(22/36)。成功率的表现也类似:UniProt和随机策略的成功率为33%(12/36),RBD结合策略为39%(14/36),三种策略结合时达到47%(17/36)。
图1B展示了组合策略中模型置信度和质量的关系。所有15个高置信度(iScore > 0.5)的预测都表现出高模型质量,平均接口骨架CαRMSD为1.1 Å。此外,18个置信度较高(iScore > 0.4)的预测中有17个质量评估结果显著。有5个低iScore的预测却有较好的模型质量。
鉴于结合RBD的抗体能够识别抗原上的不同表位,作者进一步根据实验确定的表位分析了PDB36的顶级预测。表位的层次聚类显示PDB36包含多样的表位,分为四个主要簇,每个簇的表位与其他三个簇的重叠率不超过10%。AF2Complex在所有四个主要簇和六个子簇中均有显著预测,三大簇和五个子簇中均有成功预测。图1C中四个不同表位簇的成功示例表明,预测结构与实验结构非常接近,接口RMSD小于1 Å,且IS-score显著。
区分结合RBD的抗体和随机选择的抗体
作者测试了该方法是否能在混合了471种已知RBD结合抗体(RBD471)和500种随机选取的抗体池中识别出RBD结合抗体。在基准测试中,主要关注VH和VL区域。测试使用了前述的四种策略,结果显示,使用RBD结合抗体的策略优于其他两种,且组合策略表现最佳。
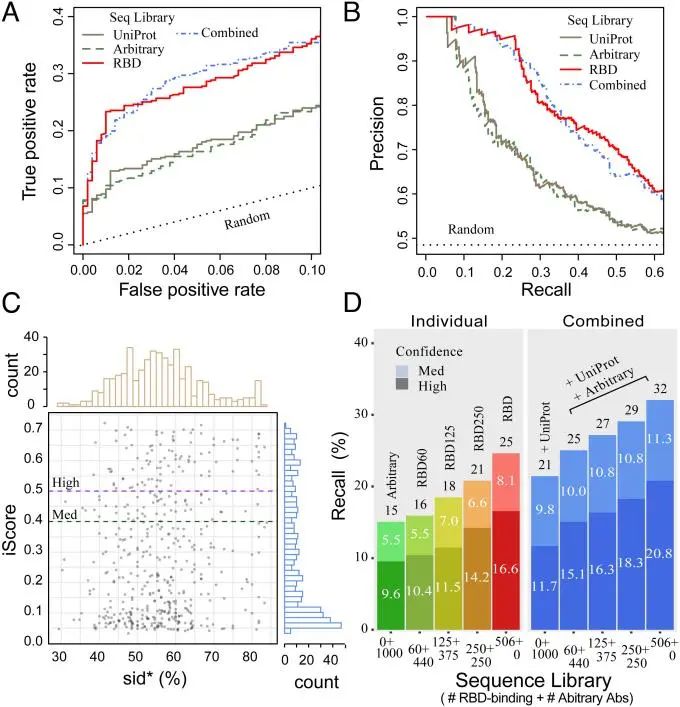
图 2
如图2A所示,在低误报率下,组合策略和RBD结合策略的AUC分别为0.28和0.27,高于UniProt和随机策略的0.17和0.16。如图2B所示,在iScore阈值为0.4时,RBD结合策略的召回率为25%,误报率为2.6%,精确度为90%,显著优于其他策略。组合策略的召回率为32%,但误报率为6.8%,精确度为82%。当iScore阈值为0.5时,RBD结合策略的召回率为17%,精确度为96%。高精确度表明该方法在寻找特定抗原的抗体结合物方面具有潜在的应用价值。
如图2C所示,组合策略中检测到的151个真阳性抗体与训练中可能使用的抗体(PDB235)不同,其中85%的sid*值低于70%,中位数为58%。进一步分析471个RBD结合抗体发现,iScore与sid*之间的相关性较弱,仅为0.17。有28%的高置信度预测是针对sid*值低于50%的难预测目标,而随机对照组的中位数也是50%。相反,48%的sid*值超过80%的情况没有被高置信度地检测到。这表明深度学习方法可以做出基于CDR-H3序列相似性以外的意外预测。为了研究提高预测所需的RBD结合抗体序列数量,作者进一步测试了三种MSA策略:RBD60、RBD125和RBD250,分别从RBD结合抗体集中随机抽取60、125和250个sid* < 70%的抗体序列,并与随机选择的440、375和250个抗体序列配对,构建输入的MSA序列库。图2D中的结果表明,使用更多的RBD结合抗体序列可显著提高预测成功率。
讨论
本研究以SARS-CoV-2刺突蛋白为例,展示了AF2Complex在预测抗体-抗原相互作用方面的能力。通过测试36个抗体-RBD目标,组合三种MSA策略可在超过60%的目标中生成显著的结构预测,并在近一半测试集中获得高置信度的预测。在扩展测试中,RBD结合策略的精确度达90%,组合策略召回率为32%,精确度为82%。使用包含针对同一抗原的抗体序列的MSA库显著提高了预测精度。然而,RBD结构复杂性及糖基化修饰仍对预测构成挑战。
编译 | 于洲
审稿 | 王梓旭
参考资料
Gao M, Skolnick J. Improved deep learning prediction of antigen–antibody interactions[J]. Proceedings of the National Academy of Sciences, 2024, 121(41): e2410529121.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢