DRUGAI
今天为大家介绍的是来自法国里尔大学Guillaume Brysbaert团队的一篇论文。大规模采样使AlphaFold能获得更多样化的结构。结合其高效的置信度排序,这一特性提升了对单体结构和蛋白质复合物的建模能力。然而,这种方法在GPU计算成本和数据存储方面面临挑战。在此,作者介绍MassiveFold,这是一个经过优化且可定制的AlphaFold版本,它能并行运行预测,将计算时间从几个月缩短到几小时。MassiveFold具有可扩展性,既可以在单台计算机上运行,也可以在大型GPU基础设施上运行,充分利用所有计算节点的优势。代码可在该仓库中获取:https://github.com/GBLille/MassiveFold。

随着DeepMind的AlphaFold和AlphaFold蛋白质结构数据库的出现,蛋白质结构空间变得更容易获取。AlphaFold最初仅针对单链蛋白质进行训练和参数化,但后来经过重新训练,使其能应用于多聚体。在证明了大规模AlphaFold采样用于蛋白质-肽段相互作用的可行性之后,AFsample工具成功应用于蛋白质复合物的建模,包括难以建模的纳米抗体复合物,并在CASP15-CAPRI复合物建模类别中排名第一。近期研究表明,大规模采样方法也可以应用于抗原-抗体相互作用等特定结合。对于单体结构,增加采样也有助于研究构象多样性。此外,增加过程中的循环次数可以提高预测质量,但会延长每次预测的计算时间。总的来说,由于无法并行运行,且图形处理单元(GPU)资源和时间方面的要求很高,增加采样和循环次数技术的应用成本很高,即使对于专门的研究团队来说也难以实际操作。
由于这些集群需求量大,承载GPU集群并为高计算需求提供资源的计算基础设施通常会限制作业运行时长,这使得无法进行长时间的AlphaFold计算。对于大型复合物,这些时长限制甚至可能导致无法完成25次预测的"标准"AlphaFold-Multimer运行。
在这篇简讯中,作者介绍了MassiveFold,它结合了AlphaFold的框架、AFsample的增强采样以及ColabFold的附加功能。MassiveFold是一个并行化引擎,可以调用结构预测工具,包括作者随MassiveFold开发的AFsample扩展版本AFmassive,或者ColabFold,然后对结果进行后处理。未来其他支持大规模采样的结构预测引擎也可以整合到MassiveFold中。
MassiveFold包含了迄今为止AlphaFold发布的所有神经网络(NN)模型,具有多个可增加结构多样性的参数,并且可以设置只保留最有希望的预测结果。该程序可以并行运行多个实例,每个GPU最少可运行一个预测,从而优化利用现有计算基础设施,将获取预测结果所需的时间从几个月大幅缩短到几小时。MassiveFold通过conda环境易于安装,使用简单的命令行和JavaScript对象表示法(JSON)参数文件即可运行。
模型部分
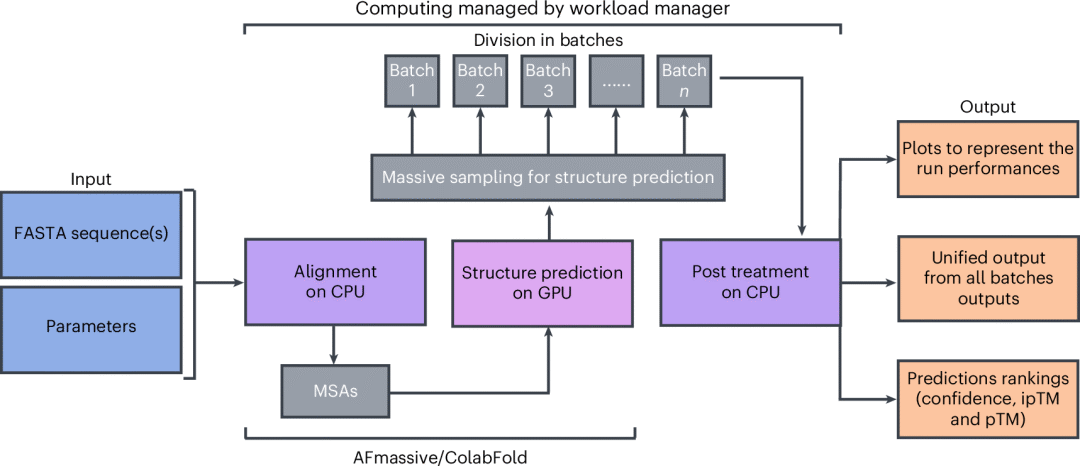
图 1
图1展示了MassiveFold的计算流程。为了充分利用多样性参数,MassiveFold集成了优化的并行化系统,包括三个部分:(1)在中央处理器(CPU)上计算序列比对;(2)在GPU上将结构推理分成多个批次;(3)在CPU上进行最终后处理,收集结果、对所有预测进行排序并生成图表。MassiveFold通过后处理整合所有预测结果并生成多个图表。这些图表包括采用AlphaFold和/或ColabFold配色方案的预测局部距离差异检验(pLDDT)和预测对齐误差(PAE)图,以及ColabFold对齐深度图。即使没有选择ColabFold作为推理引擎,也会生成这些图表。
在第1步中,当使用AFmassive时采用JackHMMer和HHblits进行比对,使用ColabFold时则采用MMseqs2。在第2步中,结构推理由AFmassive或ColabFold执行。在第3步中,如果使用ColabFold,输出会转换为AlphaFold的输出格式:结构文件名前加上其排名索引,创建ranking_debug.json文件并重新格式化pickle文件名。AFmassive原生使用这种格式。在这两种情况下,都提供了"轻量级"pickle选项,该选项在保留主要信息的同时大大减小了pickle文件的大小。第2步和第3步仅在前一步完成后才开始。值得注意的是,将预先计算的比对结果放在输出文件夹中即可使用。除非强制重新计算,否则系统会检测到这些结果并且不会再次计算。
MassiveFold的设计目的是为了便于访问多样性参数并实现计算的最优管理。它在推理步骤中充分利用GPU集群,同时对不需要GPU的多序列比对和后处理步骤使用CPU。MassiveFold也针对单GPU机器进行了优化,因为大规模采样任务可以在低优先级下运行,从而允许更高优先级的任务插入计算队列中。
MassiveFold为多聚体靶标H1140生成的相关图表
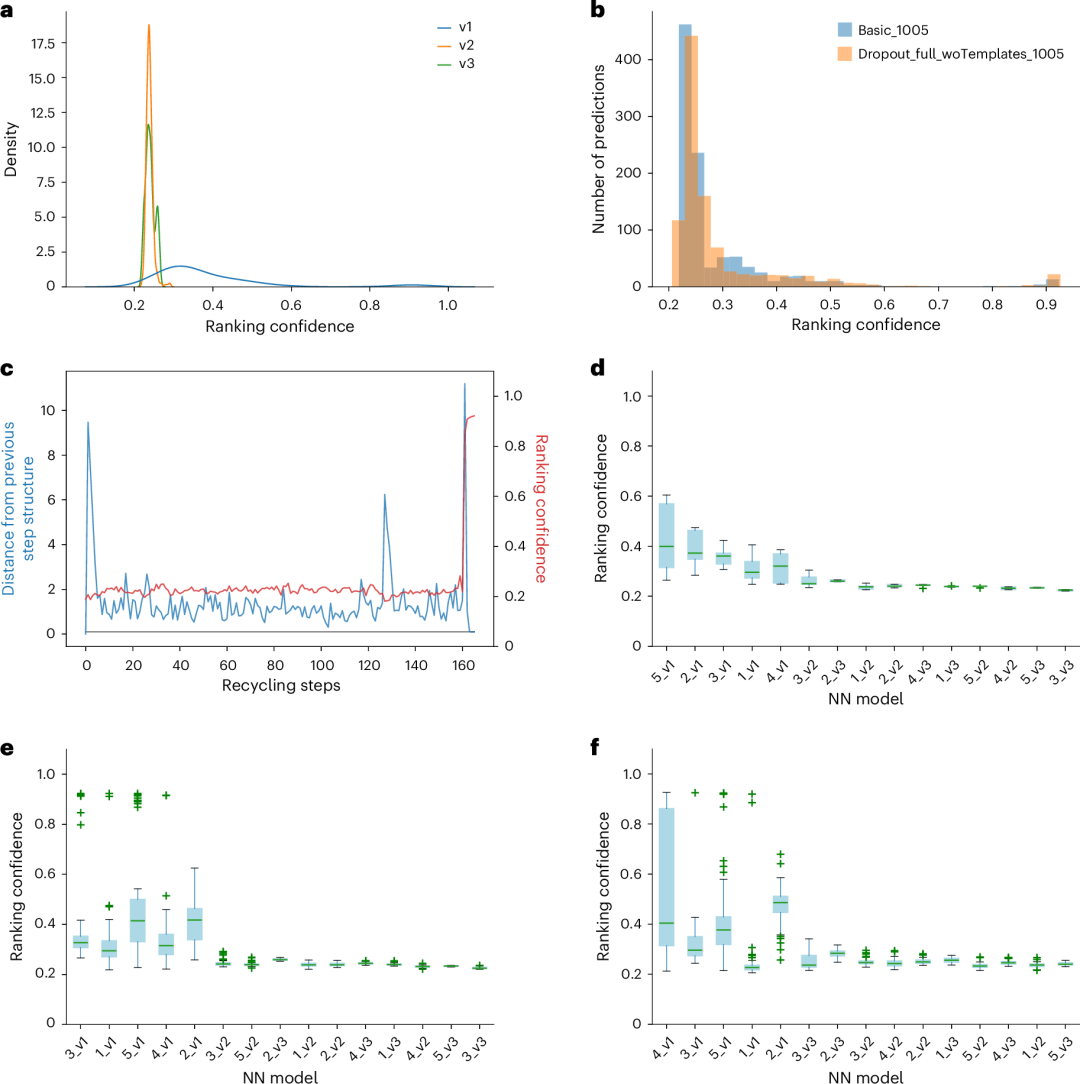
图 2
此外,MassiveFold还绘制了不同维度的置信度分数分布图,包括按AlphaFold神经网络版本(图2a)、按单个神经网络模型(图2d-f)或所有模型汇总的分布图。由于MassiveFold可以使用不同的参数组合运行,因此也可以生成比较不同参数组合下置信度分数分布的图表(图2b)。最后一个图显示了AlphaFold置信度分数在循环过程中的演变,以及连续结构之间的距离,这可以与提前停止容差参数进行比较(图2c)。
分数分布图和循环图展示了预测行为,突出显示了AlphaFold置信度分数随神经网络模型变化的多样性。图2d展示了MassiveFold对CASP15目标H1140进行默认运行时预测结果的多样性。在这里,生成了75个结构(每个神经网络模型5个),最高分数不超过0.6。当计算扩展到1,005个结构(每个神经网络模型67个,其他参数不变)时,已经产生了一些置信度分数超过0.8的异常值(图2e)。通过激活dropout并排除模板,这种分布可以得到进一步改善(图2f),证明了大规模采样策略的附加价值。这些图还表明,在这种情况下,只有v1神经网络模型产生了高置信度结构,如果只对前五个神经网络模型(均为v1)进行扩展采样,就可以减少计算时间。
通过循环参数实现大规模采样是另一种方法,这些参数在生成多样性方面发挥着重要作用。图2c展示了使用AFmassive对CASP目标H1140进行结构预测时的循环行为,该预测启用了dropout并且不使用模板,设置提前停止容差为0.1,最大循环步数为1,000。图中显示在前160个循环步骤中置信度分数较低,随后突然跃升至0.846和0.908。当提前停止容差为0.5时,十个最佳预测中只有四个显示这种跃升,而当提前停止容差为0.1时,所有十个预测都出现了这种跃升。因此,需要将广泛的循环作为大规模采样的一种可行的补充方法。MassiveFold的计算任务分割功能使这种探索变得容易实现。
尽管MassiveFold可以使用AFmassive或ColabFold作为推理引擎,但只有在采用大规模采样策略并激活多样性参数时,才能生成具有高置信度分数的异常值(图2)。
MassiveFold的多样化参数
MassiveFold包含的多样性参数包括以下内容:AlphaFold目前发布的所有神经网络模型(包括之前的版本,即5个单体模型和15个多聚体模型)、EvoFormer模块和结构模块中的dropout激活、模板的使用、循环步数以及早停的容忍阈值(当当前结构与前一个结构之间的距离低于此阈值时停止循环)。此外,MassiveFold还接受一个额外的JSON文件作为输入,用于指定单独的dropout率,从而为用户提供增加结构多样性的额外选项。
编译|黄海涛
审稿|王梓旭
参考资料
Raouraoua, N., Mirabello, C., Véry, T., Blanchet, C., Wallner, B., Lensink, M. F., & Brysbaert, G. (2024). MassiveFold: unveiling AlphaFold’s hidden potential with optimized and parallelized massive sampling. Nature Computational Science, 1-5.
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢