DRUGAI
HelixFold3 重磅升级
HelixFold3是百度螺旋桨团队基于飞桨深度学习框架和百度智能云高性能计算平台,自主研发的面向生物计算领域的大模型工作。自 24年8月首次发布以来,HelixFold3 凭借其卓越的技术架构和系统能力,在常规生物分子结构预测任务中达到 与 AlphaFold3 精度媲美的水准,获得业界广泛关注。
近期,螺旋桨PaddleHelix团队又一次重磅升级HelixFold3工作,在RNA结构预测、共价修饰、抗原抗体等复杂场景上展现出更高的准确性。
HelixFold3在RNA分子和共价修饰场景中的精度优势明显。在CASP 15 RNA数据集【图1】上的评测结果显示,HelixFold3的预测精度显著超越了AlphaFold3,同时在从PDB数据库收集的最新蛋白-RNA复合物结构预测任务中【图2】,也展现出更高的准确性。此外,在多个涉及共价修饰的结构预测任务中,HelixFold3的表现持平或高于AlphaFold3,特别是在RNA修饰相关的预测中精度更为突出【图3】。这些结果表明HelixFold3能为RNA靶点药物设计和蛋白-RNA相互作用研究提供了可靠的结构依据,助力开发针对RNA及修饰相关疾病的新型疗法。
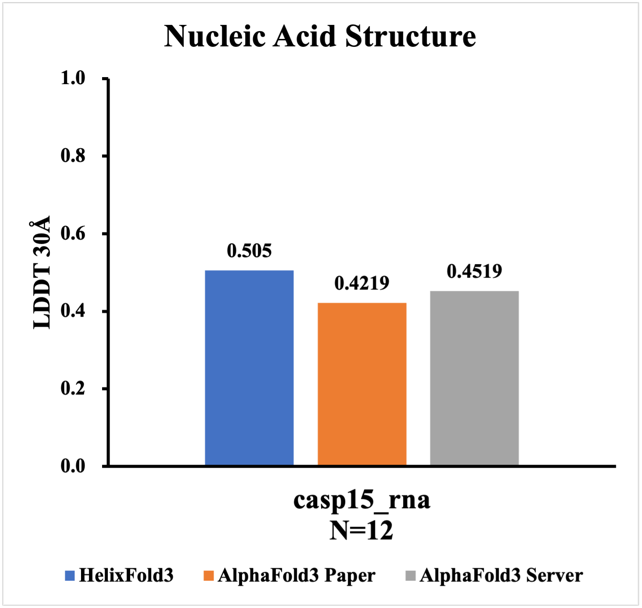
图1
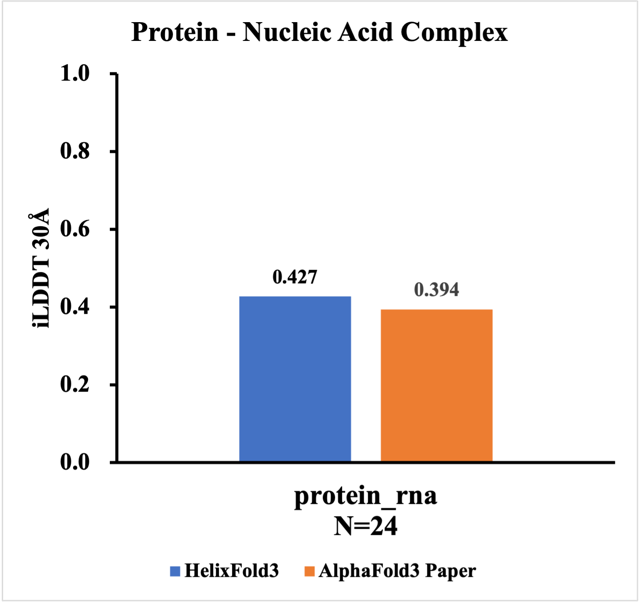
图 2
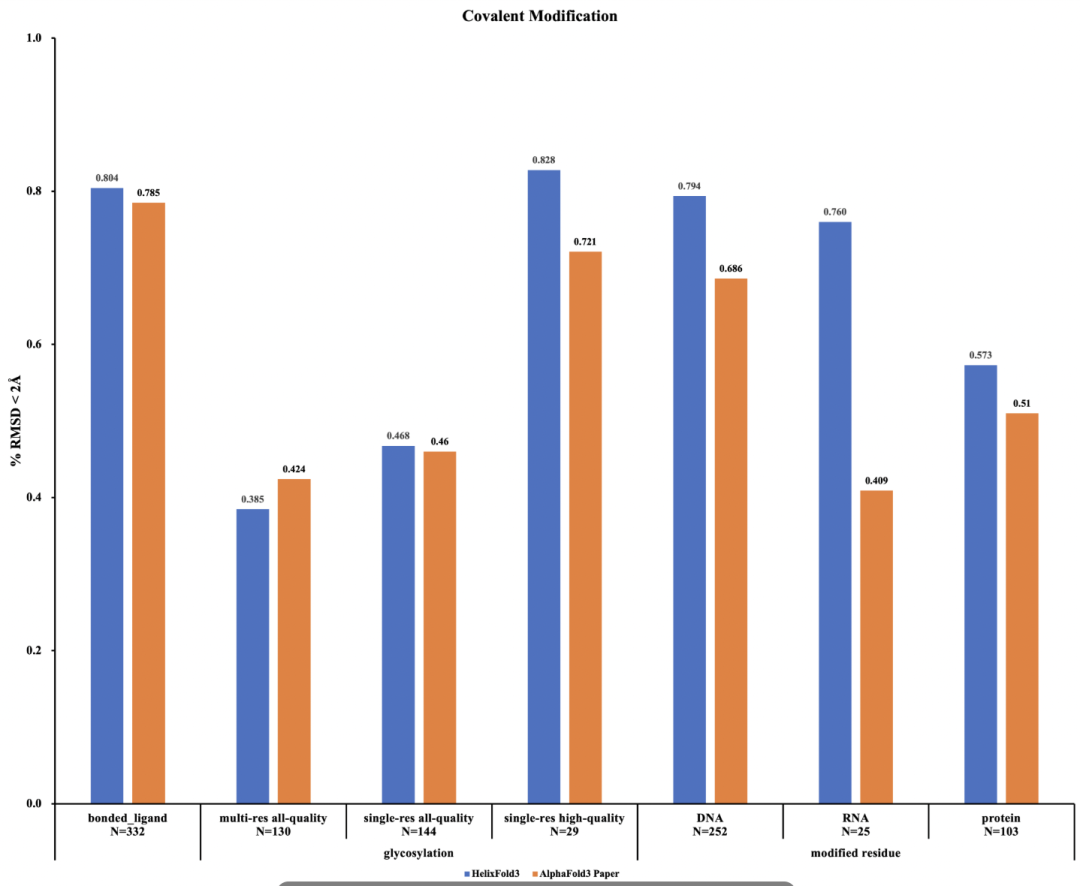
图 3
抗原-抗体结构预测一直是蛋白质复合体结构预测中的一大挑战。HelixFold3在最新的71例PDB抗原抗体结构预测的数据上精度已能媲美AF3 server【图4】,通过指定任意数量的抗原表位氨基酸,HelixFold3在抗原抗体结构预测的精度进一步得到提升,在任意指定15个表位氨基酸的数量后,HelixFold3的成功率进一步提升至80%以上【图5】,表明其为研究抗体的结合机制分析提供解决方案的潜力。
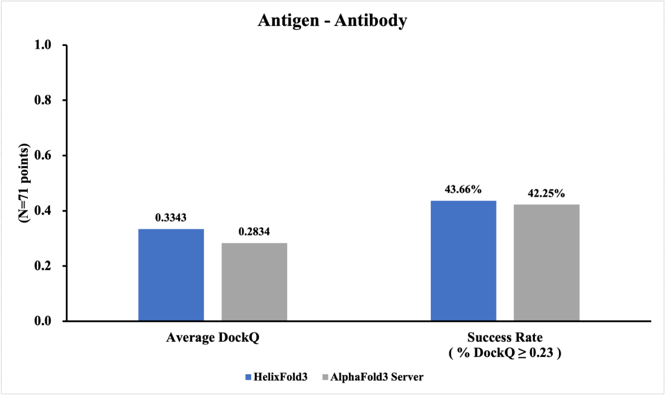
图 4
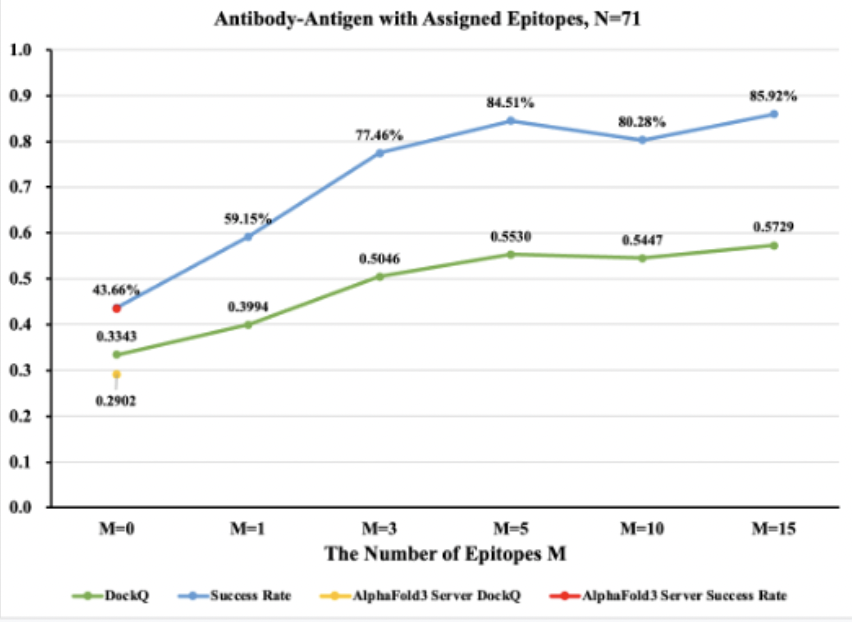
图 5
HelixFold 3在线服务功能更加丰富实用
在功能层面,HelixFold3 在线服务能同时支持小分子以CCD和SMILES格式的输入,大幅拓展了可支持的配体范围,提升在小分子药物发现场景的应用性,可以更加灵活的探索如PROTAC等的新场景,显著提升早期药物开发的效率。
HelixFold3在线服务还支持蛋白质、DNA 和 RNA 的任意侧链修饰,极大地提升了指定修饰的灵活度。用户可在服务平台【图7】轻松通过通过 SMILES 格式指定修饰基团【图6绿色框】和连接位点【图6橙色圈】。
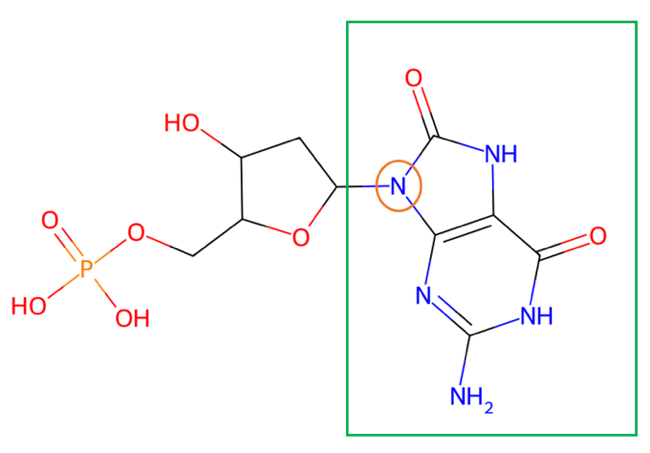
图 6
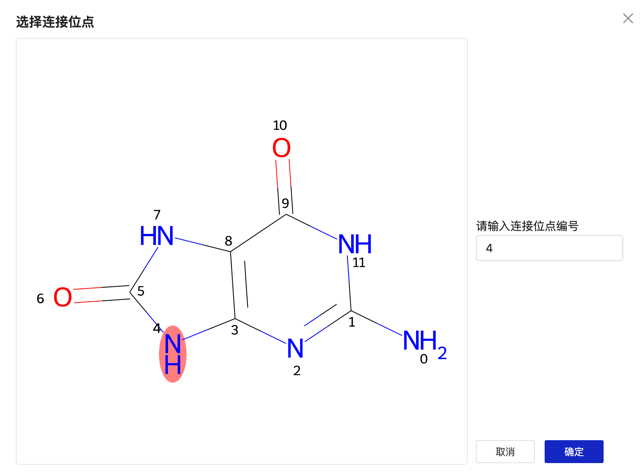
图 7
相比 AlphaFold3 仅支持 46 种预定义修饰,HelixFold3通过SMILES去定义侧链修饰所能覆盖的化学空间更广,覆盖PDB 数据库中超过 50% 的修饰场景(AlphaFold3 仅支持不到 5%),通过指定连接位点的可视化操作,满足更多样化的需求,为科研创新提供更全面的技术支持。
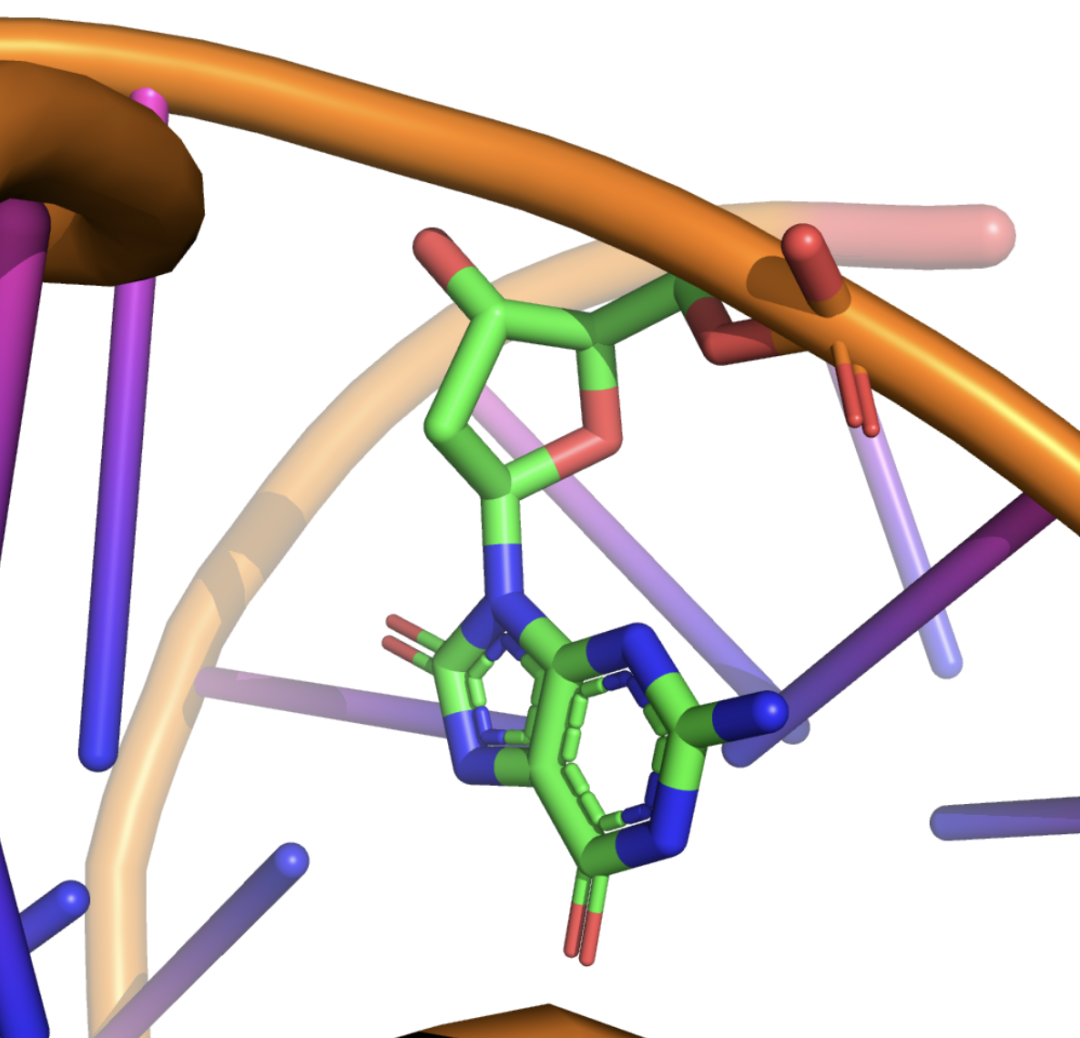
图 8
图8案例说明:图为8-氧代-2'-脱氧鸟苷(8-oxo-dG)修饰是一种关键的氧化损伤标志物,其在 DNA 分子中的存在直接反映了细胞的氧化应激水平【图7】。由于其与 DNA 的结构和功能改变密切相关,其修饰已被广泛研究,成为癌症、神经退行性疾病和衰老等病理过程的潜在早期诊断和预测指标。通过支持多种 DNA 侧链修饰的预测,HelixFold3 提供了一个强大的功能,不仅能够帮助揭示氧化应激和 DNA 修饰对细胞生物学的深远影响,还为早期诊断和精准医疗奠定了坚实基础。
HelixFold3 依托百度智能云CHPC平台强大的计算资源和高效的资源调度能力,通过MSA 检索与模型推理方面的性能优化,使得推理成本大大降低,可在数小时内完成数千次精确的生物分子结构预测,极大提升了高通量生物信息学应用的处理能力。
为满足生命科学领域研发人员的需求,HelixFold3 也提供了简洁易用的 API 服务(API连接:https://paddlehelix.baidu.com/app/tut/guide/all/helixfold3sdk)。用户通过简单的操作即可轻松调度百度智能云资源,快速完成数万次结构推理,免去昂贵硬件和复杂配置的限制。API支持与现有生物信息学工具链的无缝集成,广泛适用于科研创新和商业应用的各个领域。
HelixFold系列探索更多应用场景
小蛋白/多肽发现场景
小蛋白/多肽结合剂具有特异性强、亲和力高、稳定性好等特点,广泛应用于癌症药物研发、AAV疫苗、基因编辑、CAR-T治疗等领域,对精准医疗和药物发现场景具有重大价值。它能够特异性靶向关键蛋白质,有望带来更有效、更精准的疾病解决方案,为疾病治疗和生物科学研究提供强大的保障。
当前小蛋白设计领域的研究表明 [文献A],结构预测模型的打分,能够有效地筛选甄别出与靶蛋白有潜在相互作用的结合小蛋白。
干实验初见成效
在Cao Data 的数据 [文献B3],选取靶点IL7Ra和TrkA,各自包含15,000个有实验亲和力的小蛋白序列样本,其中有结合(亲和力kd数值低于4 μM)的小蛋白分别有20和10条序列。基于此构建的数据集用于测试 HelixFold3 在小蛋白结合剂筛选上的能力,评估多个模型的打分排名前百分之一的分子中有结合的小蛋白比例(成功率)。结果显示【图9】,HelixFold3 的成功率优于其他结构预测模型如AlphaFold2,以及传统物理的亲和力计算模型如Rosetta ddG等。
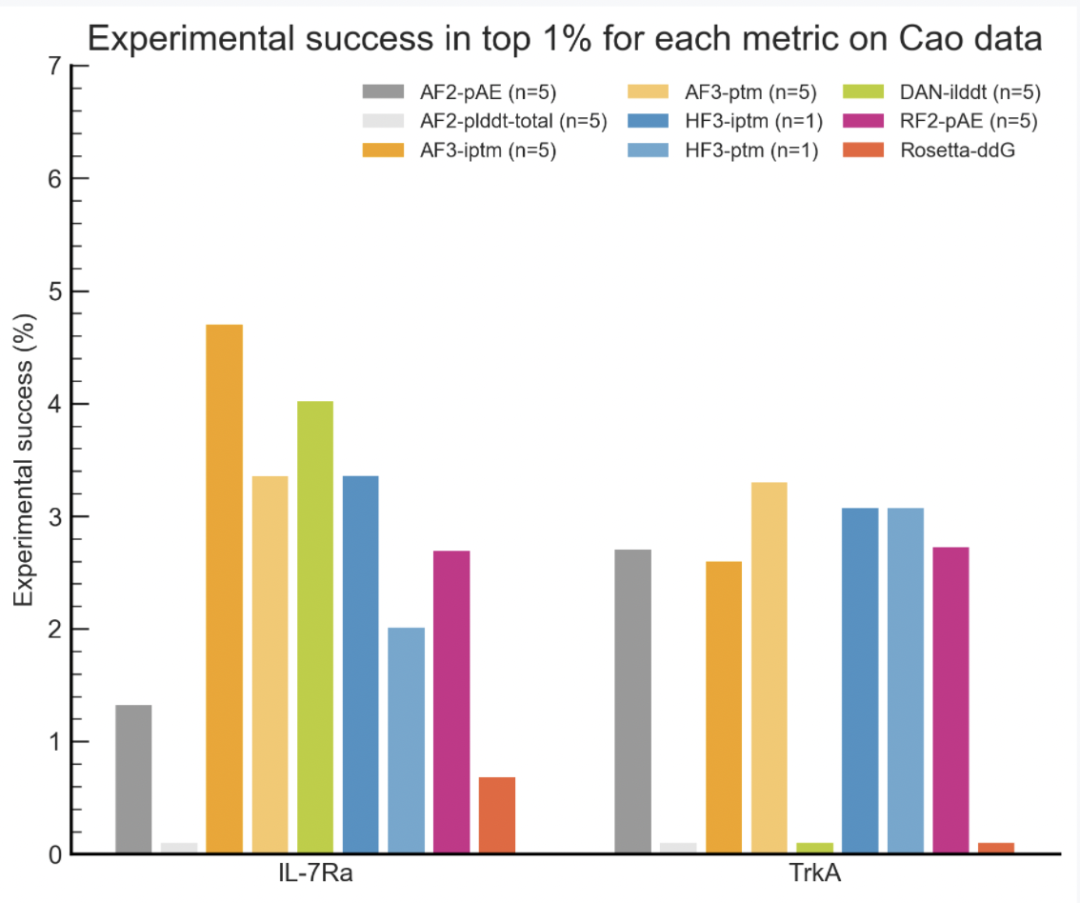
图 9
相比起反映整体结构的置信度的打分pTM,反映相互作用界面的置信度打分iPTM的指引性更强,iPTM 指标在一定程度上能够有效辨别出小蛋白与靶蛋白有无结合,对研发人员进行小蛋白筛选任务更具指导意义。
通过使用HelixFold3的API服务,可以便捷的大规模预测出小蛋白/多肽和靶点结合的强弱,以达到通过结构预测指标筛选出高潜力分子的目标。通过大通量推理服务,用户可以使用 HelixFold3 服务在一天时间内完成上述IL7Ra靶点15000个候选小蛋白的结构推理。大大提升了实验成功率。
湿实验证明落地的可行性
为了验证技术在真实药物研发场景的可用性,百度螺旋桨 PaddleHelix 团队与一家领先的多肽CRO研发企业合作,基于 HelixFold模型共同开发了多肽设计一体化技术方案。多个靶点的湿实验结果表明,该方案具有较高的成功率(结合效力小于4μM的分子占进入湿实验的分子的比例【图10】)。特别在 GPCR 类靶点上,通过算法针对GPCR 蛋白激活/抑制不同状态进行设计筛选,在多个 GPCR 靶点成功设计出多个 EC50 低于100 nM 的多肽分子,甚至在2个靶点上设计出 EC50 在皮摩尔级别的高效分子【图11】。这些成果充分验证了 HelixFold技术在小蛋白/多肽药物场景的巨大应用潜力。
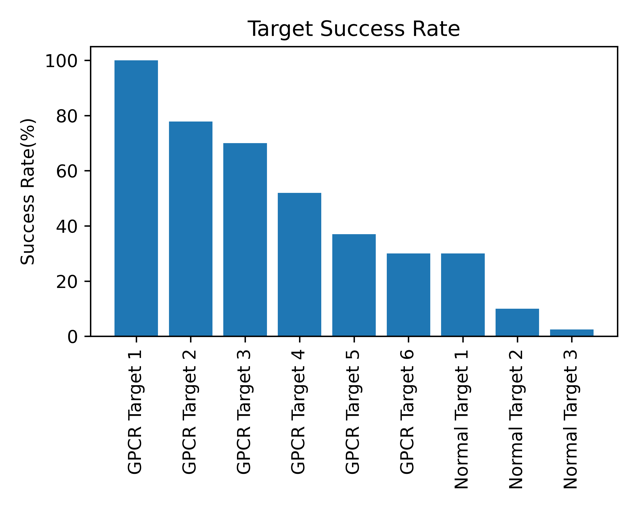
图 10
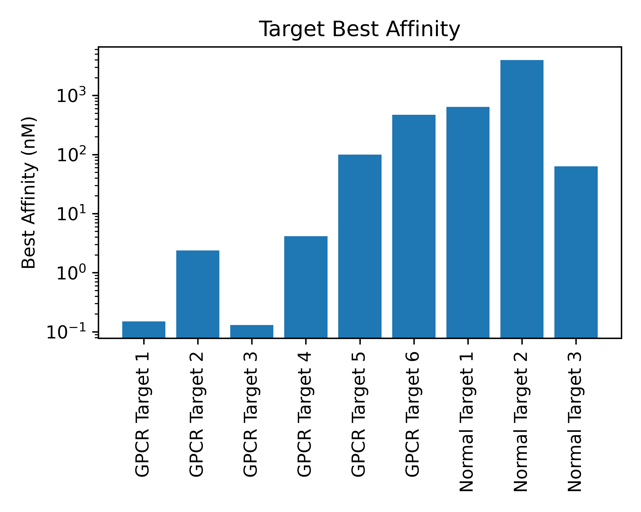
图 11
抗体发现场景
抗体药物因其靶点特异性强、稳定性高、毒性低等优势,已成为生物治疗领域的重要方向。尽管基于深度学习的蛋白质结构预测模型在常规蛋白-蛋白复合体结构预测中已取得重大突破,但在抗原-抗体复合体的结构预测上其精确仍面临诸多挑战。这种局限性不仅影响抗体结合物识别和亲和力预测等相互作用分析的准确性,也限制了抗体药物的开发。为解决这些问题,PaddleHelix 团队研发针对抗原-抗体复合体结构预测的模型,在构象预测和相互作用预测方面取得显著进展。在抗原-抗体构象预测任务中,在 RCSB 数据库最新发布的 141 例抗原-抗体复合体测试中,成功率达到 58%,显著优于目前性能最佳的模型 AlphaFold3(36%)【图12】。在抗原-抗体相互作用预测上,置信度打分iPTM在四个靶点抗原(SARS、LYSO、VEGF 和 PD-1)上成功识别出结合位点的高亲和力抗体。此外,在多个流感毒株的亲和力数据中,模型预测结果与实验数据呈现更高的正相关性。与现有的蛋白质结构预测模型 AlphaFold-Multimer 和基于蛋白质语言模型的 ESM2 相比,HelixFold-Multimer 在预测精度上实现了显著提升【图13】。
这些进展充分证明了更高的抗原抗体结构预测模型,对在抗体药物开发起到重要的推动作用,不仅助力加速抗体药物的筛选和优化,还为生物治疗领域的技术创新提供了强有力的支持。目前,HelixFold-Multimer 的能力已全面整合至最新的 HelixFold3 模型中,用户可通过 HelixFold3 的 Web 服务和 API 接口便捷体验其功能。
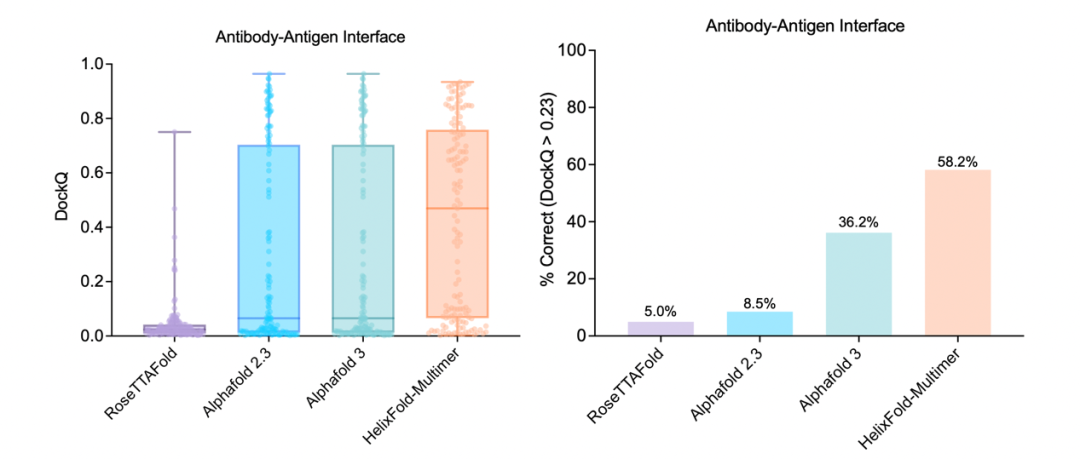
图 12
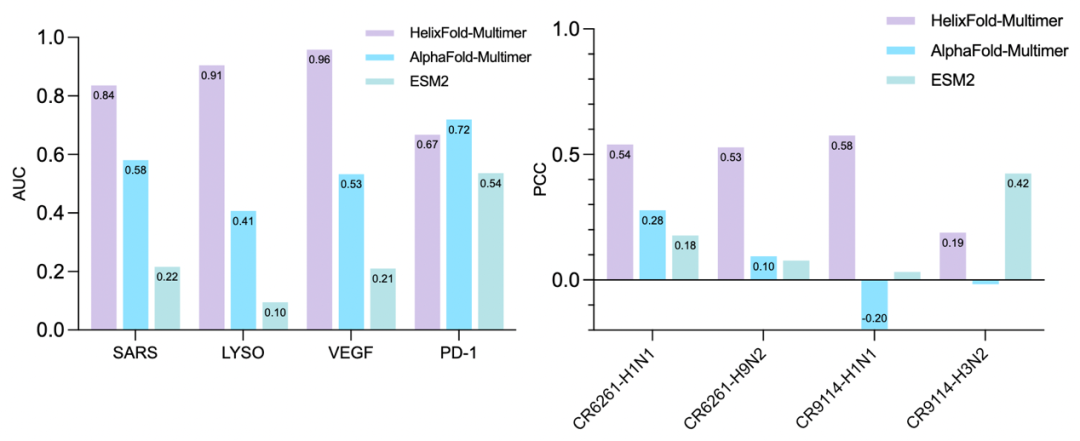
图 13
酶改造场景
自然界提供了丰富的蛋白质资源,其中大多数蛋白作为生物催化剂酶存在在自然界中。酶蛋白能高效地催化特定化学反应,加速生产过程,广泛应用于绿色化工、污染治理、能源转化、农业育种等领域。酶蛋白领域的研究表明 [文献A] ,结构预测模型可辅助筛选甄别出有特定功能和活性的酶。为了验证HelixFold3在酶改造的应用潜力,PaddleHelix团队基于COMPSS数据集 [文献B1/B2/B3]验证测试HelixFold3在酶蛋白筛选上的潜力。该数据集包含大量与 MDH 和 CuSOD 酶相关的蛋白序列及其实验测定的酶活性数据。在测试中,HelixFold3 通过其置信度评分(pLDDT)对候选酶蛋白进行打分排序,其预测精度(AUC-ROC)显著优于现有的结构预测模型,包括 AlphaFold2 和 Chai-1。此外,与传统分析方法(如溶剂可接触表面积 SASA、带电性 Charge、一致性 Identity 等)相比,HelixFold3 也展现了更强的筛选能力,在准确性上也明显超越了蛋白语言模型 ESM-1v【图14】。
这一结果充分证明了 HelixFold3 在酶蛋白筛选和设计中的强大潜力,不仅为酶改造提供了高效的解决方案,还为进一步推动绿色技术和生物工业创新奠定了坚实基础。
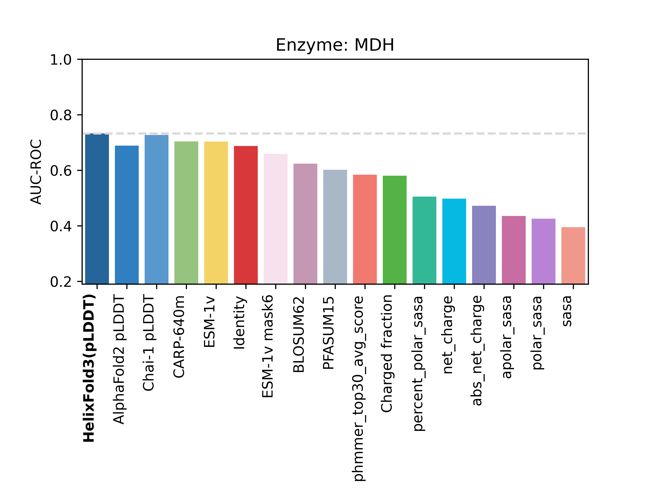
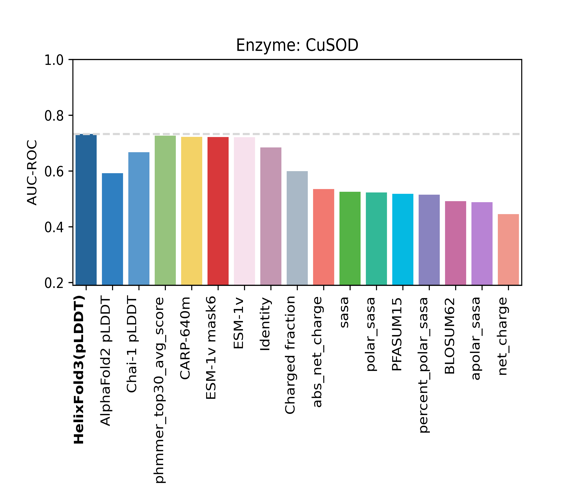
图 14
One more thing
HelixFold3 线上服务已经更新到v3.1版本,在模型精度、功能支持、应用场景适配上均做了升级优化。感兴趣的小伙伴们可以通过在线服务进行访问。
HelixFold3 Server:
https://paddlehelix.baidu.com/app/all/helixfold3/forecast
PaddleHelix方案相关文献:
HelixFold3:https://arxiv.org/abs/2408.16975
GPCR-peptide:https://www.biorxiv.org/content/10.1101/2024.11.27.625792v1
HelixFold-Multimer:https://arxiv.org/pdf/2404.10260
AIStudio应用场景教程:
小蛋白设计:PaddleHelix工具集应用:蛋白结合剂的筛选 - 飞桨AI Studio星河社区
酶设计:PaddleHelix工具集应用:酶蛋白的筛选 - 飞桨AI Studio星河社区
百度智能云CHPC架构介绍

高性能基础设施层。高性能基础设施层提供了持续更新的IaaS高性能计算、存储和网络。
在计算方面,提供小规格计算能力,带来更好性价比。同时,提供基于百度太行架构的弹性裸金属服务器,在大算力作业、并发作业上能获得更好的性能。针对复杂的3D前后处理场景以及一些GPU加速的工业仿真场景,我们还提供了各种类型的GPU实例可以满足客户要求。
在网络方面,在配备公网、VPC等基础网络之外,还提供了大规模的RDMA组网能力,可提供低延时、高带宽通信能力,满足高性能计算对网络的严苛要求。
在存储方面,在配备文件存储、对象存储之外,还为CHPC配备了高性能并行文件系统 PFS,满足高性能计算要求。同时创新性和百度网盘进行对接,满足部分行业客户的海量数据分发。
HPC调度以及任务管理能力。
HPC调度方面,传统调度器集成丰富,例如SLURM,OpenPBS,SGE等等,同时也支持商业版的调度器,可以为客户提供更多可选择性。另一方面我们也有自研的类似Serverless调度能力,可以完全忽略laas层和CHPC管理层,只要提交作业即可直接运行,节省了IT基础设施的运维和管控层维护需求。
生物领域应用。
HelixFold3 全原子相互作用大模型,实现小分子、蛋白质、DNA、RNA的相互作用。
灵活的API调用服务,按需使用,云端弹性调用。
可视化前端展示界面,无代码操作平台。
参考资料
文献A,COMPSS,Johnson, Sean R., et al. "Computational scoring and experimental evaluation of enzymes generated by neural networks." Nature biotechnology (2024): 1-10.
文献B1,AlphaProteo,Zambaldi, Vinicius, et al. "De novo design of high-affinity protein binders with AlphaProteo." arXiv preprint arXiv:2409.08022 (2024).
文献B2,BindCraft,Pacesa, Martin, et al. "BindCraft: one-shot design of functional protein binders." bioRxiv (2024): 2024-09.
文献B3,cao data,Bennett, Nathaniel R., et al. "Improving de novo protein binder design with deep learning." Nature Communications 14.1 (2023): 2625.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢