
点击蓝字 关注我们

微末生物
Nanomega CryoAI
端粒酶是一种独特的内源性真核生物逆转录酶(RT),维持线性染色体末端,是细胞衰老、干细胞更新和肿瘤发生的高度调控的决定因素。
戳此了解此前发表的关于端粒酶的文章内容:染色体小尾巴的秘密守护者
端粒酶RNA(TER)包含一个与端粒重复序列互补的区域,该区域被用作向染色体3 '端添加连续重复序列的模(例如人类重复序列TTAGGG的添加)。端粒酶催化的端粒重复序列合成的专门循环已被广泛研究了人类酶和来自模式生物的生化类似酶(如端粒丰富的纤毛虫嗜热四膜虫)。
虽然体外端粒酶的催化活性只需要TERT和TER,但生理功能全酶是一个多亚基RNP复合体。通过亲和纯化、质谱分析和亚基标记实验,已鉴定出四膜虫端粒酶全酶的8种特异性亚基,每种均对端粒长度维持至关重要。其中三个亚基是催化活性RNP在体内组装所必需的:TERT, TER和La家族蛋白p657。
p65 La和RRM1结构域增加了p65对TER的亲和力,但只有C端xRRM2结构域(图1a)对TER折叠至关重要,从而在体外和体内增强TERT RNP组装。两个TER区域,p65 xRRM2相互作用位点旁边的环4 (L4)和TERT模板上的高亲和结合元件(TBE) 5 '(图1a),与TERT高亲和RNA结合域(TRBD)结合。TERT TRBD、RT结构域和CTE延伸(CTE)形成一个环,环绕活性位点空腔,第四个TERT N -末端(TEN)结构域(图1a)位TER依赖但未知的位置。
对于体内端粒延伸所需的另外5种四膜动物端粒酶全酶蛋白(p75, p50, p45, p19和Teb1)的结构和作用知之甚少;只有Teb1具有已知的结构域结构。与单链DNA (ssDNA)结合因子复制蛋白A的同源大亚基一样,Teb1具有四个OB-fold结构域NABC(图1a);A和B以高亲和力结合ssDNA,而C是全酶结合所必需的。Teb1对天然组装的四膜虫端粒酶全酶与DNA产物的高稳定性相互作用至关重要,表现为体外的高重复添加持续性(RAP)。全酶亚基p75、p19和p45,在这里被指定为7-1-4,形成一个亚复合物,在微球菌核酸酶诱导的其他全酶亚基体外解离中保持组装。目前对p50知之甚少,它在亲和纯化全酶的银染色凝胶上表现为亚化学计量。
为了探究端粒酶全酶的物理和功能网络结构,美国加州大学分子与细胞生物系Juli Feigon和Z. Hong Zhou教授团队通过冷冻电镜探究了四膜虫端粒酶全酶的天然结构并进行了体外完全重构。
整体结构与亚基定位
通过亲和纯化制备四膜虫端粒酶全酶用于电镜分析。实验使用10种不同菌株,这些菌株的TERT或其他全酶亚基携带N端或C端3×FLAG与串联Protein A标签(分别缩写为zzf或fzz)。通过比较TERT-fzz纯化端粒酶的负染色电镜(EM)图像类平均与冷冻电镜(cryoEM)图像(图1b),发现负染色未改变结构的实验分辨率。除主要构象(“稳定构象”,占总颗粒的31%)外,类平均图像显示存在其他构象和亚基组成的颗粒亚群。负染色与冷冻电镜图像均显示颗粒在碳膜上呈高度一致的取向偏好。
为克服取向偏好与结构异质性,采用自动化随机锥形倾斜(RCT)方法进行电镜图像采集与三维重构。TERT-f与Teb1-f全酶的三维结构无差异,且Teb1-f全酶产率更高。基于2,220个颗粒的三维重构,获得了约500 kDa的Teb1-f全酶结构(图1c),分辨率为~25 Å,显示全酶密度分布有序,占据约200×150×80 Å的空间。为定位全酶结构中的各蛋白亚基,从携带fzz标签的TERT、p75、p65、p45、p19或Teb1菌株中纯化端粒酶。纯化过程中,蛋白酶切去除标签的zz部分,仅保留短f标签(3×FLAG),并用单克隆抗FLAG Fab进行标记。每个标记蛋白最多可结合三个Fab分子,类平均图像与三维密度图显示Fab结合位点明确(图1c)。除p45通过排除法确定外,其余全酶蛋白亚基均通过Fab标记定位。此外,通过MS2衣壳蛋白(MS2cp)识别TER茎2(S2)的小发夹标签(MS2hp),纯化功能完整的端粒酶(图1c)。MS2cp二聚体在类平均图像中表现为额外密度,从而确定了模板上游TBE的大致位置(图1a)。通过Fab标记的端粒酶颗粒三维重构,精确定位了TERT与Teb1的C端以及p50的N端。p65、p75和p19的C端因纯化产率低,仅通过类平均图像获得大致定位。结合已知高分辨率结构(TERT、TER、p65、Teb1)与不同复合物的类平均及三维重构对比,构建了亚基边界近似模型(图1d)。
数据显示:在“正面视图”(图1c,d)中,p65位于底部,TERT占据颗粒中心,7-1-4亚基构成顶部,Teb1从中层向右延伸,p50作为中层枢纽连接TERT、7-1-4和Teb1,亚基间的密集接触形成了复杂且高度轮廓化的表面。
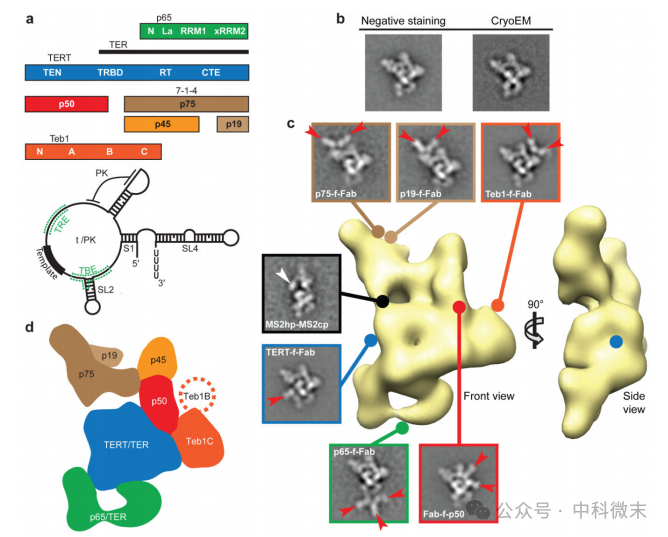
图1 四膜虫端粒酶全酶的电镜重建及亚基定位
RNP催化核的结构
基于确定的亚基位置,该研究在Teb1-f端粒酶的EM密度图中拟合TER和蛋白质结构域的晶体和核磁共振结构,以生成整个RNP催化核心的模型(图2a)。
四膜虫TERT的RT和CTE结构域是根据Tribolium castaneum TERT晶体结构同源建模的,并结合了四膜虫部分TRBD15和TEN16结构域的晶体结构。模型TERT TRBD-RT-CTE只在一个方向上拟合(图2a、b)。TERT CTE的位置与TERT- ffab端粒酶三维重建中鉴定的C端位置一致(图1c)。模拟的四膜虫TERT TRBD与CTE相互作用,这与Tribolium TERT晶体结构一致,并分离出人类TERT结构域相互作用。存在连接TEN和CTE区域以及TEN和TRBD区域的EM密度,这可能对应于潜在非结构化TEN-TRBD连接物和高分辨率结构中缺失的相邻TRBD序列的~ 70个氨基酸,它与ssDNA交联并改善模板边界定义。
p65的C端位于粒子底部,位于TERT下方(图1d)。在类平均和3D重建中,我们观察到颗粒亚群在颗粒的左下方缺乏部分密度(图2c),这是由全酶纯化过程中常见的p65亚基部分蛋白水解导致p65 N 端结构域的丢失和/或剩余的La和RRM1结构域的位置约束丢失造成的。因此,p65 C末端结构域xRRM2:stem 4 (S4)络合物的结构可以定位并适合所有3D重建中的密度(图2a, d)。p65N端结构域(S1附近)和xRRM2中被晶体结构删除的长β2-β3环可以占据剩余的未建模密度(图2a)。这些接头一起提供了p65- TER相互作用的整体拓扑结构(图2a, d)。
四膜虫TER包含两个主要结构域,模板/伪结(t/PK)和SL4,它们通过S1连接(图1a)。从TERT18活性位点、MS2cp标记的S2和p65 ×RRM28配合物中SL4模板的确定位置开始(图2d),并考虑区域长度、模型结构和拓扑限制,研究者追踪了整个TER的潜在轨迹(图2a,d,e)与剩余EM密度的拟合。模板识别元件(TRE)似乎靠近TEN结构域,而PK的底部靠近L4(图2a,d)。模型中TER元素的位置与TER的大量生化数据一致。特别重要的是两个TRBD结合元件TBE和L4的位置确定,其分别与TRBD的两个不同区域结合,分别靠近TERT环的左上角和底部(图2b、d、e)。值得注意的是,只有SL4的顶端环与分配给TERT的密度相连,导致TER S1/SL4与p65结合形成的全酶底部呈u形。(图2a, d, e)。
远端SL4与TERT的相互作用解释了它如何在trans35,36中加入t/PK时刺激端粒酶活性,以及p65 ×RRM2与S4的结合如何介导p65- ter与TERT8-10的分层组装。EM结构和模型显示,TRBD桥接TBE和L4,它们相距约40 Å。在模型中,远端SL4接近PK的3 '端,与报道的L4和内部PK位置之间的高FRET一致。尽管详细的相互作用有待于高分辨率结构,但在该模型中,TBE与t -口袋和CP-motif区域(T-CP口袋)的TRBD接触(图2f),并且远端SL4被CTE和TRBD侧翼(图2g),可能有助于TERT在TER周围折叠。
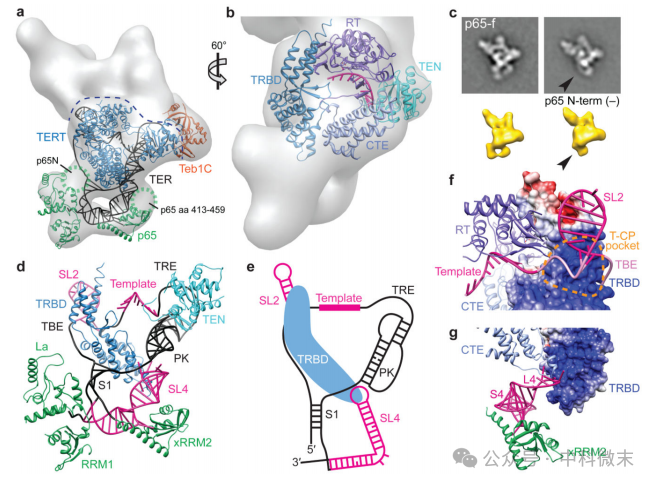
图2 RNP催化核的结构
P50锚定辅助蛋白
使用zzf-p50纯化的端粒酶的催化活性具有较低的RAP,而使用p50-fzz纯化的全酶则具有较高的RAP活性(图3a)。
Fab标记显示p50的N端和Teb1的C端相对靠近(图1c)。比较f-p50和Teb1-f端粒酶的类平均可以发现,所有的f-p50端粒酶都缺乏Teb1的密度,并通过差异图揭示了Teb1的边界(图3b)。通过比较仅含有RNP催化核心的小亚类颗粒的类平均值进一步定义p50的边界,该亚类仅在ms2cp结合的MS2hp端粒酶中观察到,类平均值含有p50(图3c)。Fab-f-p50和Teb1-f端粒酶在缺乏Teb1的TERT-f端粒酶上的3D重建叠加表明,与f-p50 n端结合的Fab与Teb1占据相同的空间(图3D)。
总之,这些结果确定了Teb1和p50的位置,表明p50直接与RNP催化核心相互作用,并揭示Teb1靠近p50的N端和TERT。因此,全酶结构解释了纯化的N端标记p50的低RAP活性(图3a),因为N端标记zzf-p50的酶纯化破坏了Teb1的结合。除了分配给RNP催化核心,Teb1和p50的密度外,顶部的剩余密度由7-1-4亚复合物占据。
综上所述,三维重建揭示了p50在全酶结构中的中心位置,即作为TERT, 7-1-4和Teb1之间的相互作用枢纽(如图3f所示)。
功能分配给子单元架构
与结构分析并行,研究者开发了一种完全重组亚基的全酶重组方法。将含有TERT-f的RNP催化核心预先组装在兔网织细胞裂解液(RRL)中,然后与RRL表达的p50和/或7-1-4和/或细菌表达的Teb1BC结合(图3g)。
直接引物延伸实验表明,单独添加7-1-4或Teb1不会改变RNP催化核心的低rap产物合成曲线,但单独添加p50刺激酶活性和多重复产物的合成(图3g)。对于含有p50的酶,单独添加7-1-4增加了产物的数量(图3g),单独添加Teb1增加了产物的长度。并且它们组合成完整的全酶对提高活性水平和长产物合成具有协同作用。在5分钟的反应时间内,所有的产物DNA都可以通过凝胶电泳(图3g)或额外的时间和[dGTP]进行解析。7-1-4和Teb1的p50依赖性活性解释了p50几乎存在于所有的EM结构中。
先前的重组实验结合了RRL组装的重组RNP催化核心、细菌表达的Teb1和内源性组装的四膜虫蛋白,这些蛋白在纯化的全酶微球菌核酸酶处理后仍与p45-f结合。通过这种方法分离的7-1-4复合物并没有完全解离p50, p50作为全长蛋白与p45-f共迁移。在之前的分析中,残留p50通过RNP催化核心和Teb1的互补来重建全酶样催化活性。使用完全重组全酶亚基的重组表明,7-1-4或Teb1对RNP催化活性的影响依赖于p50(图3g),这与它们在EM结构中的位置一致(图3e, f)。
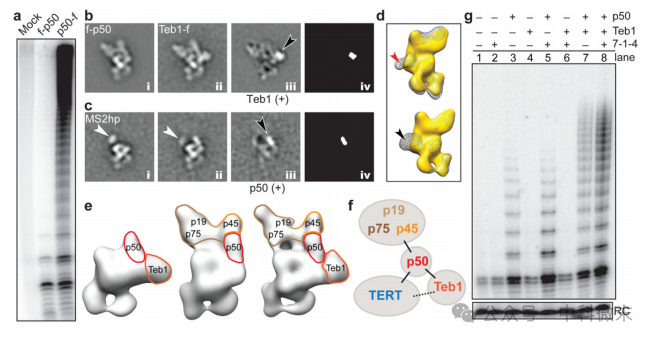
图3 p50锚定TERT,7-1-4和Teb1
Teb1结构域在位置上是灵活的
在类别平均和3D重建中,除了标记Teb1的端粒酶制剂外,所有端粒酶制剂都有约5%的Teb1颗粒缺失密度(图3e),因为Teb1在全酶亲和纯化过程中对蛋白质水解和解离很敏感。
为了鉴定结构中特定的Teb1结构域,该研究构建了在TERT内源位点上仅用串联蛋白A结构域(TERT-zz)进行C端标记的菌株,并在整合转基因中表达了N端标记的Teb1C或Teb1BC。通过TERT-zz/f-Teb1C或TERT-zz/fTeb1BC串联亲和纯化分离的全酶颗粒具有预期的高RAP(图4a),并且几乎相同的类平均和3D重建彼此之间以及纯化的Teb1-fzz(图4b-d)。由于Teb1C介导Teb1与其他全酶蛋白的结合,并且Teb1BC上的N端标签要求相关全酶含有Teb1B,得出Teb1B结构域具有位置柔性的结论,因此没有出现在类平均和3D重建中。同样,对于Teb1-fzz纯化的颗粒,它可以包含所有的Teb1NABC结构域,在大多数类平均中只有Teb1C可见。根据Fab标记确定的c端位置和最符合EM密度形状的位置,将Teb1C建模成EM密度图(图2a)。围绕y轴的方向不是确定的。观察到一小组f-Teb1BC和Teb1-f端粒酶的类平均(含有<5%的颗粒)显示Teb1C以上的弱额外密度(图4c,d)并分配给Teb1B。Teb1 NAB结构域相对于Teb1C的构象灵活性与复制蛋白A的旁系大亚基的结构研究一致,复制蛋白A具有三个DNA结合OB-fold结构域,只有在与ssDNA结合时才变得相对有序。
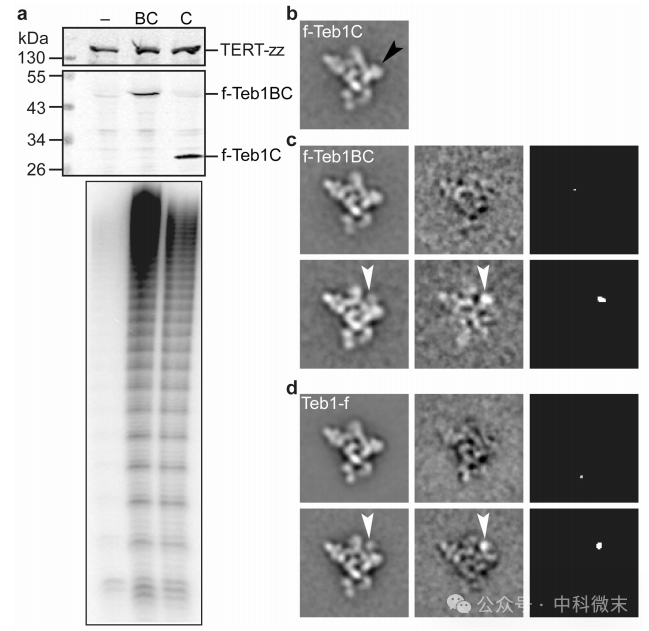
图4 Teb1结构域对全酶结构和活性的作用
7-1-4子复合体有多个方向
目前未知7-1-4亚基的高分辨率结构和确切的生物学功能。EM结构和之前的生化数据都表明它们构成了一个稳定组装的亚复合物。由于没有观察到缺少单个7-1-4亚基的类平均,因此只能从p19和p75的c端类平均和Fab标记推断亚基间边界,这表明这两个亚基非常接近。在所有类平均和3D重建中,主要颗粒类具有7-1-4构象,p75位于RNP催化核心的顶部(图1c),处于稳定构象(图5)。所有端粒酶样品都显示出7-1-4的其他位置,其中它远离RNP催化核心,同时保持与p50的物理连接(图5)。在这些其他构象中,7-1-4所在的类平均区域比粒子的其他部分显得“模糊”,这与端粒酶全酶的这部分构象变异性一致。在每次纯化的类别平均值的约10%中,包括标记p75或p45的类别,没有可见的7-1-4密度,表明相对于RNP催化核心,没有很好地定义一些7-1-4位置(图3e2)。MS2cp与MS2hp全酶的结合似乎有利于p75从稳定位置位移(图1c)。对各种构象的分析表明,7-1-4是作为一个完整的亚结构旋转的(图5)。端粒酶产物DNA可以与p45以及TERT TEN域交联,这表明7-1-4至少有一些取向必须使p45接近DNA。
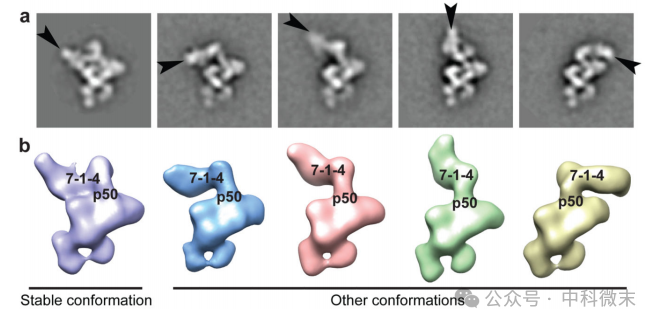
图5 7-1-4的位置动态
对全酶组装和功能的影响
TERT和TER包含一组最小的端粒酶亚基,用于体外重复合成,但是TERT和TER之间的3D排列和分子相互作用尚未明确。尽管在大小和序列上存在差异,TER包含一个t/PK结构域,几乎无一例外地包含一个单独的激活结构域(四膜虫中的SL4),每个结构域都有一个TERT结合位点。
在此研究中,研究者明确地定位了构成TERT已知结合位点的四膜虫TER的两个元素,并模拟了整个TER的路径。在脊椎动物的TER中,与Tetrahymena TER远端SL4功能相当的是P6/P6.1,它与L4所在的TRBD区域交联。因此,该研究的模型结构提供了对人类端粒酶TERT-TER相互作用的见解,并解释了TERT结合元件在TER的t/PK和激活域上的可分离相互作用。通过对16种不同标记的端粒酶样品进行分类和/或三维重建,能够确定全端酶的亚基排列。EM结果与重组酶活性分析一起表明全酶功能单元的细分:TERT-TER-p65催化核心,p50-Teb1和7-1-4。重组全酶亚复合物的结构测定和活性测定都确定了p50的中心作用,p50是一个缺乏任何预测结构域折叠的亚基。EM数据显示,7-1-4是一个相对于全酶其余部分具有动态取向的结构单元。研究者认为7-1-4配位端粒酶全酶的位置动力学具有额外的端粒合成或加工活性。p50与RNP催化核心的组装极大地增加了重复序列的合成,这表明p50稳定了酶与ssDNA的关联,其潜在的功能类似于酵母Est3和脊椎动物TPP144-48,有利于产生TERT TEN结构域构象和/或提供额外的ssDNA接触。随后p50依赖性的Teb1募集进一步增强了活性,推测这可能是通过p50和Teb1C OB折叠的相互作用发生的。EM测定的p50的物理位置和重构测定的p50的功能意义表明,p50可能是体内全酶组装的关键决定因素。
总结
该研究揭示了RNP催化核心的精细结构,并阐明了赋予持续性和连接端粒酶与端粒的亚基组织。第一次提出了对完整的端粒酶全酶结构的解析,为端粒酶与端粒底物偶联的生物学机制提供了前所未有的理解。
更多关于端粒酶的文章戳下方链接了解~
冷冻电镜成像技术(Cryo-EM,Cryo-ET)能够在分子生物学研究、细胞学研究乃至医学临床研究中发挥决定性的作用。鑫研微末团队在生物大分子及其复合体的三维结构解析方面技术成熟、经验丰富,能够在原子级分辨率解析多种生物大分子材料的三维结构。我们致力于为药企和高校医院科研工作者提供一站式、自动化、原子级分辨率的结构解析服务,助力创新药物研发和科研成果转化,帮助科研人员实现“原子结构自由”。
#衰老 #Cryo-EM #蛋白质 # 多蛋白复合物 # 端粒酶
加州大学洛杉矶分校化学与生物化学系Jiansen Jiang和Edward J. Miracco为本文第一作者,Z. Hong Zhou, Kathleen Collins和 Juli Feigon为本文共同通讯作者。
原文链接:https://www.nature.com/articles/nature12062
点击“阅读原文”,查看文章↓
如果您对特定研究领域感兴趣,欢迎通过微信公众号搜索 “Cryo-EM”、“膜蛋白”、“纳米颗粒”等关键词,查阅我们过往发布的相关文章。
您还可以添加我们的研究助理,以便进行更详细和具体的咨询。我们将竭诚为您提供帮助。
内容中包含的图片若涉及版权问题,请及时与我们联系删除





评论
沙发等你来抢