药物发现与预测模型的必要性

Drug-Target Interaction/Affinity Prediction: Deep Learning Models and Advances Review
https://arxiv.org/abs/2502.15346
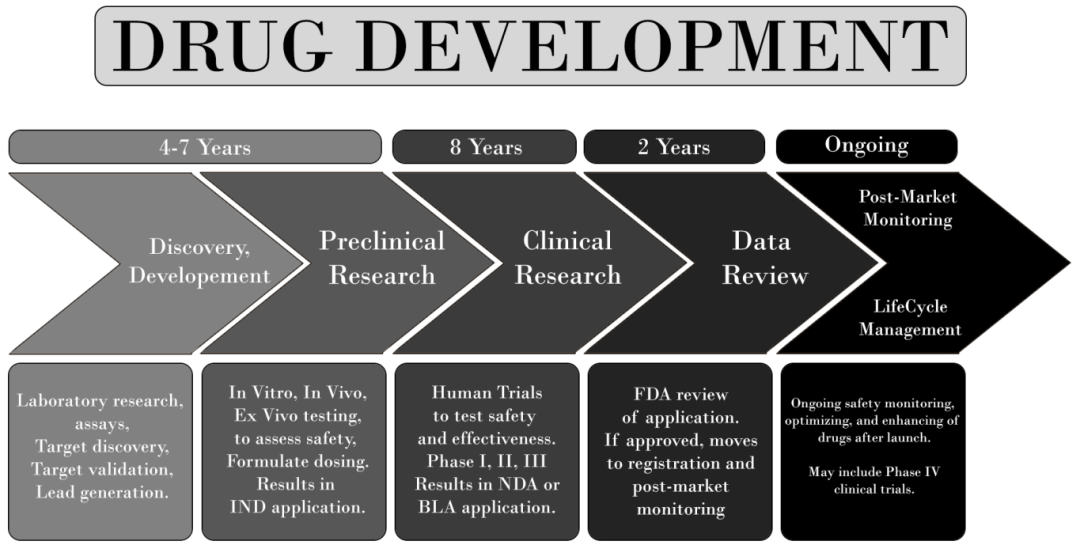
论文指出,传统实验方法(如高通量筛选)受限于分子结构的复杂性,难以捕捉药物与靶点间的细微作用机制。传统药物研发成功率仅0.01%,平均需筛选百万分子才能获得一种有效药物。而深度学习通过构建复杂的神经网络模型,能够从海量数据(如药物分子结构、蛋白质序列)中自动提取特征,精准预测药物-靶点相互作用(DTI)。深度学习模型在亲和力预测(DTA)任务中,MAE可低至0.5 pKd单位,较传统方法提升30%以上。例如,2016年提出的DeepDTA模型,首次尝试将蛋白质序列和药物SMILES字符串编码为神经网络输入,通过双通道卷积层融合信息,显著提升了预测精度。
这一技术的核心在于“数据驱动”。随着公共数据库(如PubChem、PDB)的飞速增长,研究者得以训练出能覆盖百万级分子对的模型。然而,挑战依然存在:如何平衡模型的预测能力与计算效率?如何解决“冷启动”问题(即对新靶点或新药物的预测能力不足)?这些问题成为后续研究的关键方向。
输入表示方法解码“分子密码”:药物与靶点的“语言翻译”
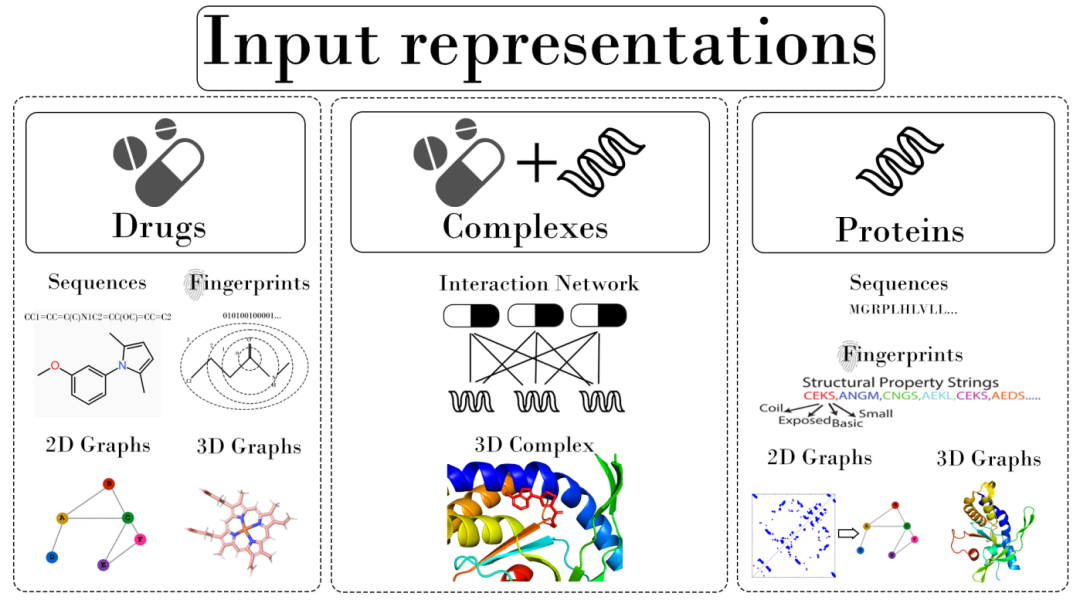
药物与靶标的高效表示是模型性能的基础。要实现精准预测,首先需要破解药物与靶点的“分子语言”。论文将输入分为药物、靶标及复合体三类:
1.药物表示:
序列表示:SMILES和SELFIES是主流符号系统,RDKit库可实现分子图与序列的互转,从“字母串”到“功能图谱”。
结构表示:分子结构(如SMILES、蛋白质口袋图)提供了更丰富的几何信息。深度学习模型通过图神经网络(GNN)或卷积神经网络(CNN),解析这些结构中的局部与全局特征。分子指纹(如ECFP、MACCS)通过二进制编码描述子结构;分子图(2D/3D)则用节点(原子)和边(化学键)建模拓扑关系。GNNs(如GCN、GAT)擅长处理分子图的拓扑结构。例如,DGraphDTA将蛋白质接触图转换为GNN输入,结合药物分子图,通过跨图注意力机制预测亲和力。
3D空间表示:点云、体素和网格化表示能捕捉立体构象信息,但计算复杂度较高。3D-CNN则聚焦于蛋白质口袋的三维空间网格,捕捉原子间距离与化学键信息。典型案例是PLANET模型,它将靶蛋白的3D结构分解为等效图(EGCL)和分子图(JT-VAE),通过多任务学习同步优化亲和力预测、接触图生成和分子距离矩阵,展现了结构建模的强大潜力。
2.靶标表示:
蛋白质序列:对于蛋白质靶点,氨基酸序列是最基础的输入形式。早期模型(如SVM、随机森林)依赖人工设计的特征(如氨基酸出现频率、二级结构标签)。而深度学习彻底改变了这一范式:Word2Vec与ProtBERT等预训练模型,将氨基酸序列映射为高维语义空间,捕捉远距离依赖关系。Transformer架构(如MT-DTI)通过自注意力机制,动态识别蛋白质中对结合关键的区域。以DeepAffinity为例,该模型采用seq2seq自编码器,将蛋白质和药物序列分别编码为上下文感知的特征向量,再通过交叉注意力模块计算交互能量。这种端到端的学习方式,无需手动设计特征,显著提升了模型的泛化能力。FASTA格式结合预训练语言模型(如ProtBERT、ESM-2)生成上下文感知嵌入。
结构信息:接触图(Contact Map)通过残基距离阈值构建稀疏图,AlphaFold预测的3D结构逐渐成为主流。
结合位点(Pocket):基于几何或深度学习的口袋检测算法(如PSG-BAR)显著提升了靶标特异性。
跨界融合,突破单一模态局限。许多模型意识到,单纯依赖序列或结构均存在信息缺失。于是,研究者提出序列-结构混合输入策略,这类模型在Cold setting(冷靶点/药物)任务中表现突出,因其能利用已知结构的“隐式知识”填补数据空白。
药物-靶标复合体的3D空间网格(如Pafnucy)或交互网络图(如SIGN)直接建模相互作用,但依赖对接模拟或晶体结构数据。
DeepFusionDTA将药物分子图(GNN)与蛋白质序列(BiLSTM)融合,通过多尺度注意力机制整合局部与全局特征。
PocketDTA更进一步,引入蛋白质口袋的几何特征(如溶剂可及性、静电势),结合药物分子的2D距离图,构建多模态输入体系。
药物-靶标复合体的3D空间网格(如Pafnucy)或交互网络图(如SIGN)直接建模相互作用,但依赖对接模拟或晶体结构数据。
DeepFusionDTA将药物分子图(GNN)与蛋白质序列(BiLSTM)融合,通过多尺度注意力机制整合局部与全局特征。
PocketDTA更进一步,引入蛋白质口袋的几何特征(如溶剂可及性、静电势),结合药物分子的2D距离图,构建多模态输入体系。
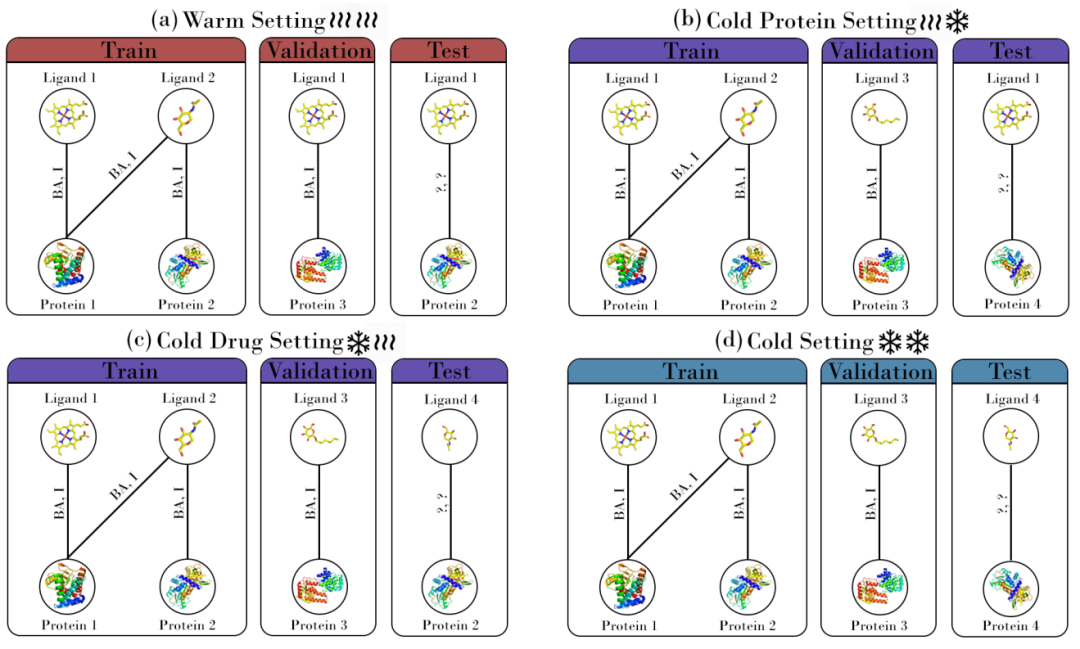
不同环境下药物-靶标相互作用问题概述。BA:结合亲和力,I:二元相互作用。(a)温热环境,(b),冷蛋白质环境,(c),冷药物环境,(d)冷药物-蛋白质环境。
重要性模型基于从谷歌学术获得的引用次数,近年来权重更高。由 https://www.wordclouds.com/ 制作
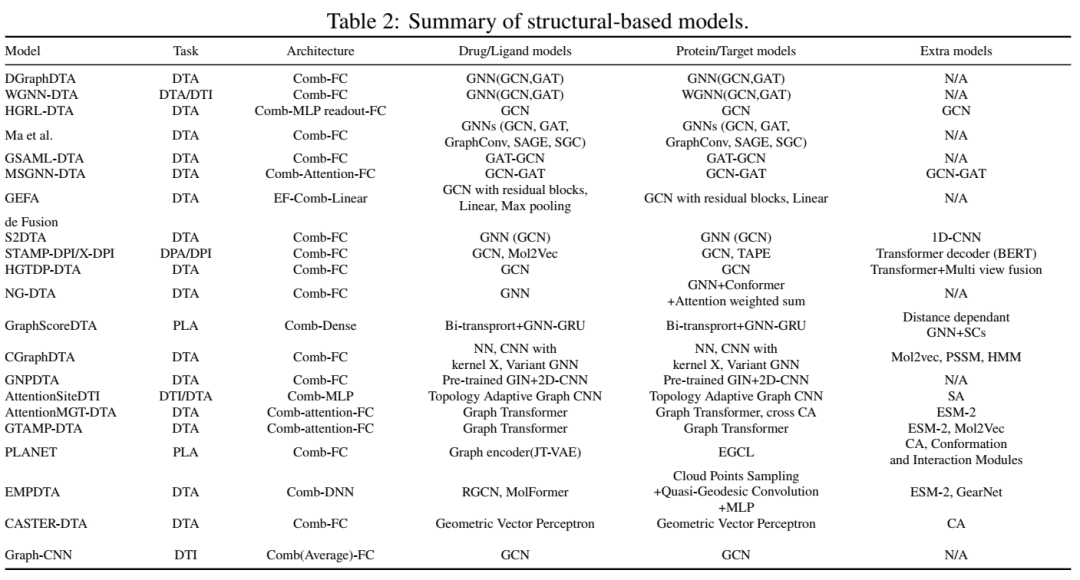
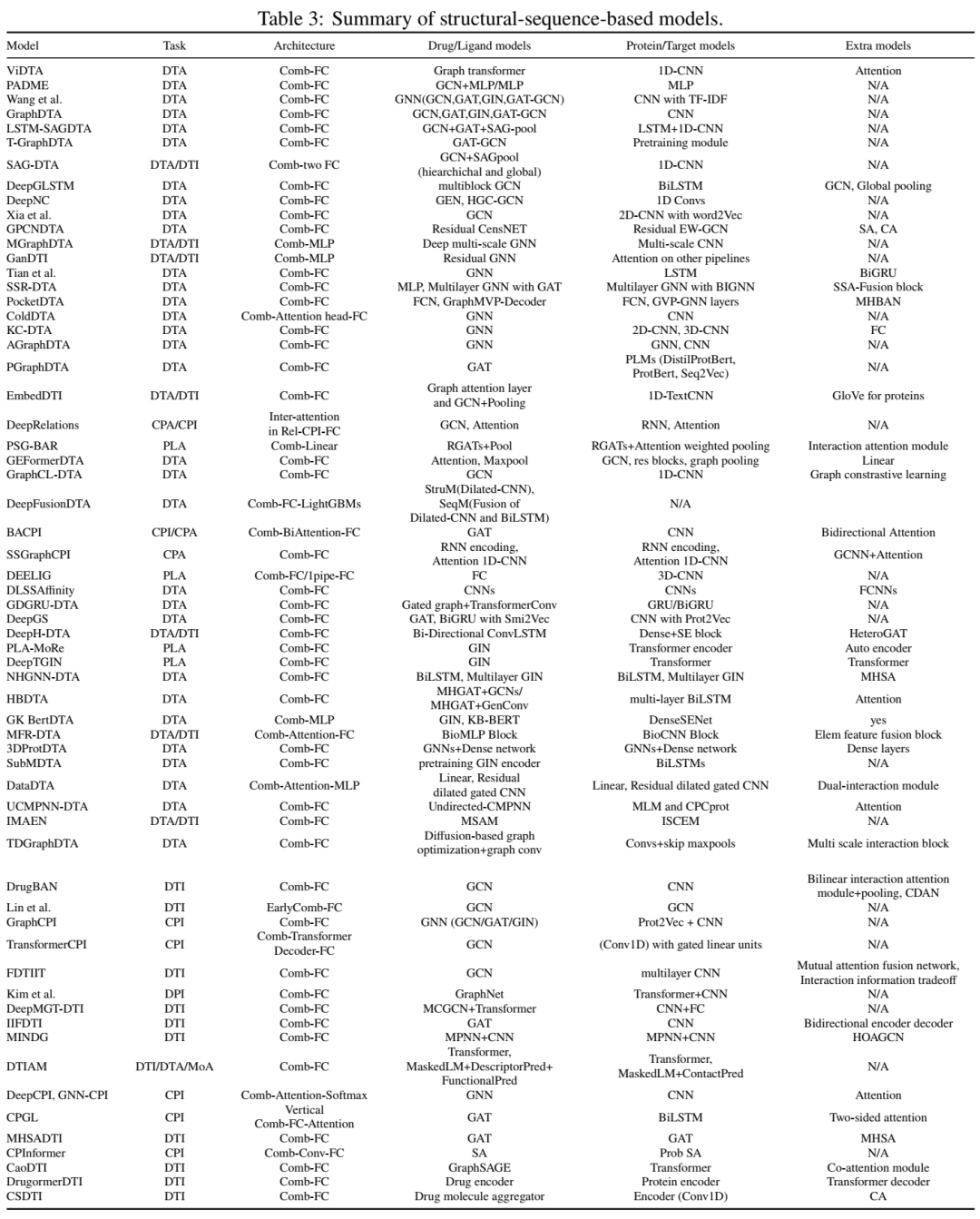
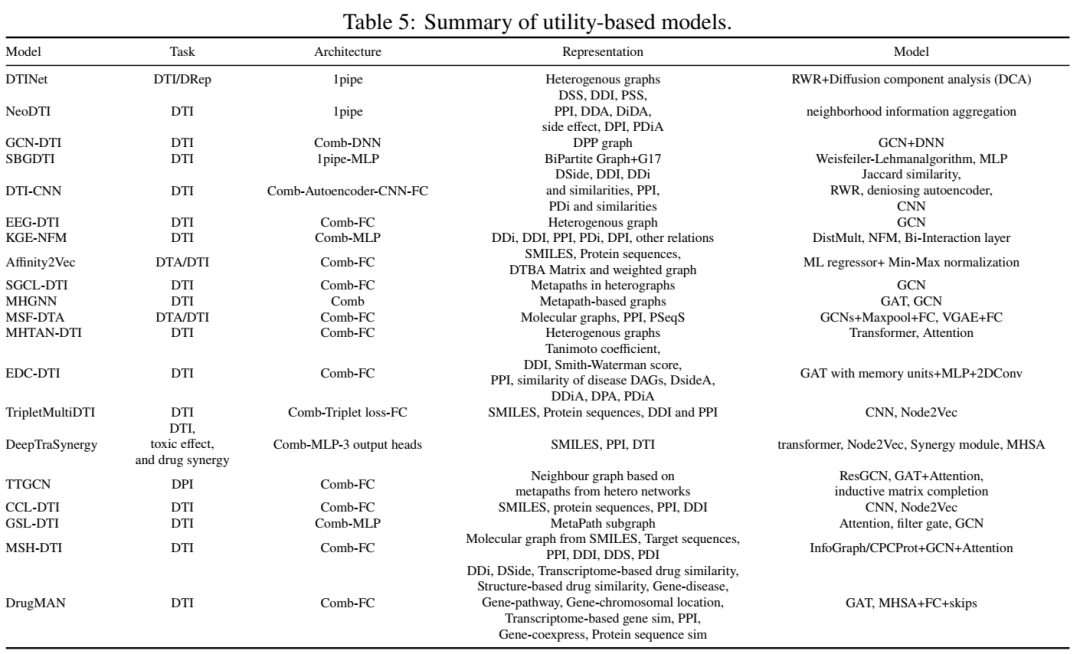
论文系统梳理了180种深度学习模型,按输入类型划分为四大类:序列、结构、混合及复合体四类,并总结了代表性工作:
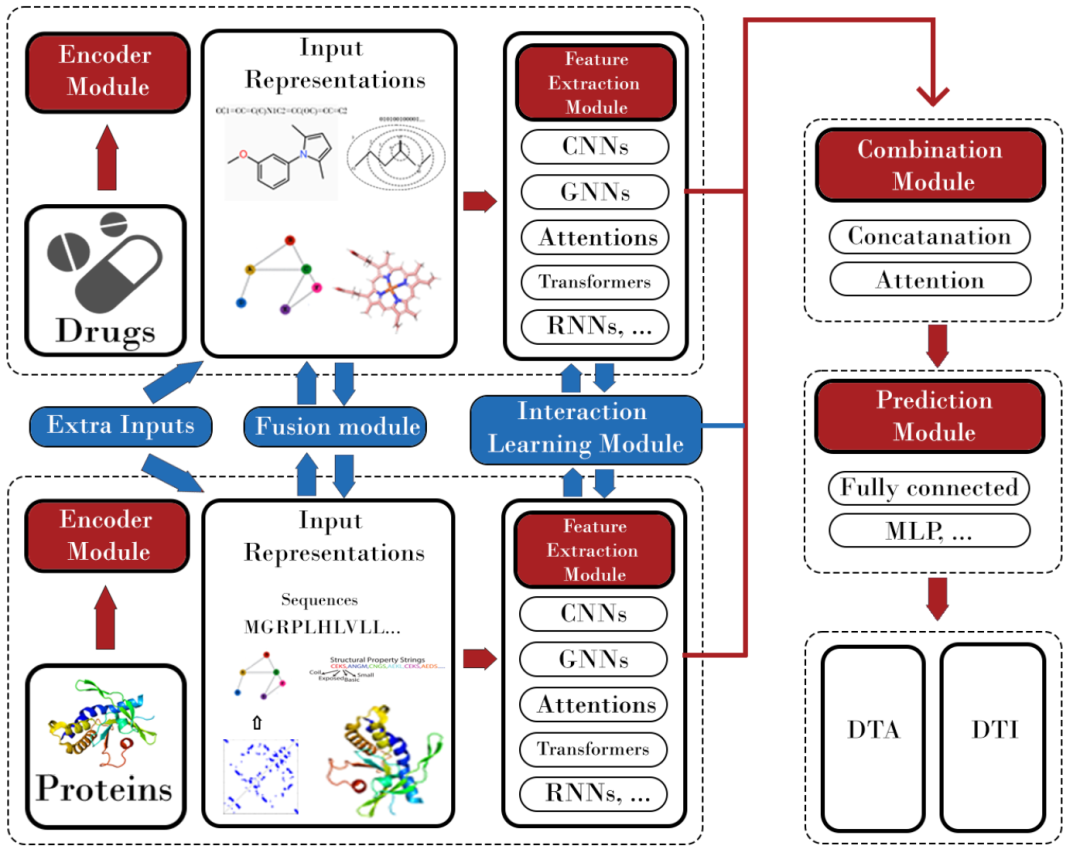
深度学习方法中使用的一般框架概述。大多数模型中都可以看到红色块。蓝色块是模型具有的模块。
1.序列模型:从“单兵作战”到“协同进化”
早期的序列基模型(如DeepDTA)仅独立处理药物和靶点序列,而新一代模型(如ML-DTI)通过互学习机制实现双向信息传递。此类模型在小样本场景(如罕见病靶点)中展现出优势,但需注意过拟合风险。
DeepDTA:双通道CNN分别处理SMILES和蛋白质序列,首次验证端到端学习的可行性。
WideDTA:引入配体最大公共子结构(LMCS)和蛋白质域特征,提升局部模式捕获能力。
TransformerCPI:利用预训练语言模型(如ChemBERTa)生成上下文敏感嵌入,AUPR提升15%。
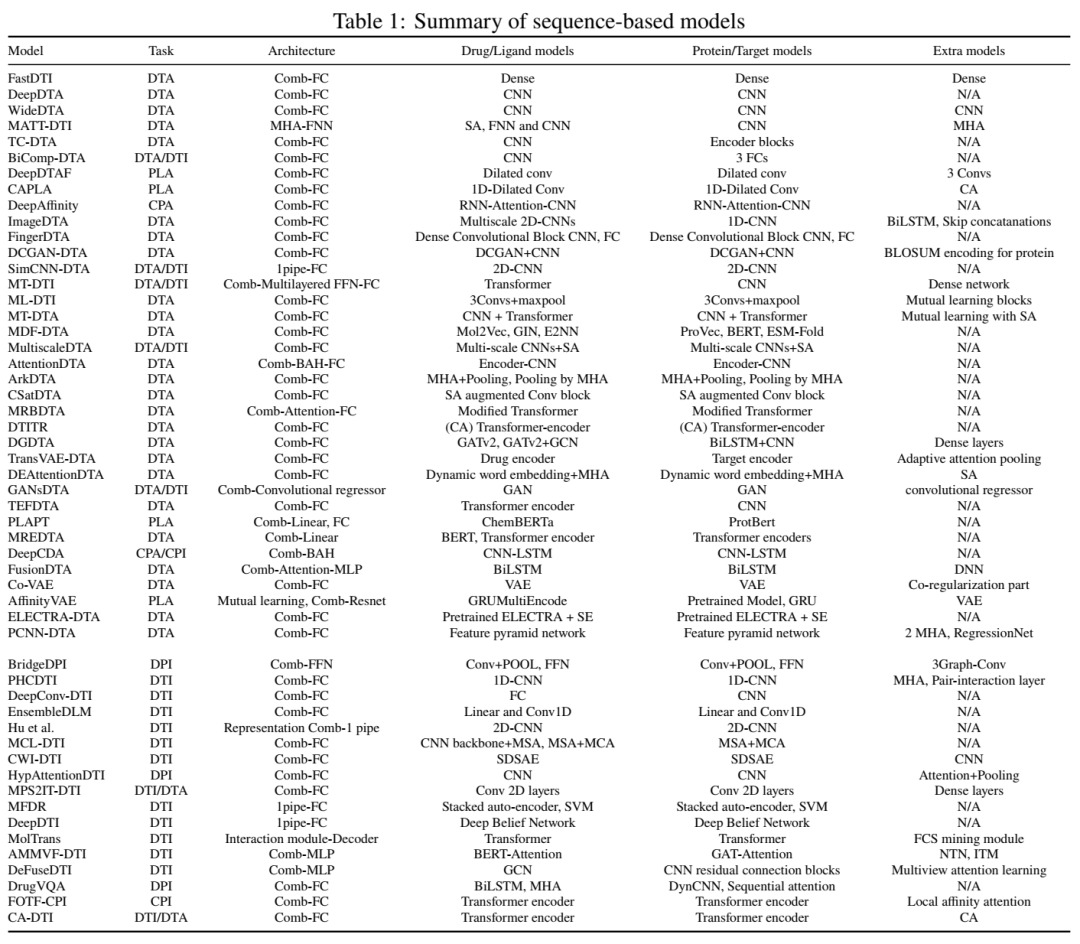
DGraphDTA:基于接触图的GNN编码蛋白质全局拓扑,Morgan指纹描述药物子结构。
PLANET:等变图卷积(EGCL)建模口袋动态构象,多任务学习预测结合亲和力与接触图。
DeepFusionDTA:融合序列(BiLSTM)与结构(GNN)特征,结合LightGBM集成策略,在KIBA数据集上RMSE达0.62。
MGraphDTA:多尺度GNN捕获原子级与基序级特征,Grad-CAM提供可解释性热图。
Kdeep:3D-CNN处理体素化复合体,物理化学通道(如疏水性、电荷)增强特征表达。
OctSurf:八叉树空间划分减少计算冗余,在PDBbind数据集上推理速度提升5倍。
Davis:包含442个激酶与68个抑制剂的pKd值,经去噪后保留9,125个高置信样本。
KIBA:整合Ki、Kd和IC50的标准化评分,涵盖229个靶标与2,111个药物。
PDBbind:2020版包含23,496个复合体结构,细化集(5,316样本)用于基准测试。
回归任务:MAE、RMSE、Pearson相关系数(PCC)和置信区间(CI)。
分类任务:AUPR、AUROC、F1分数,其中AUPR对类别不平衡更鲁棒。
数据泄漏问题:随机划分训练集可能导致模型过拟合已知药物-靶标对,冷启动评估(如孤儿靶标)更贴近真实场景。
负样本定义模糊:部分研究将未知相互作用视为负样本,可能引入噪声。
动态构象建模:开发异构图神经网络,统一建模分子、靶点、疾病之间的关系。结合AlphaFold 3预测的蛋白质动态构象与分子动力学模拟,开发等变时空图模型(如SE(3)-GNN),捕捉结合过程中的构象变化能量景观。
跨尺度建模:联结分子动力学模拟(原子级别)与深度学习(表观特征),建立从微观相互作用到宏观药效的完整链条。探索Transformer架构在时序建模中的应用(如蛋白质构象变化的时序预测)。构建蛋白质动态构象预测模型,结合AlphaFold2与深度学习,探索构象变化对结合力的影响。
多模态数据融合:整合基因组学(如单细胞测序)、表观组学(如疾病相关通路)与蛋白质组学数据,构建多尺度异质网络(如DrugMAN)、全景式药物-靶点图谱,提升靶标成药性预测。
可解释性与反事实推理:基于SHAP或反事实生成(CFG)解析关键原子/残基贡献,指导理性药物设计。例如,AttentionSiteDTI通过图注意力定位结合热点。研究注意力机制可视化工具(如CAM、Grad-CAM),揭示模型关注的关键残基或子结构。设计基于规则的约束网络(如Autoencoder with Logic Gates),引导模型生成符合化学先验的候选分子。设计可解释的几何注意力GNN,显式建模非共价相互作用,推动理性药物设计。
自主进化模型:构建强化学习框架,使模型能根据实验反馈动态调整预测策略。开发数字孪生靶点,通过虚拟突变库持续优化模型适应性。
低资源与小样本学习:采用元学习(MAML)或对比学习(SimCLR)从少量标注数据中泛化,解决罕见靶标或新药预测难题。开发自监督多模态DTI预测框架,融合基因组、表型与化学数据,突破小样本瓶颈。提出基于联邦学习的隐私保护DTI平台,促进跨机构数据协作,加速罕见病药物研发。
生成-预测联合框架:将扩散模型(如DrugDiff)与亲和力预测结合,Diffusion模型从头生成高亲和力分子(如Insilico的Chemistry42),生成高结合力分子并优化ADMET属性,实现闭环药物设计。
KIDA框架:通过知识蒸馏将教师模型(基于复合体)的物理约束迁移至学生模型(仅需2D结构),在无晶体数据时MAE降低12%。
3DProtDTA:利用AlphaFold预测的蛋白质结构与3D分子图,结合几何深度学习,在孤儿靶标上AUROC突破0.85。
- 个性化医疗:基于患者基因组的精准靶点预测。
- AI辅助新药发现:从虚拟筛选到临床前候选物的全流程自动化。
- 全球健康普惠:通过开源模型加速罕见病药物研发。
2.结构模型:从“静态图”到“动态演化”
GNNs虽在静态结构建模上取得成功,但真实生物系统是动态的。最新进展(如EMPDTA)开始融合动态信息。利用AlphaFold预测蛋白质构象变化,构建时间轴上的结构演变模型。通过云点采样(Cloud Points Sampling)捕捉蛋白质口袋的柔性区域。这一方向有望突破传统结构建模的刚性限制,实现更真实的相互作用模拟。
3.混合模型:从“二维平面”到“四维时空”
少数尖端模型(如PotentialNet)直接处理蛋白质-配体复合物的3D结构。将原子坐标编码为高维张量,通过三维卷积捕捉局部化学环境。结合几何注意力(如EGNN),显式建模非共价相互作用(氢键、疏水效应)。尽管此类模型计算成本高昂,但其对物理规律的显式建模,为理性药物设计提供了新思路。
4.复合体模型:
结构模型(如GEFA)在冷靶标设定下表现稳健,AUROC达0.92,较序列模型高10%。复合体模型(如SIGN)依赖高质量结构数据,预测精度最高(MAE=0.45),但泛化性受限。

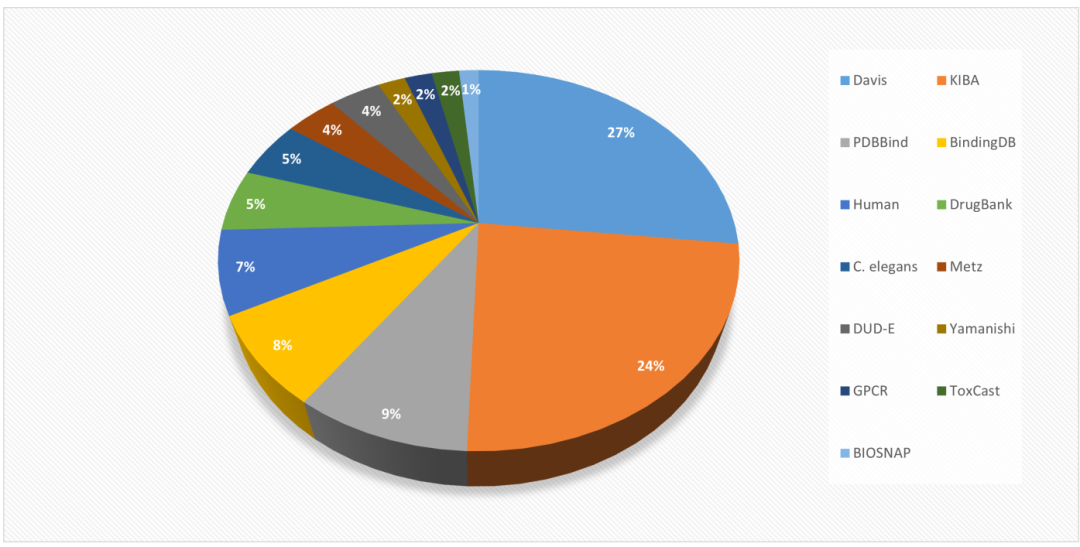
在检查的模型中出现的数据集的部分。
数据集与评估标准
模型的性能离不开高质量数据集与科学评估指标。论文详细梳理了主流数据集与评价指标:
1.数据集的“三重困境”
规模不足:公开数据集(如KIBA、Davis)仅覆盖数万对药物-靶点,远小于实际需求。标注偏差:多数数据集来自实验室筛选,缺乏真实临床环境的噪声数据。冷靶点稀缺:罕见病靶点的数据几乎为零,模型泛化能力面临挑战。为解决这一问题,研究者提出数据增强策略(如SMILES扰动、虚拟筛选)和联邦学习框架,以安全合规的方式共享私有数据。
2.评估指标的“双刃剑”
回归任务(预测亲和力)常用RMSE、MAE,但高值不等于高结合活性。分类任务(预测相互作用)需谨慎选择阈值,避免误判“假阳性”。论文呼吁建立更全面的评估协议,结合体外实验(如表面等离子共振)验证模型预测结果。
3.挑战与陷阱:
未来研究方向与创新点:从“预测”到“设计”的跨越
基尽管深度学习已显著提升DTI预测能力,但距离真正的药物设计仍有差距。于论文总结的挑战,提出以下前沿方向:
创新案例:
通过持续的技术迭代与跨学科合作,深度学习必将重塑药物研发的范式,为人类健康开辟新纪元。
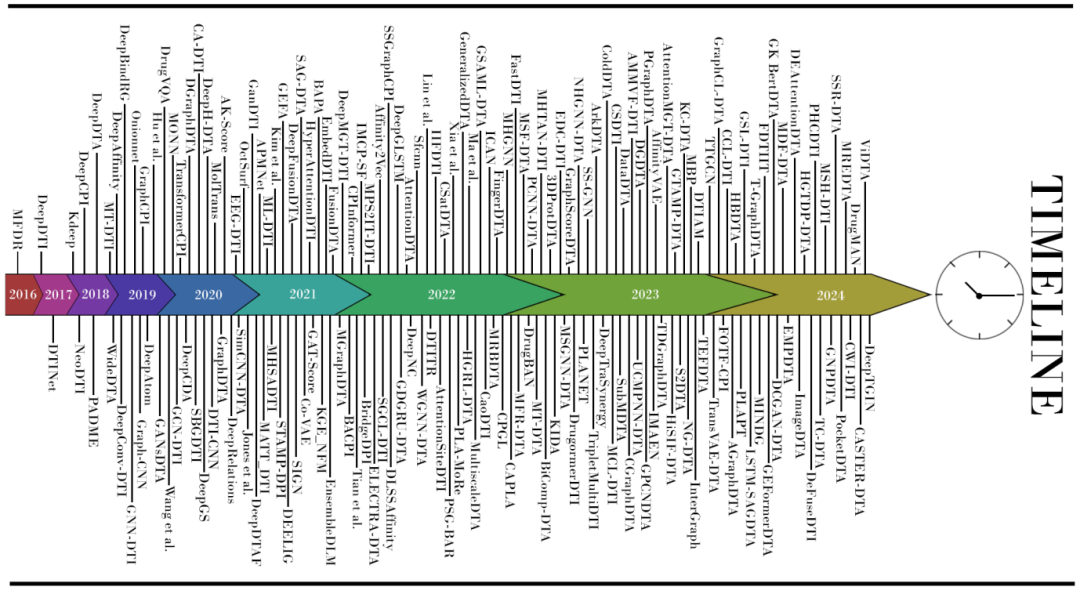
模型时间线发布日期
结语:AI制药的下一个十年
本文系统回顾了深度学习在药物-靶标预测中的进展,从输入表示、模型架构到评估标准均提供了深度解析。未来,随着多模态学习与生成式AI的融合,药物发现将步入“AI驱动设计”的新纪元。研究者需在提升精度的同时,关注模型的可解释性与临床转化价值,最终实现从算力到疗效的跨越。
这篇论文不仅是技术演进的总结,更是对未来方向的宣言。随着深度学习与生物计算的深度融合,我们有理由期待:
然而,技术突破的背后仍需警惕过度依赖数据的风险——毕竟,生物学系统的复杂性远超当前算法的想象。唯有坚守科学伦理,平衡创新与严谨,才能真正让AI成为“救命药”的守护者。

微信群


内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢