
如今,Evo迎来了重大升级——Evo 2。Evo 2基于NVIDIA的DGX Cloud平台构建,并在覆盖生命三域(原核生物、古菌、真核生物)超12.8万个物种基因组上进行训练,累计处理9.3万亿核苷酸序列。与仅关注原核基因组的Evo相比,Evo 2纳入了来自人类、植物以及真核生物域中其他更复杂的单细胞和多细胞物种信息,实现了前所未有的跨物种泛化,并显著拓宽了其应用范围。
在技术层面,Evo 2模型使用了一种名为StripedHyena 2的新架构,其基于卷积的多混合设计,可实现三倍优于传统Transformer的训练效率提升;并能捕捉基因组的相互作用,自主学习外显子—内含子边界以及转录因子结合位点等信息。该模型还具有400亿个参数,与Meta、DeepMind或OpenAI发布的当前主流大语言模型处于同一量级。Evo2囊括了生物学的基本语言(DNA、RNA 和蛋白质),显著扩大了上下文窗口,能一次性处理多达100万个碱基对,这使得其能够理解基因组中相距较远部分之间的关联。
Arc研究所的联合创始人Patrick Hsu博士表示,这种长上下文处理能力突破基因组远程调控解析的技术瓶颈、解锁了多个分子尺度,可从短生物分子(如tRNA)或基因簇(如操纵子)到整个细菌基因组或真核生物染色体,这使Evo 2成为多模态和多尺度生物建模领域的领导者。
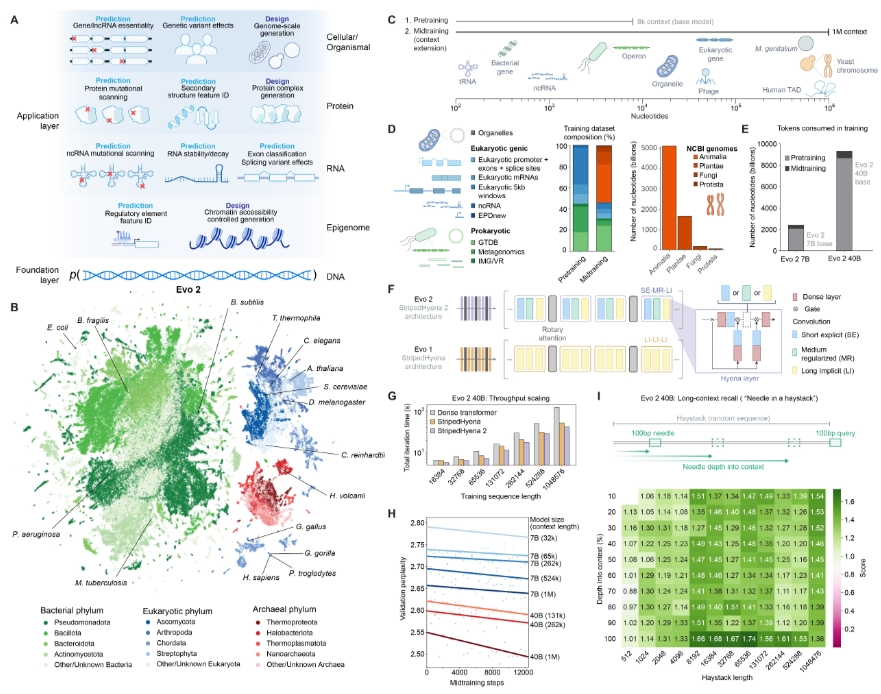
Evo 2 的扩展训练数据和优化架构使其能够在各种生物应用中表现卓越。
在医疗保健领域,了解哪些基因变异与某种疾病相关,对于治疗方法的研发来说至关重要。初步验证表明,Evo 2模型可以确定基因突变如何影响蛋白质、RNA和生物体适应性,预测BRCA1基因(与乳腺癌和卵巢癌风险相关)致病突变的准确率超90%。
Evo 2是唯一能够预测编码突变和非编码突变影响的模型。Patrick Hsu博士说道:“对于编码突变的预测,它是第二优秀的模型;但在非编码突变方面,它处于最先进的水平,其超越了DeepMind的AlphaMissense等单任务模型无法对非编码突变进行评估这一局限。”
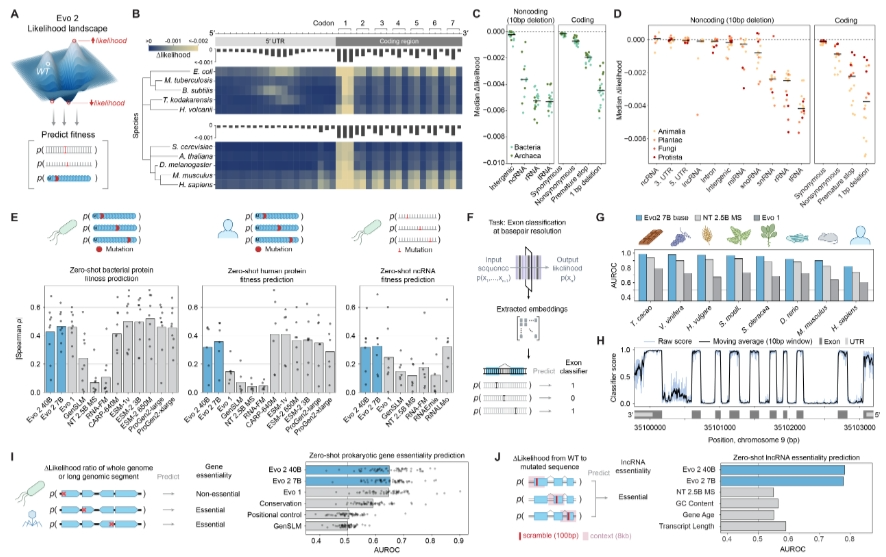
由于生物功能并非由单个蛋白质分子独立完成,构建合成基因组可为研究更广泛的生物背景提供有利条件,而Evo 2正致力于此。斯坦福大学化学工程助理教授Brian Hie表示:“到目前为止,许多生物设计都聚焦于分子层面。如果我们有一个强大的模型,能够在完整生物体的尺度上进行生成,这将开启许多具有广泛应用场景的下游任务。”
在预印本论文中,研究团队描述了Evo 2在三项跨越不同基因组复杂程度的设计任务中的表现:1.线粒体基因组;2.常用最小基因组模型——生殖支原体原核基因组;3.代表真核生物的酵母染色体。在这三项任务中,Evo 2均展现出良好的基因组连贯性。例如,在线粒体基因组设计中,成功构建了编码电子传递链所有成分的基因(由AlphaFold 3预测);在酵母染色体设计中,不仅存在天然同源物,还准确重构了内含子等更复杂的基因组结构。
在农业方面,Evo 2可通过提供对植物生物学的见解并帮助科学家开发更具气候适应性或营养更丰富的作物品种来帮助解决全球粮食短缺问题。在其他科学领域,Evo 2可用于设计生物燃料或设计分解石油或塑料的蛋白质。
·END·
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢