DRUGAI
近年来,单细胞RNA测序(scRNA-seq)技术的快速发展为解析组织异质性提供了前所未有的视角。然而,仅依赖基因表达数据难以全面捕捉细胞通路和复合物的变化,这些变化在蛋白质层面更为明显。此外,scRNA-seq 数据本身存在高噪声和零膨胀等挑战。为此,研究人员提出了一种融合蛋白质互作网络的分析方法,基于图神经网络的双视图架构,联合建模基因表达与蛋白互作信息。该方法在特定生物背景下学习基因间关系,并通过注意力机制优化细胞间表示。评估结果表明,scNET在基因注释、通路表征、基因关系识别以及细胞聚类和通路分析方面表现更优,适用于多种细胞类型与生物条件。

单细胞RNA测序(scRNA-seq)极大地推动了研究人员对复杂生物系统中细胞异质性的理解。传统分析流程通常采用基于图的聚类方法识别不同细胞群体,并进行差异表达分析以发现关键功能基因。
然而,scRNA-seq 数据存在严重的零膨胀问题,这些零值混合了生物信号和技术噪声,导致真实信号被掩盖,进而削弱基因间相关性,限制了对通路和复合物的识别。尽管已有多种数据填补方法被提出,如概率模型、邻近细胞平滑和潜在空间学习等,但它们多聚焦于数值恢复和细胞聚类,难以有效挖掘基因间功能关联。
同时,部分方法试图从技术误差中还原真实信号,但普遍缺乏对先验知识的利用。蛋白质互作网络(PPI)作为一种强有力的功能背景资源,能够揭示通路、复合物及信号转导关系。然而,PPI 网络多基于整体尺度构建,未能体现细胞类型或生物状态下的动态变化。
因此,将PPI网络与scRNA-seq数据融合,有望提升下游分析性能。研究人员此前已展示数据特异性PPI加权策略在识别细胞间信号通路中的潜力,并提出利用PPI结构传播基因表达、筛选关键基因等方法,有助于改善降维、填补和相似性分析等任务。
与此同时,自然语言处理和大语言模型的进展也被引入单细胞研究,尽管这些方法在大规模数据中表现出色,但通常需要监督微调,限制了其在小规模、无标签数据集中的应用。因此,亟需一种适用于未标注scRNA-seq数据的无监督框架。
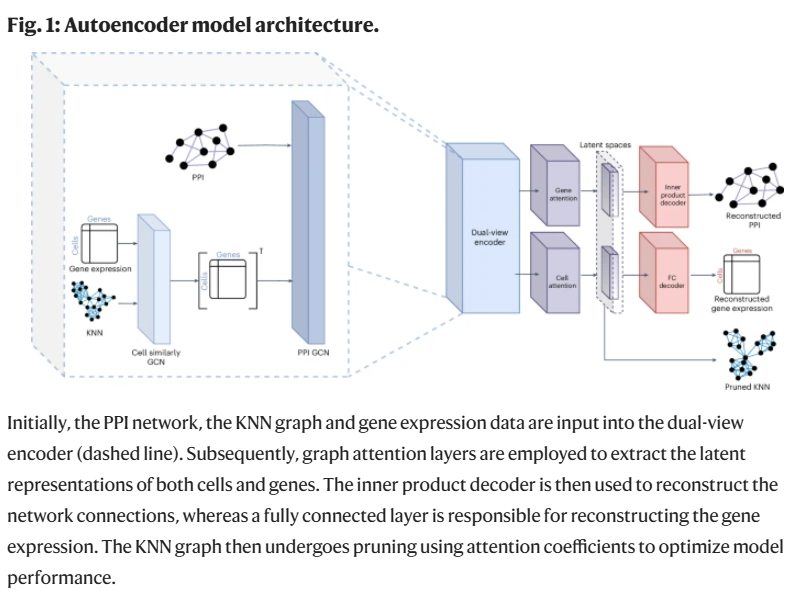
在此背景下,研究人员提出 scNET ——一个同时建模基因–基因与细胞–细胞关系的图神经网络方法。scNET 在蛋白互作网络和细胞表达相似性图上交替传播信息,实现数据去噪与上下文特异性嵌入学习。该方法引入注意力机制优化细胞KNN图,打破每个细胞连接数固定的常规假设,更贴近真实生物系统。基因与细胞的双视角表达建模有助于捕捉多层次的生物学关系,并广泛应用于下游任务。
实验结果表明,scNET在基因关系解析、细胞聚类和差异通路识别等方面优于现有方法,展示出更强的泛化能力和生物学解释力。
结果
研究人员提出了 scNET ——一种融合 scRNA-seq 与 PPI 网络的深度学习框架,通过双视角编码器同时建模基因–基因和细胞–细胞关系。在多个数据集中,模型表现出良好的收敛性和高效的计算效率。scNET 能根据基因表达动态地整合 PPI 信息,有效构建条件特异的嵌入空间。
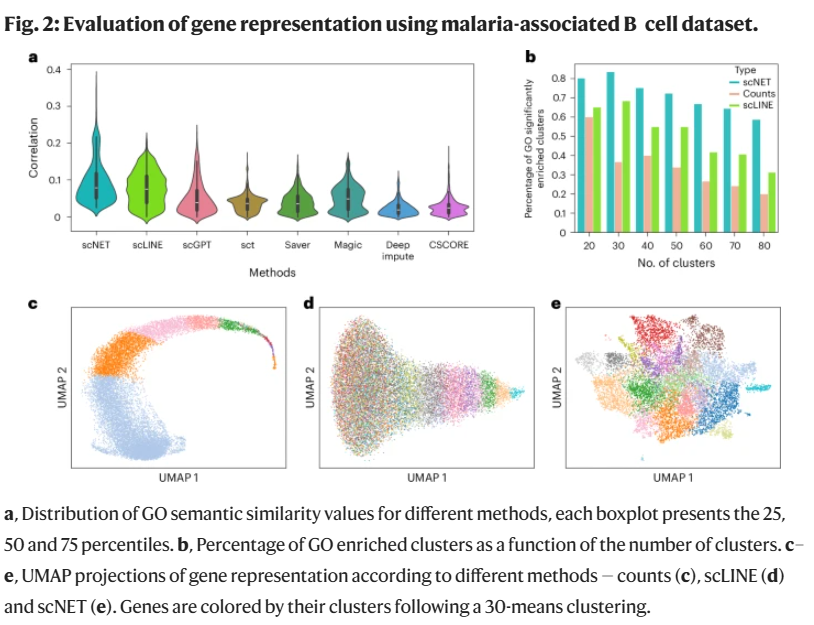
scNET 基因嵌入更好地捕捉功能注释信息
研究人员首先评估了嵌入空间与已知生物功能之间的相关性。scNET 显著提高了 GO 功能相似性与基因对间嵌入相关系数的平均值(最高相关性达 0.5),明显优于多种主流方法。基于 K-means 聚类及 GSEA 富集分析,scNET 显示出更强的功能聚类能力。同时,UMAP 可视化结果表明其在基因空间中的聚类更清晰,结构更紧凑。使用多标签分类模型预测 GO 注释进一步验证了嵌入空间的功能表示优势。
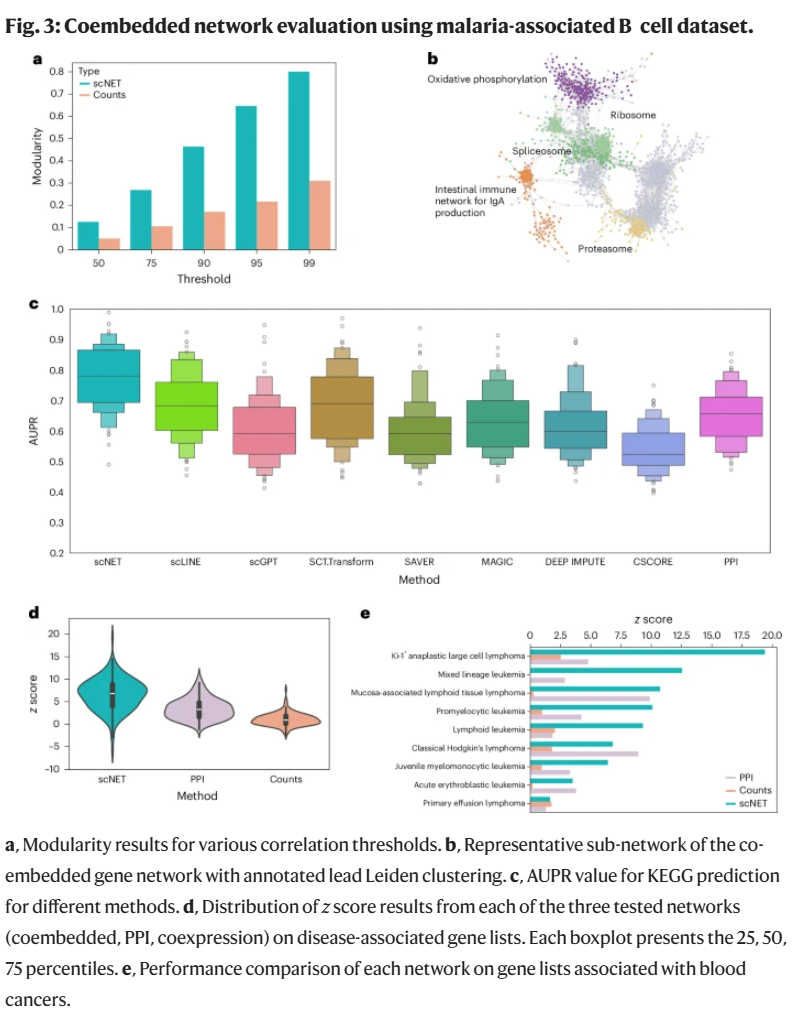
共嵌入网络捕捉生物通路结构
研究人员构建了融合 PPI 与共表达关系的共嵌入网络,并分析其模块性和路径重构能力。在不同相关性阈值下,scNET 嵌入空间构建的网络均表现出更高的模块性。在 KEGG 通路重构实验中,scNET 显著优于原始表达数据和 PPI 网络。在疾病相关基因预测中,scNET 网络在大多数白血病和淋巴瘤列表中表现最佳,展示其在稀疏网络中的泛化能力。
进一步地,研究人员在小鼠脑胶质母细胞瘤数据中构建了差异网络,揭示了 CD8+ T 细胞中 PD1 关联基因的变化,识别出如 Il2ra、Ifng、Cd28 和 4-1BB 等关键免疫调控因子,反映出该方法在功能关联层面的敏感性。
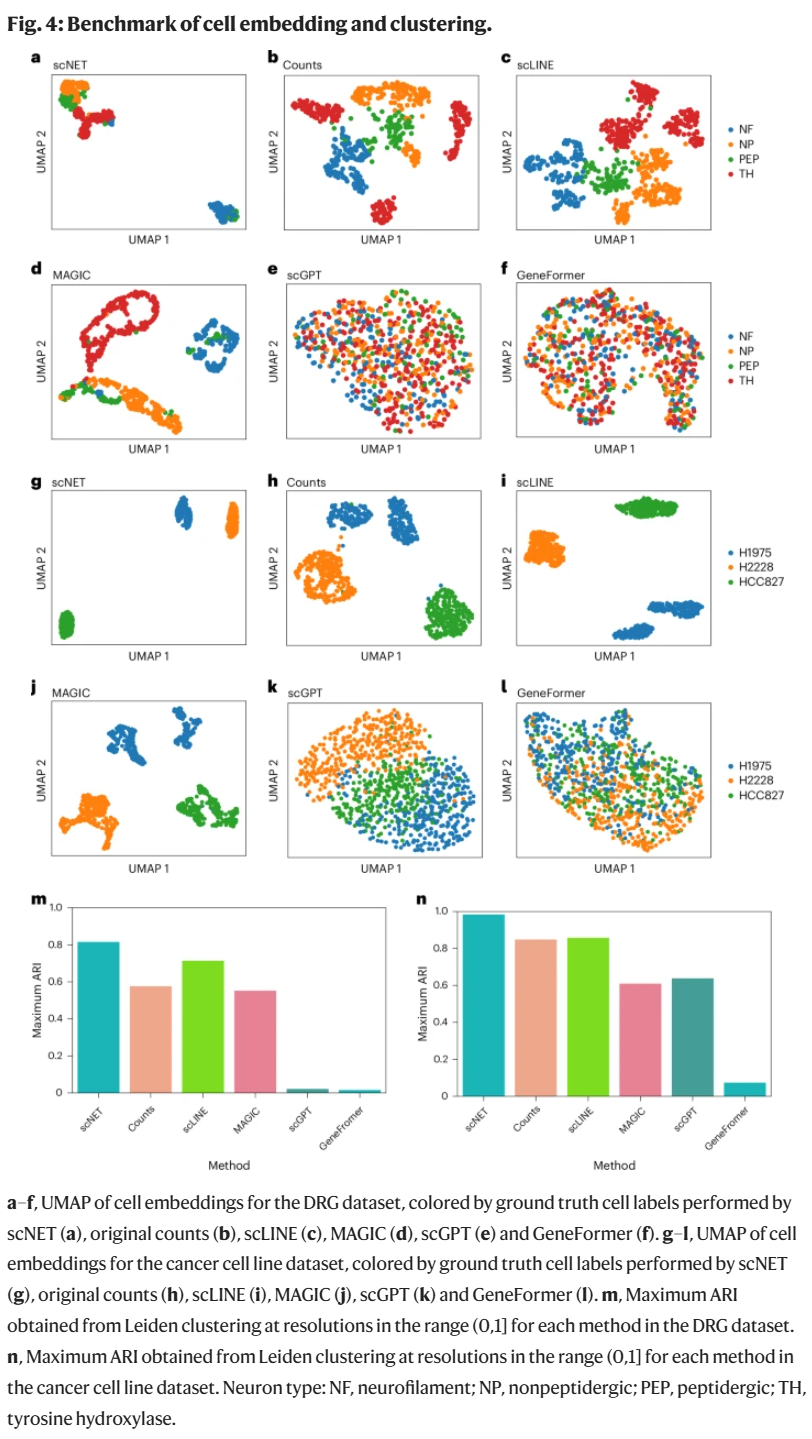
优化的细胞嵌入改善细胞聚类表现
在 Usoskin 和癌症细胞系两个带有真实标签的数据集上,scNET 明显优于传统方法和预训练模型(如 scGPT 和 GeneFormer),在 ARI 评分上表现最佳。UMAP 可视化进一步显示出细胞类型的清晰分离,尤其在难分群细胞如 H1975 和 G1/S 状态细胞中效果突出。scNET 的注意力机制成功识别出高质量与低质量边之间的差异,提高了 KNN 图的表达准确性。
重建表达减弱零膨胀,提升通路识别能力
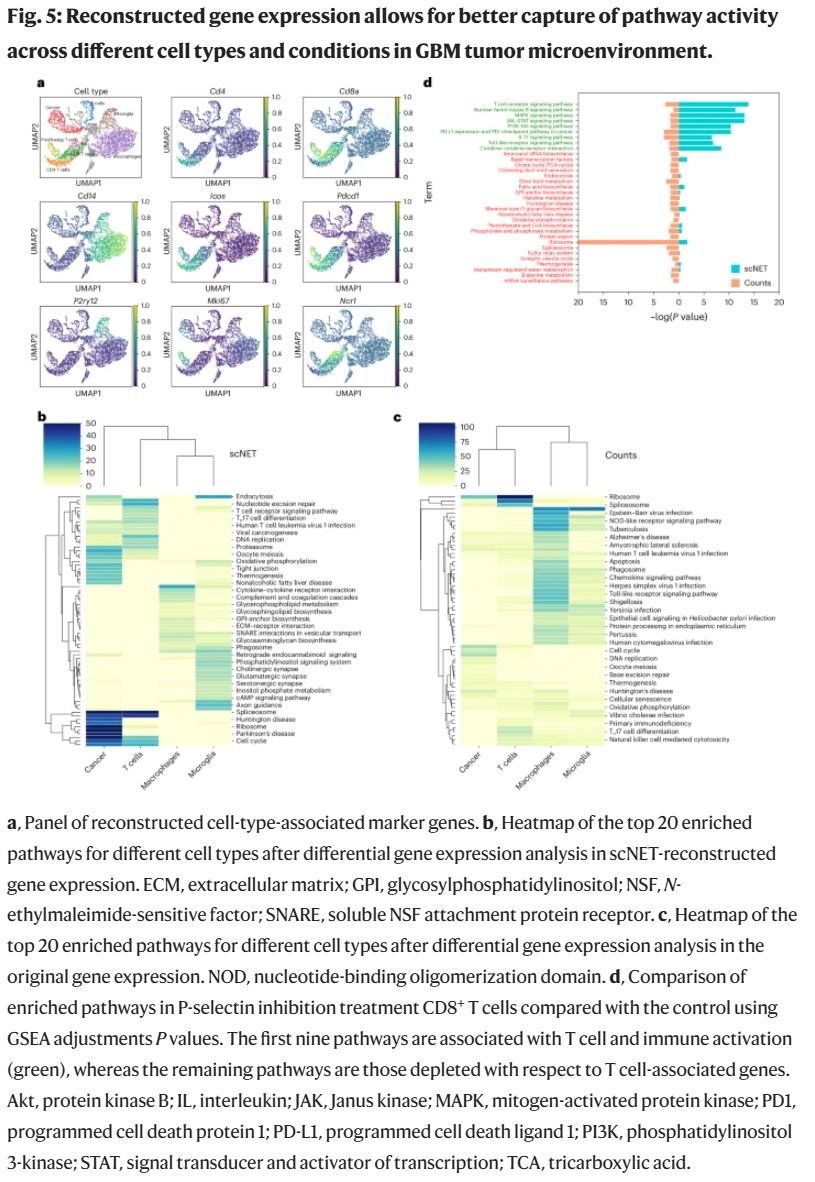
scNET 所重建的基因表达谱不仅重现了细胞特异性表达,还增强了对生物通路活性的识别能力。在 GL261a 脑瘤模型中,scNET 有效区分了 T细胞、巨噬细胞、小胶质细胞和肿瘤细胞,显著提升了 marker 基因的识别率,AUPR 得分均高于 MAGIC 和 DeepImpute。
在通路富集分析中,scNET 捕捉到了多个与各细胞类型密切相关的 KEGG 通路,例如 T细胞的 TCR 信号通路,小胶质细胞的突触调控通路,以及肿瘤细胞的增殖与代谢通路。相比之下,原始数据存在明显偏向,通路富集集中在巨噬细胞,且缺乏特异性。
在进一步分析 CD8+ T细胞对 P-selectin 抑制处理的响应中,scNET 能准确识别出多条与激活相关的 T细胞通路(如 TCR 与 NF-κB 信号),而原始数据未能检测到这些变化。使用阴性通路集进一步验证其通路识别的特异性,scNET 显示出更低的富集倾向,表现出更强的生物学可信度。
讨论
系统生物学的比较分析中,识别通路和复合物在不同生物状态下的激活差异,是深入理解复杂生物变化的关键。在传统的 bulk RNA-seq 分析中,共表达及差异共表达方法广泛应用于此类推断。然而,scRNA-seq 数据存在严重的零膨胀和弱相关性问题,导致上述方法在单细胞层面效果受限。因此,开发能够克服这些限制、深入挖掘状态特异性通路激活的新工具,对于发挥单细胞组学的潜力至关重要。
在本研究中,研究人员提出了一种前沿的深度学习框架 scNET,融合了 scRNA-seq 数据与蛋白质互作网络(PPI)。与传统数据集中样本和特征的明确分离不同,scNET 所构建的是一个双重视角的数据结构:细胞通过基因表达向量表征,基因通过其在不同细胞中的表达构成特征向量。研究人员认为,这种利用数据双重性的建模方式能够实现更优的表现。
为此,研究人员构建了一个基于图神经网络的自动编码器框架,包含两个图结构与一个节点特征矩阵。一个图捕捉细胞之间的表达相似性,另一个则表示基因间的互作关系。在模型训练过程中,表达信息在两个网络之间交替传播,使得模型能够同步优化细胞–细胞与基因–基因关系的表达。
通过一系列精细化的验证实验,研究人员展示了 scNET 在功能注释、通路识别与表达重建等方面相较现有方法的显著优势。特别是在 P-selectin 抑制处理后的 GBM 肿瘤微环境中,scNET 成功揭示了标准分析方法未能捕捉的通路激活变化,强调了其在更高维度生物学机制识别中的潜力。这一发现表明,与其局限于单基因水平,不如借助嵌入空间全面考察通路层次的变化,以获得更具系统性的洞察。
需要指出的是,尽管 PPI 网络为基因间关系提供了重要的先验信息,但其本身未覆盖诸如转录因子等间接调控机制,而这些调控在基因表达调节中至关重要。部分转录因子在不同条件下表达稳定,但其功能状态受定位或磷酸化等因素调控,只有结合如单细胞 ATAC-seq 等多组学技术,才能更全面补充现有框架的生物学上下文。
此外,研究人员还提出了一种独特的细胞–细胞相似性图优化策略。通过在 KNN 图中引入注意力机制,模型能够有效筛除低质量连接,突破了传统“每个细胞与 K 个邻居相似”的简化假设。这一机制不仅提升了嵌入质量,也为未来在 scRNA-seq 中发展基于注意力机制的细胞相似性建模方法提供了新方向。
值得一提的是,KNN 方法在更广泛的数据填补与建模任务中也有广泛应用。scNET 将其自然融入图神经网络架构中,为处理高噪声与缺失值提供了统一策略。因此,该框架具备良好的可迁移性,未来可应用于具有相似“样本–特征”双视角结构的其他数据类型中,如图像、用户行为矩阵、多组学融合等任务。
综上,scNET 不仅在单细胞组学中提供了强有力的分析工具,也为构建跨领域图学习模型提供了通用思路。
整理 | WJM
参考资料
Sheinin, R., Sharan, R. & Madi, A. scNET: learning context-specific gene and cell embeddings by integrating single-cell gene expression data with protein–protein interactions. Nat Methods (2025).
https://doi.org/10.1038/s41592-025-02627-0
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢