DRUGAI
在小分子、核苷酸和金属离子等配体的环境下进行蛋白质序列设计,对于酶、配体结合蛋白及生物传感器的开发至关重要。然而,现有基于深度学习的方法难以有效建模非蛋白组分。研究人员提出了一种新方法——LigandMPNN,可显式建模生物分子体系中的所有非蛋白组分。在与小分子(63.3% vs. 50.4%/50.5%)、核苷酸(50.5% vs. 35.2%/34.0%)和金属离子(77.5% vs. 36.0%/40.6%)相互作用残基的原序列恢复任务中,LigandMPNN显著优于Rosetta与ProteinMPNN。该方法不仅生成序列,还预测侧链构象,从而实现对结合相互作用的精细评估。研究人员已利用LigandMPNN设计出100余种经实验证实具有高亲和力和高结构准确性的结合蛋白,其中包括4个X射线晶体结构。此外,该方法可显著提升已有设计的结合能力,亲和力提升最高达100倍。LigandMPNN有望广泛应用于新型结合蛋白、传感器和酶的设计中。

蛋白质的de novo设计可用于构建具备全新功能的蛋白质,如催化、小分子/金属/DNA结合以及蛋白互作等。该过程通常包括三步:第一步,生成适合目标功能的蛋白骨架结构;第二步,为每个骨架设计氨基酸序列,以实现正确折叠及功能所需的特异性相互作用(如酶活性位点);第三步,利用结构预测方法筛选序列与结构的兼容性。
本研究聚焦于第二步——蛋白质序列设计。目前已有基于物理模型(如Rosetta)和基于深度学习的方法(如ProteinMPNN、IF-ESM等)来解决这一问题。深度学习方法在骨架序列设计中优于传统物理方法,但现有模型尚无法考虑非蛋白原子或分子。例如,ProteinMPNN仅考虑蛋白主链坐标,忽略了对酶、核酸结合蛋白和传感器等设计至关重要的原子环境。为此,研究人员提出了新的解决方案。
结果
为实现多样化的蛋白质功能设计,研究人员开发了一种新型深度学习模型 LigandMPNN,用于在完整非蛋白原子上下文中进行蛋白质序列设计。该模型在ProteinMPNN的架构基础上扩展,引入小分子、核苷酸和金属等配体原子的结构信息。
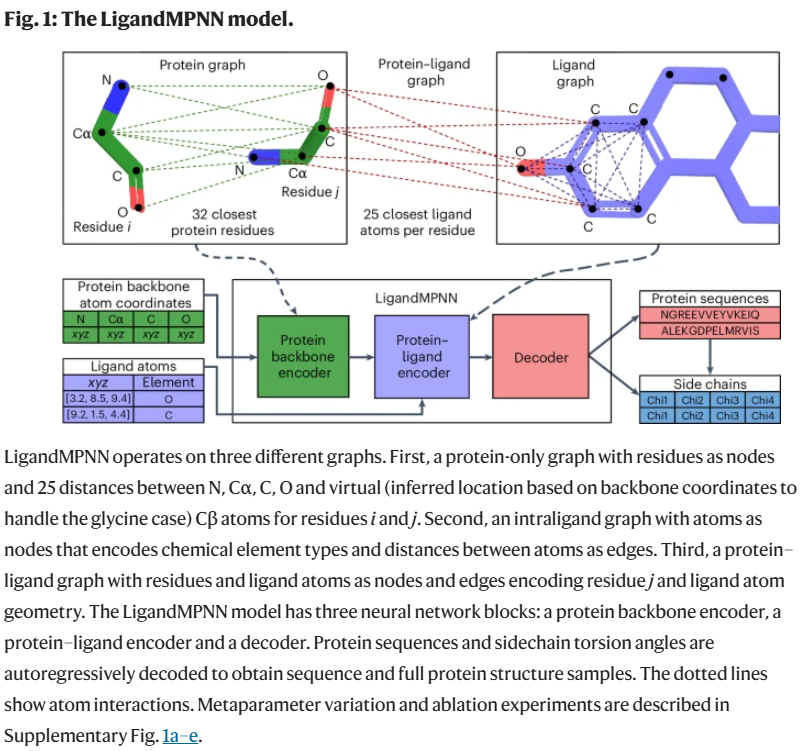
LigandMPNN将蛋白残基作为图中的节点,基于Cα–Cα距离建立蛋白骨架图,再与配体原子构建蛋白–配体图,进行消息传递。最终的图表征通过编码器与解码器协同作用生成目标氨基酸序列。相比ProteinMPNN(166万参数),LigandMPNN模型包含262万参数,计算效率依然较高(单核处理100个残基约0.9秒)。此外,研究人员还基于该框架开发了侧链构象预测网络,用于预测每个残基的四个主构象角(χ1–χ4),提升结合位点评估精度。
LigandMPNN在结构分辨率优于3.5 Å的蛋白复合物上进行训练,测试集中包括含小分子、核酸和金属离子的蛋白结构。在与非蛋白原子5.0 Å内的位点序列恢复任务中,LigandMPNN在小分子(63.3%)、核苷酸(50.5%)和金属(77.5%)背景下均明显优于Rosetta和ProteinMPNN。该方法还具备较强的不确定性评估能力,预测置信度与序列恢复率高度相关。
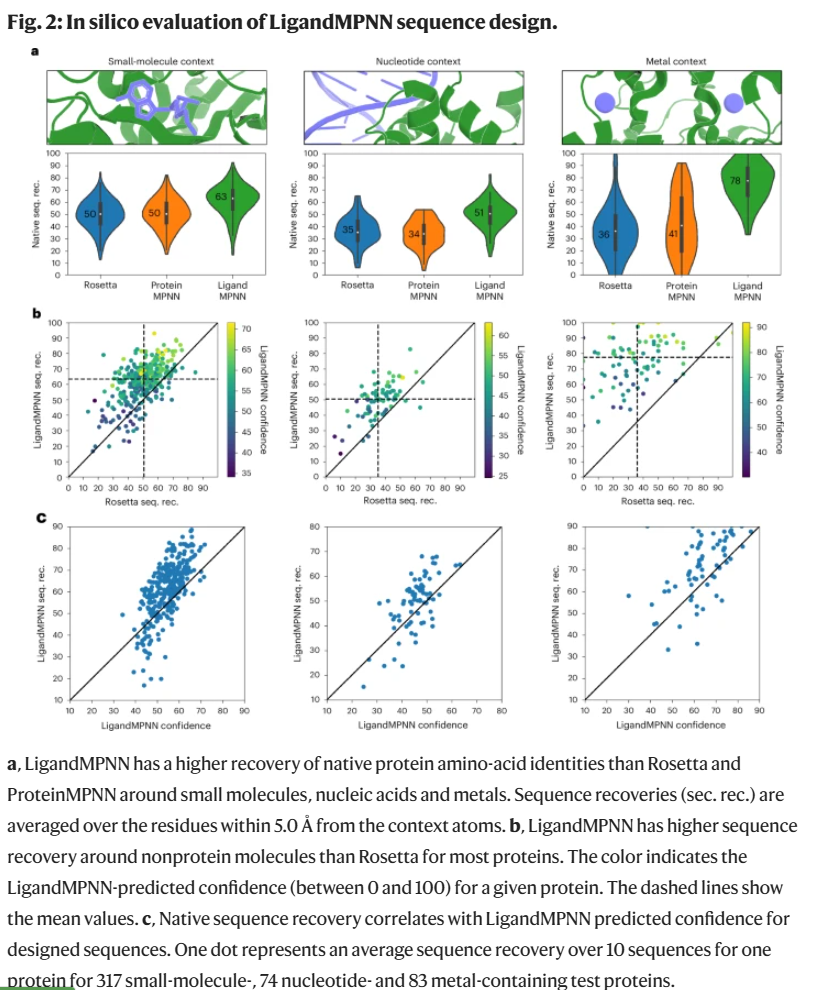
模型性能的提升源于多个关键设计:例如,选择每个残基最近的25个配体原子构建局部图、引入配体元素类型编码等。对模型特征进行消融分析表明,配体信息和化学元素编码对金属结合位点尤其重要。
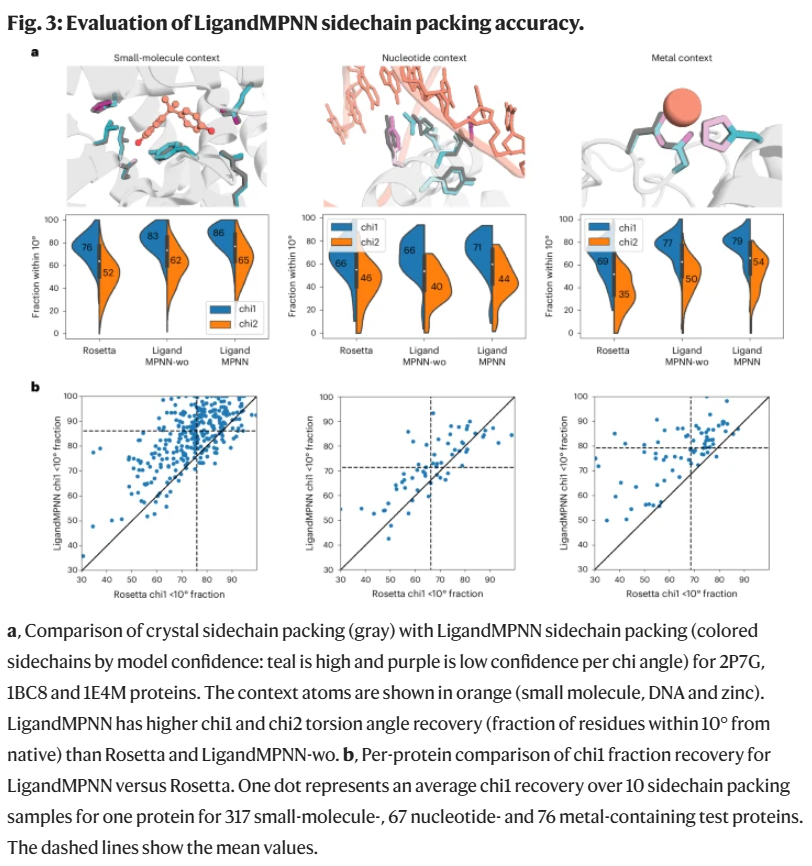
在侧链构象预测方面,LigandMPNN对χ1角的恢复率高于Rosetta和去除配体信息的LigandMPNN-wo模型,并在多个残基(如Gln、Arg、His)上表现出更低的构象偏差,突显其对功能位点精细建模的能力。
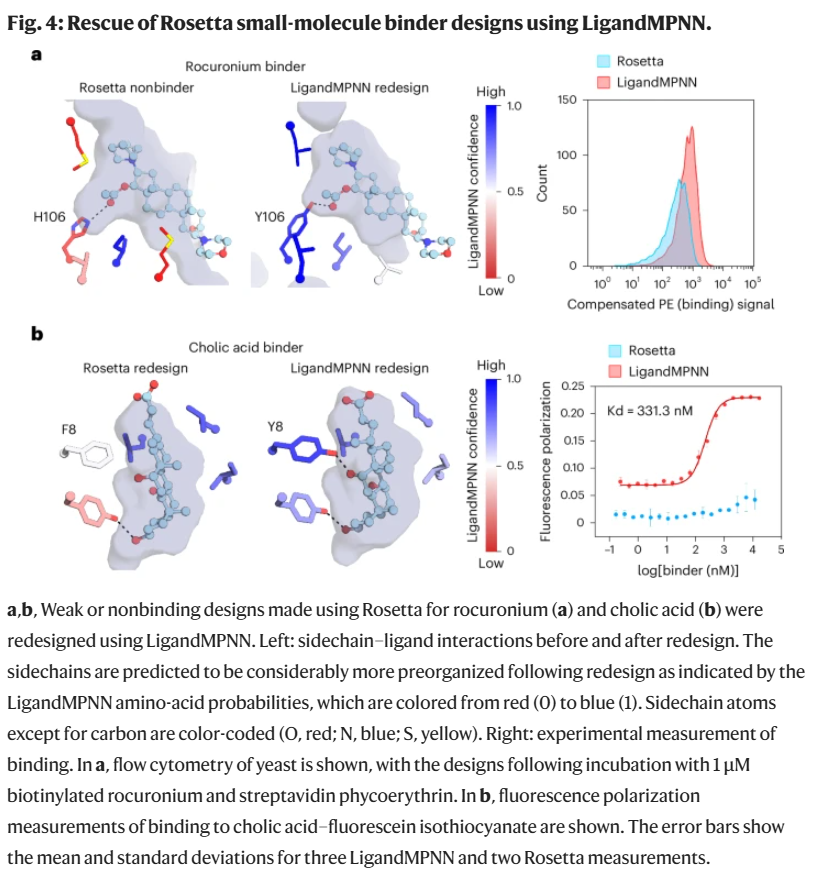
研究人员进一步验证了LigandMPNN在真实配体结合设计中的应用。通过对原本结合能力较弱甚至无结合能力的Rosetta设计进行优化,LigandMPNN显著提升了结合亲和力,其中包括对肌松药物rocuronium和胆酸结合蛋白的成功改造,部分案例的亲和力提升高达100倍。这些结果展示了LigandMPNN的强泛化能力与广泛应用潜力,未来有望在结合蛋白、传感器及酶的设计中发挥重要作用。
讨论
基于深度学习的 LigandMPNN 在设计与非蛋白分子(如小分子、核酸、金属等)相互作用的氨基酸方面,显著优于传统物理模型方法(如Rosetta)。该方法不仅速度快(约为Rosetta的250倍,因绕过了高开销的侧链蒙特卡洛优化),而且在氨基酸类型和构象的恢复准确性上更高。同时,使用门槛更低,无需为新配体手动调整能量函数或力场参数。
研究人员原本担心配体相关训练数据有限会影响性能,但结果表明,对于大多数配体,已有数据量已足够训练出有效模型。不过,面对PDB中极少或未出现过的化学元素时,建议谨慎使用,必要时可将稀有元素映射为常见元素。未来,结合物理模型与深度学习方法或许可在数据稀缺场景下进一步提升序列与构象设计的能力。
LigandMPNN已在多个研究中被成功应用于DNA结合蛋白与小分子结合蛋白的设计,并获得了广泛的实验验证。例如,研究人员使用该方法设计的DNA结合蛋白,其晶体结构高度符合设计模型(PDB: 8TAC);还成功设计了结合17α-羟孕酮、阿哌沙班、SN-38、胆酸、甲氨蝶呤、甲状腺素、洋地黄素、血红素和胆素等配体的蛋白,结合亲和力达纳摩尔级。
截至目前,已有100多个蛋白–DNA或蛋白–小分子结合界面设计被实验验证,其中5个共晶结构高度还原了设计模型,进一步证实了LigandMPNN的可靠性与实用价值。
正如ProteinMPNN在蛋白设计中的广泛应用一样,研究人员预计LigandMPNN也将成为结合蛋白、传感器与酶设计的有力工具。
整理 | WJM
参考资料
Dauparas, J., Lee, G.R., Pecoraro, R. et al. Atomic context-conditioned protein sequence design using LigandMPNN. Nat Methods (2025).
https://doi.org/10.1038/s41592-025-02626-1
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢