DRUGAI
本文介绍了浙江大学药学院侯廷军、谢昌谕、潘培辰和康玉团队发表的一篇论文。该研究提出了一种基于帕累托算法(Pareto)和蒙特卡洛树搜索(Monte Carlo Tree Search)的多目标分子生成方法(PMMG)。PMMG通过帕累托前沿(Pareto Front)评估多个分子属性之间的非支配关系,并在此基础上引入蒙特卡洛策略动态引导分子生成过程,使模型能够探索并趋向多样化的最优解区域。评估结果表明,PMMG在七个目标同时优化的任务中,其生成分子的超支配体积相比基准方法提升了31.4%,成功率提升了2.5倍,展现出在多属性分子设计中实现高效平衡优化的能力。此外,该方法在EGFR/HER2双靶标抑制剂设计场景中也成功发现了性质优越的分子,展示了显著的应用潜力。

研究背景
药物研发中,分子的设计与优化通常被视为一种多目标优化问题,一个理想的潜在成药分子往往需要同时满足多个属性,如生物活性、可合成性、药代动力学性质与安全性等。但这些性质往往彼此冲突而很难同时满足,并且随着目标数量的增加,计算优化的难度呈指数级上升。因此,如何在多个冲突的优化目标间进行权衡,是分子生成领域的一大挑战。
目前,大多数分子生成方法解决多目标优化问题的方式是利用加权求和或乘积转化,将多目标问题简化为单一目标问题。这一策略通常通过设置各属性的权重来表达其重要性,相对简单且易实现,但往往无法实现真正意义上的多目标优化。过度强调某一属性会掩盖其他目标的不足,导致最终分子在整体上达不到平衡最优。此外,这类方法限制了化学空间的探索能力,可能生成无法满足全部属性要求的分子。为突破这一瓶颈,本研究基于帕累托算法和蒙特卡洛树搜索提出了一种新的分子生成方法PMMG,以帕累托最优性(Pareto Optimality)为基础,定义了在多个冲突目标之间的“平衡最优解”,为系统识别潜在最优方案提供了指导。PMMG以SMILES作为分子表示方式,能够高效探索庞大的化学空间,挖掘出同时具备多种理想属性的新型分子。
PMMG架构
PMMG由两个核心模块组成:基于循环神经网络(RNN)的分子生成器,以及用于识别帕累托前沿的蒙特卡洛搜索树(MCTS)。图1展示了PMMG的详细流程图。该方法通过基于帕累托最优的蒙特卡洛搜索树对RNN进行引导,生成满足多重目标性质要求的分子。
具体而言,模型首先通过训练使得RNN模型学习分子的SMILES表示规则,在扩展(Expansion)与模拟(Simulation)阶段通过预测下一个SMILES字符的概率分布来完成分子的逐步生成。随后在整个生成过程中,MCTS通过构建搜索树并迭代执行选择、扩展、模拟和反向传播四个步骤来不断优化搜索方向。该过程中,中间节点的选择基于其上置信界(Upper Confidence Bound, UCB),搜索会持续进行,直至遇到终止符号为止。
与传统方法不同的是,PMMG在反向传播步骤中将奖励函数被重新定义为一个由 n 个维度组成的向量,其中每一维代表一个待优化的目标属性,而非传统的通过加权求和所得到的单一数值。同样在选择步骤中,上置信界(UCB)也被重新定义为一个 n 维向量,由平均奖励值、节点的总访问次数 (n_visits) 和探索系数 (c) 共同构成,用于引导节点选择的方向。此外,过程中针对每一个父节点都定义了一个最优子节点集。在模拟与反向传播过程中,每一个子节点都会被赋予一个 n 维的UCB向量。在进入选择步骤之前,对所有子节点进行两两比较,在UCB的每一个维度上进行判断:若某个子节点在所有维度上均不劣于另一个子节点,则被定义为“非支配节点”。这些节点分别对应生成SMILES字符串中的一个字符(token),以此组成最终分子结构。随后,将所有被支配子节点剔除,仅保留所有非支配子节点,构成最优子节点集,并据此形成非支配解集。所有生成的分子根据其 n 维奖励向量与已有分子进行比较,最终形成包含所有非支配分子的最优解集合。
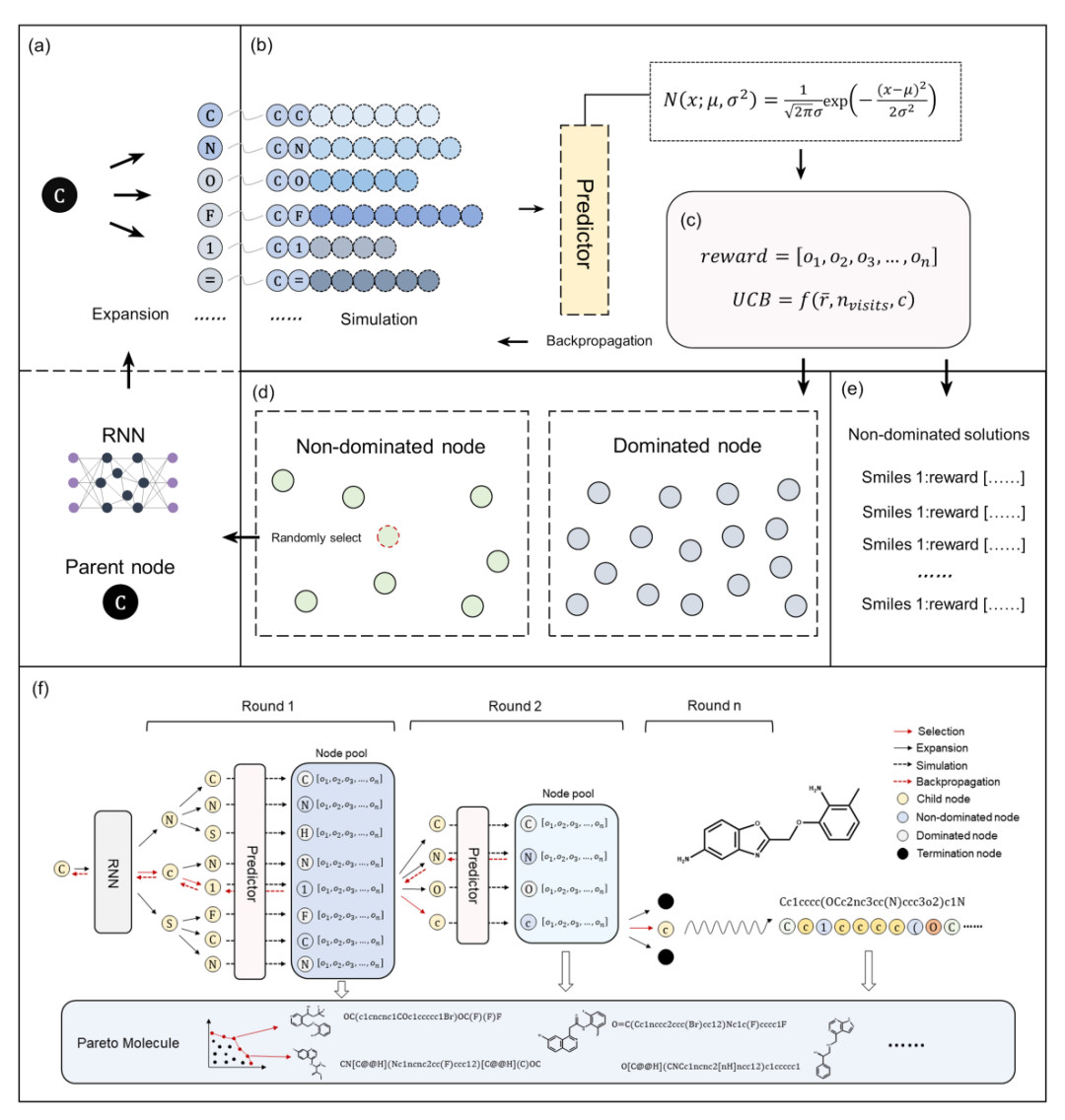
图1. PMMG架构示意图。a)扩展:扩展父节点以获得其对应的子节点;b)模拟:对所有子节点进行模拟,预测其分子属性,并在其中识别所有非支配节点;c)反向传播:根据模拟结果计算每个节点的上置信界,并通过反向传播更新节点状态;d)选择:从非支配节点池中随机选择一个非支配子节点作为新的父节点,其中非支配节点指在至少一个属性上不劣于其他任何节点的节点;e)非支配解:在整个生成过程中持续收集采样得到的分子,剔除被支配分子,得到最终的非支配解集合;f)PMMG的整体工作流程。
实验结果
为了评估PMMG的性能,实验选取了7种SOTA方法与PMMG进行比较,并将其分为两大类:一类为基于SMILES表示的方法,包括基于遗传算法的SMILES-GA、基于强化学习的REINVENT等。另一类为基于图表示的方法,包括基于图神经网络与马尔可夫链蒙特卡洛采样的MARS、图遗传算法Graph-GA等。
在分子生成过程中,实验综合考虑了7个优化目标,涵盖了生物和非生物性质。生物性质包括分子对EGFR的抑制活性;非生物性质则包括水溶性、膜通透性、代谢稳定性、毒性、可合成性以及类药性。并采用三个关键评价指标对比其性能:超支配体积指标(Hypervolume Indicator, HV)、成功率(Success Rate, SR)和多样性(Diversity, Div)。所有优化目标均被归一化至[0,1]区间,以确保实验的一致性与可比性。
PMMG总体性能表现
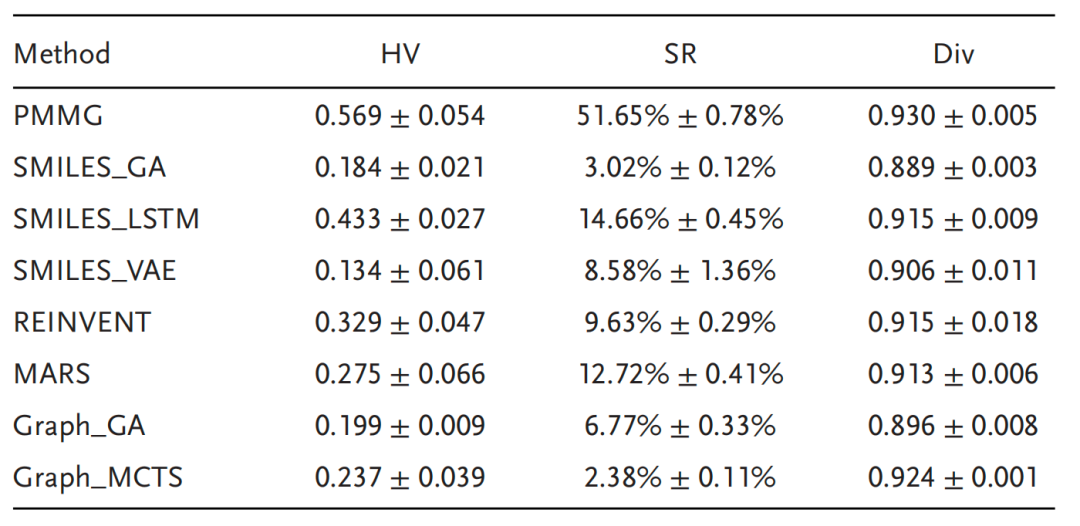
结果如表1所示。结果表明,PMMG在三项指标上均表现优异。在HV方面,所生成的Pareto前沿超支配体积达到0.569,较最优基准方法高出31.4%;在SR方面,PMMG成功生成满足全部优化目标的分子比例达51.65%,远超其他方法,说明其在化学空间中对Pareto前沿的探索能力更为突出。
表1. PMMG与基准方法评估结果。
PMMG单一目标上的性能表现
进一步从单一目标性能维度分析,图2展示了各方法所生成分子在不同属性上的分布情况。可以看到,PMMG在多项指标上均表现出色,尤其是在EGFR抑制活性上优势明显;在溶解度、膜通透性、代谢稳定性等方面与最佳基准方法基本持平;而在毒性和可合成性两个指标上,PMMG生成分子的表现同样亮眼,其中超过90%分子的毒性评分低于3.0,合成评分低于4.0。整体来看,PMMG不仅在多个目标之间实现了高效协同优化,其生成的分子也更多地分布在性能优越的区域。
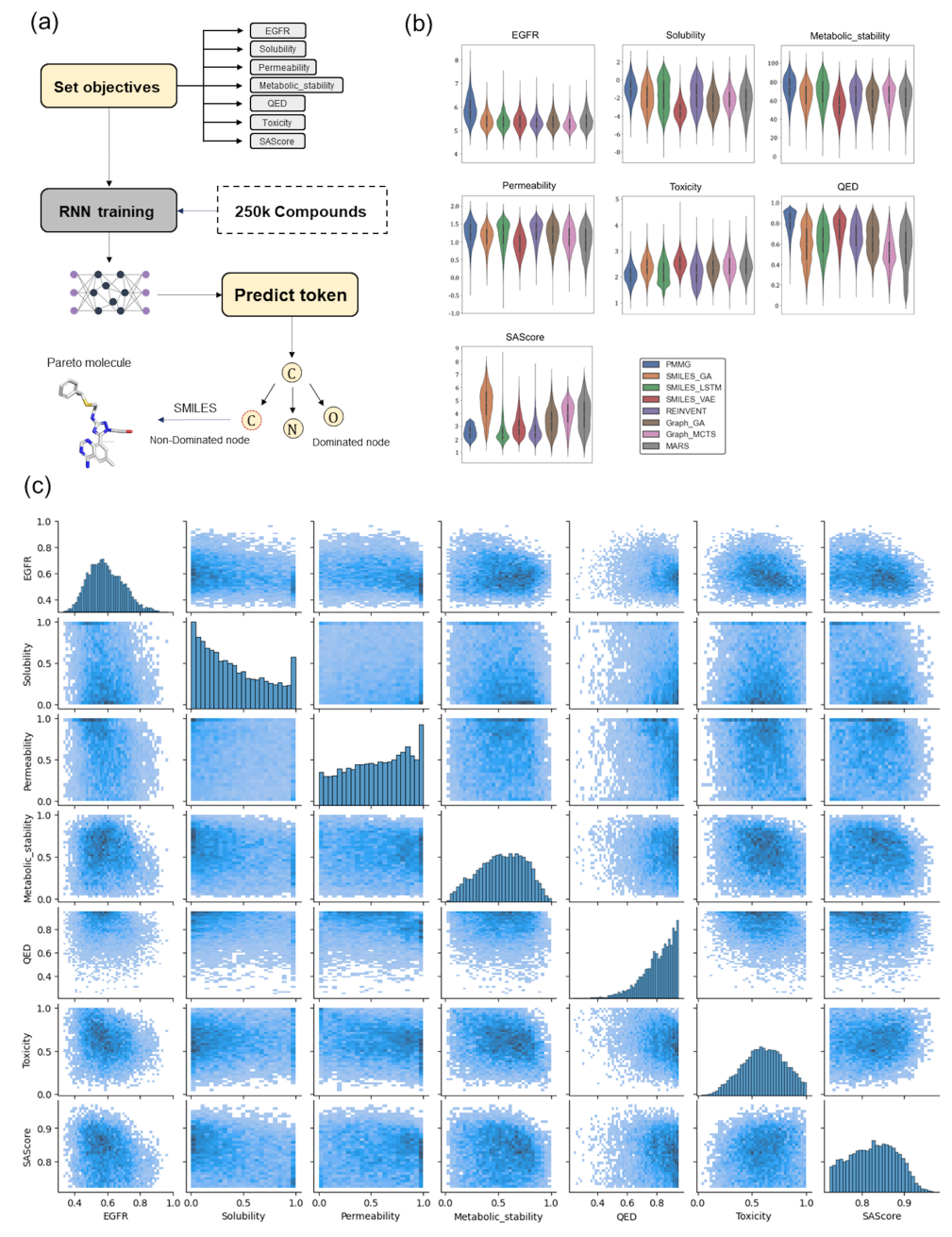
图2. 单一目标优化结果。a) 实验中PMMG的工作流程。b) 每种方法在七个目标上的分子分布琴状图。c) 每对目标之间的散点图。
结构多样性与可合成性分析
为了可视化分子结构的多样性,作者采用了UMAP对生成分子进行分析。如图3所示,不同模型生成的分子分布具有显著相似性。PMMG模型生成的分子能够覆盖其他模型所探索的大部分化学空间,这与表1中呈现的多样性(Div)结果一致。
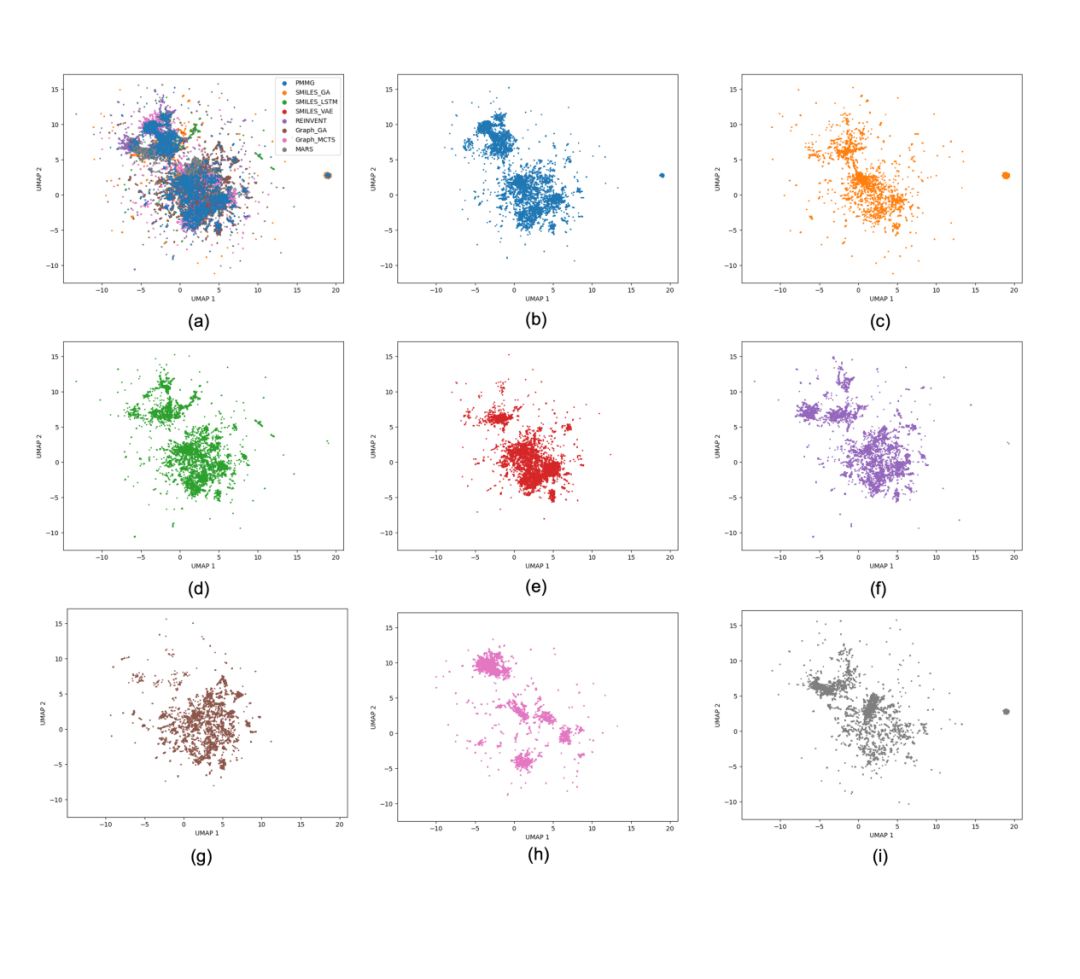
图3. 分子UMAP分布图。
此外,分子的可合成性是评估生成模型实际应用性的重要因素。除了评估目标分子的合成相关属性(如QED和SAScore)外,作者还使用了反合成工具AiZynthFinder进行了更深入的分析。如表2所示,PMMG生成的分子平均可合成性达到了74.33%,进一步突显了PMMG在实际场景中的应用潜力。
表2. 分子可合成性分析结果。

EGFR/HER2双靶点抑制剂的设计
EGFR(表皮生长因子受体)是一类定位于细胞膜表面的受体酪氨酸激酶,在多种癌症中呈现异常高表达,因此成为肿瘤靶向治疗的重要对象之一。HER2(人类表皮生长因子受体2,亦称ERBB2)同属该受体家族,也是一种关键的跨膜蛋白,在乳腺癌等多种肿瘤中频繁出现过度表达。研究表明,EGFR与HER2在某些癌症类型中存在协同作用,共同驱动癌细胞的异常增殖和转移。因此,针对EGFR和HER2的双重抑制策略被认为能够更有效地阻断相关信号通路,从而提高抗肿瘤疗效。目前,Lapatinib是唯一获得FDA批准的EGFR/HER2双重抑制剂,为双靶点药物开发提供了重要参考。
基于这一背景,为进一步验证PMMG在真实药物设计场景中的应用价值,作者开展了以EGFR与HER2为双靶点的分子生成实验,在原有基础上扩展至对8个性质维度的综合优化,其中既包括EGFR和HER2的抑制活性,也涵盖了如LogP、膜通透性、代谢稳定性、毒性、可合成性和类药性等关键药物属性。在分子筛选环节中,采用了Glide对生成分子与两个靶标分别进行分子对接,同时利用ADMETlab 2.0对其ADMET相关性质进行全面预测。根据对接得分和多项性质指标,筛选出一批具备高潜力的候选分子,并进行了详细分析。
图4展示了部分代表性分子,这些分子在EGFR和HER2的对接评分上与Lapatinib表现相当,甚至在某些维度上表现更优;在LogP、膜渗透性、代谢稳定性、可合成性与类药性等方面,也展现出优异的预测性能。这些结果不仅验证了PMMG在多目标优化任务中的强大能力,也凸显了其在双靶点药物早期发现中的应用潜力。
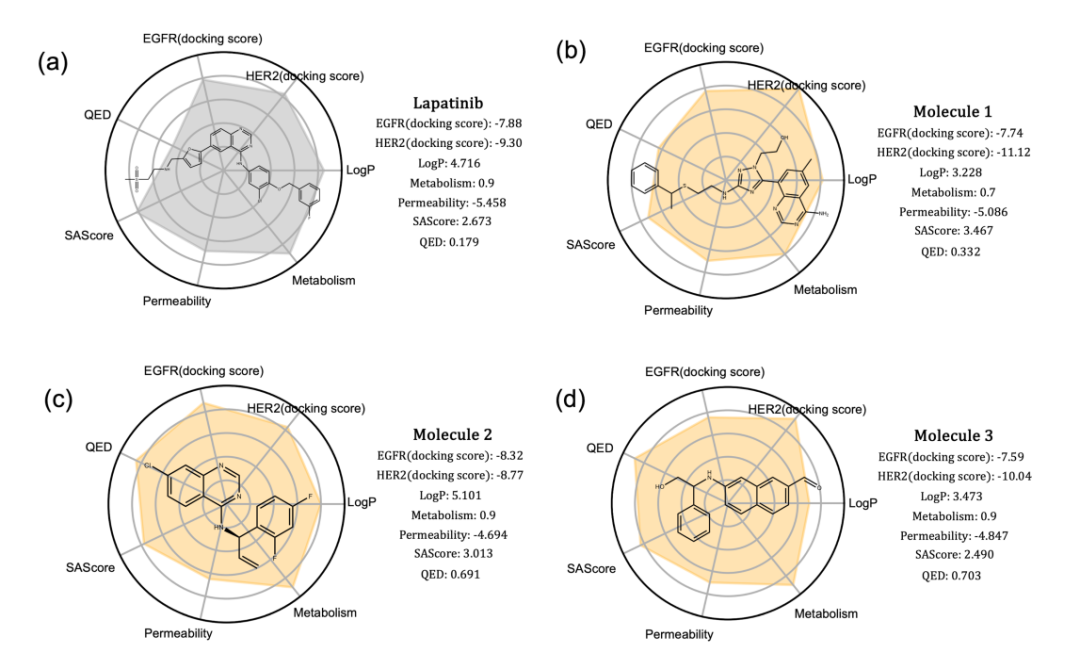
图4. 示例分子及Lapatinib预测性质.
对接构象分析
图5展示了3个分子及Lapatinib与EGFR和HER2的对接构象。对接构象表明,分子1、2和3能够有效地结合到EGFR的活性位点,并通过多个氢键和疏水相互作用稳定结合。分子1的羟基与EGFR中的关键氨基酸ASP863形成强氢键,这可能显著增强其结合亲和力。分子1的芳香环结构与HER2的疏水口袋相互作用,而其氨基与关键残基MET801形成稳定的氢键。分子2和3同样通过芳香环和极性基团与两个靶标形成有效的疏水相互作用和氢键,展示了出色的多靶点结合能力。
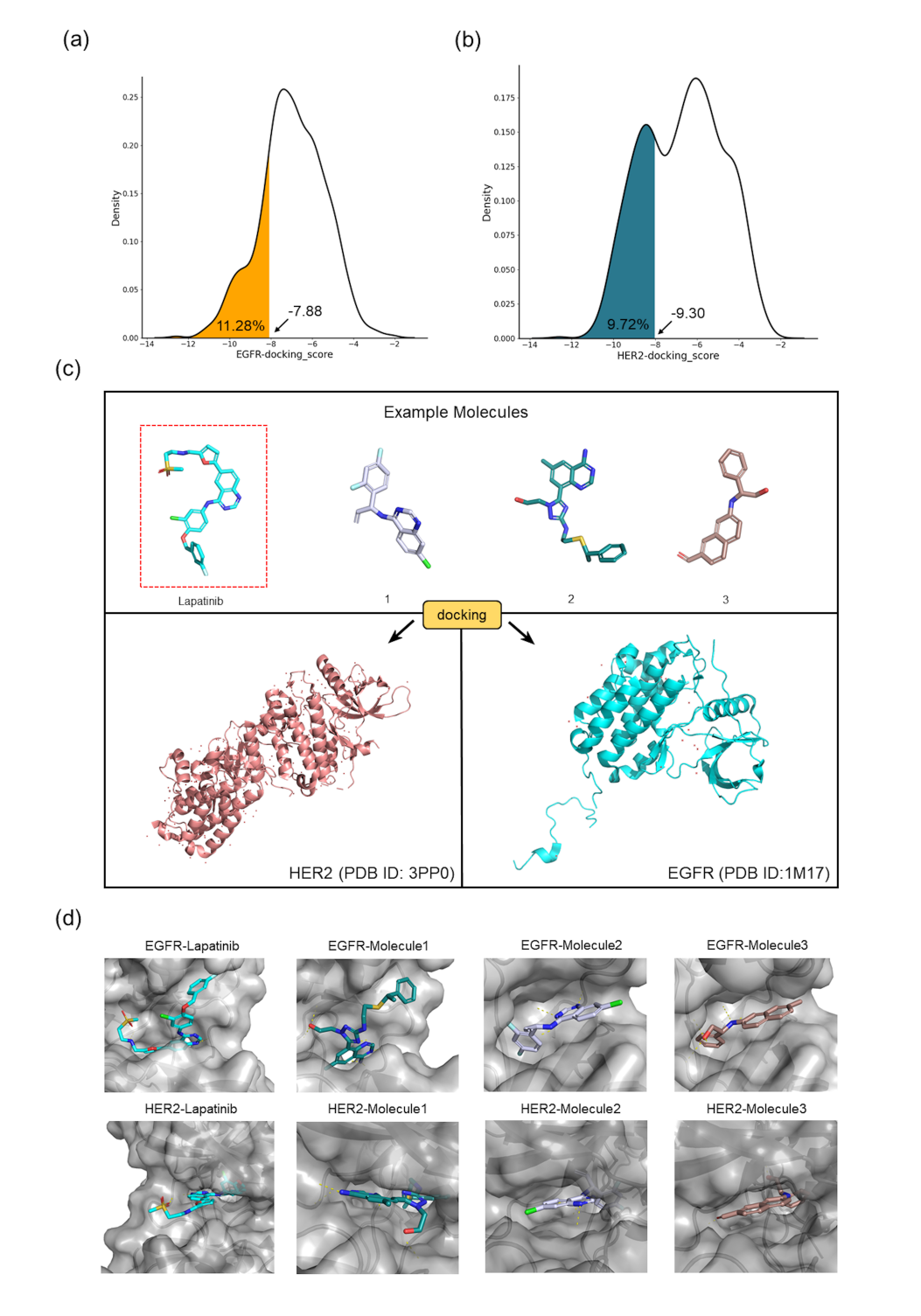
图5. 分子对接结果。a) 分子在EGFR上的对接得分分布。b)分子在HER2上的对接得分分布。c) 示例分子的对接示意。d) 分子1、2、3和Lapatinib与EGFR和HER2的对接构象。
总结
在本研究中,作者提出了一种全新的多目标分子生成框架PMMG。该方法巧妙融合了帕累托优化算法与蒙特卡洛树搜索(MCTS),致力于在分子生成过程中同时优化多个关键的药理和化学属性,包括靶点选择性与结合亲和力、ADMET性质、可合性(SAScore)以及类药性(QED)等核心指标。为全面评估模型性能,作者将PMMG与多种主流分子生成方法进行了系统对比。实验结果表明,在超支配体积(Hypervolume, HV)、成功率(Success Rate, SR)和多样性(Diversity, Div)三大核心指标上,PMMG均展现出卓越表现。特别是在生成同时满足所有优化目标的分子方面,PMMG的成功率高达 51.65%,远超现有方法,充分展示了其强大的多目标优化能力。为了验证PMMG的实际应用潜力,作者进一步将其应用于EGFR/HER2双靶点药物设计任务,成功生成了一批候选分子,在对接打分和性质预测上可与已上市药物Lapatinib相媲美,充分展示了模型的实际应用和有效性。
尽管PMMG仍有优化空间,但其在当前阶段已在多目标分子生成领域取得了突破性进展。通过在多种药物属性之间实现高效权衡与协同优化,PMMG为应对药物设计中固有的复杂性与目标冲突问题提供了有力工具。随着未来更先进的机器学习技术融入,以及化学空间探索能力的持续拓展,PMMG有望进一步推动智能化药物设计的发展边界。
参考资料
Liu Y, Zhu Y, Wang J, Hu R, Shen C, Qu W, Wang G, Su Q, Zhu Y, Kang Y, Pan P, Hsieh CY, Hou T. A Multi-Objective Molecular Generation Method Based on Pareto Algorithm and Monte Carlo Tree Search. Advanced Science.
https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202410640
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢