DRUGAI
今天为大家介绍的是来自耶鲁大学Gregory W.Kyro团队的一篇论文。深度学习已经改变了蛋白质设计领域,实现了精确的结构预测、序列优化和从头开始的蛋白质生成。通过AlphaFold2、RoseTTAFold、ESMFold等在单链蛋白质结构预测方面的进展已经达到接近实验精度的水平,这启发了后续工作通过AlphaFold Multimer、RoseTTAFold All-Atom、AlphaFold 3、Chai-1、Boltz-1等扩展到生物分子复合物。生成模型如ProtGPT2、ProteinMPNN和RFdiffusion已经突破了基于自然进化的限制,实现了序列和主链设计。最近,包括ESM3在内的序列-结构联合共同设计模型,将这两种模式整合到统一框架中,从而提高了可设计性。尽管取得了这些进展,但在建模序列-结构-功能关系以及确保训练数据所涵盖的蛋白质空间区域之外的稳健泛化方面仍然存在挑战。未来的进展可能会集中在序列-结构-功能共同设计框架上,这些框架能够比独立处理这些模式的模型更有效地建模适应度景观。目前的能力,加上令人眩晕的进步速度,表明该领域即将实现快速、合理地设计具有定制结构和功能的蛋白质,超越自然进化所施加的限制。

在这篇综述中,作者讨论了深度学习方法在蛋白质设计中的当前能力,重点关注一些最具革命性和能力的模型(表1),涉及它们的功能性和它们所实现的应用,直至当前该领域面临的挑战和最佳前进路径。由于篇幅限制,本推文仅展示综述中的部分内容。
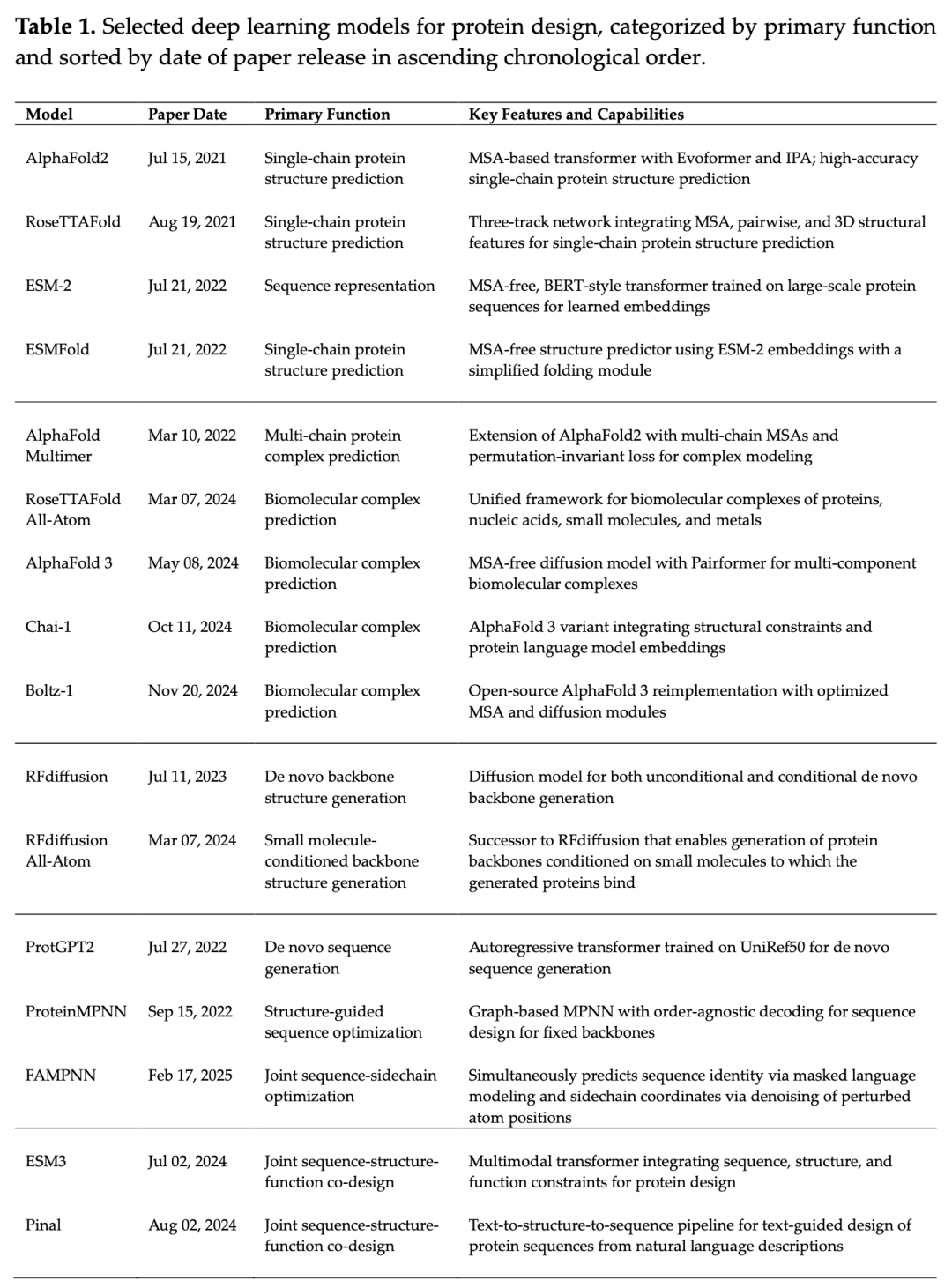
表 1
蛋白设计的生物意义
蛋白质从根本上定义了细胞功能,它们的结构和动态特性决定了它们在催化、分子识别和调控等重要生化事件中的作用。因此,设计具有特定功能特性的蛋白质能够直接调节重要的生化过程。
已经有酶(即通过稳定过渡态来催化化学反应的蛋白质)的例子,这些酶已被优化以提高底物特异性、增强反应动力学和改善在不同条件下的稳定性。此外,细胞因子(即由免疫细胞分泌并与靶细胞上的特定受体结合以调节其行为的小蛋白质)和生长因子(即与特定细胞表面受体结合以刺激细胞生长、增殖、分化和愈合的蛋白质)已被设计成具有增强的特异性,从而在免疫调节、再生医学和肿瘤学中改善治疗效果。
抗体已被设计成具有高亲和力结合,并改善了对靶向抑制与疾病相关蛋白质的选择性。除了抗体外,还有多种合成蛋白质结合物的实例,这些结合物被设计成以高亲和力与蛋白质靶标的表面结合,以抑制蛋白质-蛋白质相互作用,诱导激活或抑制功能的构象变化,通过阻断天然配体来调节信号传导,以及招募降解机制以消除致病蛋白质。这些例子,连同许多其他例子,展示了理性蛋白质设计如何使作者能够直接调节关键细胞功能并提供重要的治疗解决方案。
蛋白质设计的目标
蛋白质设计的目标通常可以分为通过靶向修饰改进现有蛋白质的功能,或构建全新蛋白质(即从头设计,de novo design)。对天然蛋白质的靶向修饰可以提高它们的稳定性、特异性或催化效率,使其在不同环境条件下表现更佳。例如,向酶中引入靶向突变可以优化底物结合或扩大其操作范围,使其在非原生条件下发挥功能。同样,改变现有蛋白质的结合界面可以扩大其特异性,使其能够与新靶标结合或破坏以前无法接触的蛋白质-蛋白质相互作用。
除了优化天然蛋白质以提高功能外,理性工程努力(rational engineering efforts)可以将它们重新用于全新功能;最初为一种生物学角色进化的蛋白质可以通过结构适应被重新编程以执行不同的功能。这种方法已成功应用于酶工程,其中催化域被重新设计以处理新型底物或介导替代反应机制。同样,蛋白质结合物可以重新配置以靶向不同的配体,从而在药物发现和合成生物学中得到应用。
从头蛋白质设计提供了一个更具一般性的框架,能够实现需要自然界中不存在功能的应用。这种策略通常包括识别支持预期功能的结构基序,构建能够准确呈现这些基序的稳定主链结构,以及选择能够折叠成这种结构的氨基酸序列。这种方法已经使具有新型折叠方式、精确分子识别特性和高度稳定组装体的蛋白质的开发成为可能。此外,从头设计策略在设计用于蛋白质-蛋白质相互作用和小分子识别的蛋白质方面已经证明是成功的。在许多情况下,从头设计比改造现有蛋白质结构以实现预期功能更有效;从头设计提供了对蛋白质结构和序列的完全控制,而天然蛋白质往往功能和稳定性都处于临界状态。
蛋白设计中的深度学习
蛋白质设计从根本上受到可能的氨基酸序列的巨大组合空间的限制,以及序列和功能之间的复杂、非线性关系的制约。适应度景观将这种关系概念化,定义了序列变异如何对应功能特性。即使对于中等长度(100个氨基酸)的蛋白质,搜索空间也有20^100(约1.27×10^130)个可能的序列,暴力探索是不可行的,这就需要能够有效地模拟和导航这个景观的方法。
深度学习通过实现能够建模序列-结构-功能关系的数据驱动框架,从根本上重塑了蛋白质设计。通过一系列多样化的深度学习方法,已经实现了以高精度从序列预测蛋白质结构、生成为稳定性和功能而优化的新序列,以及设计全新的蛋白质主链的能力。然而,目前还不存在一个能够有效解决蛋白质设计问题所有方面的单一框架。相反,专门的架构已经出现,每种架构都解决不同的挑战。
蛋白质结构预测仍然是计算生物学的基石,因为蛋白质的功能在很大程度上由其三维构型决定。深度学习模型如AlphaFold2、RoseTTAFold和ESMFold在从氨基酸序列预测单链蛋白质结构方面展示了非凡的精确度。这些进展,连同其他的进展,启发了能够预测多组分生物分子复合物结构的模型的发展,例如AlphaFold Multimer、RoseTTAFold All-Atom、AlphaFold 3、Chai-1和Boltz-1。
除了结构预测,序列设计的逆任务(即确定可靠地折叠成目标结构的氨基酸序列)为蛋白质工程提供了另一项极其有价值的能力。然而,传统的基于能量的优化方法难以有效地探索序列空间,因此激发了基于学习的替代方法的发展。ProtGPT2通过建模约5000万个蛋白质序列的统计分布来解决从头序列生成问题,捕捉自然界中观察到的序列级模式,但缺乏明确的结构约束。ProteinMPNN通过基于图的消息传递框架学习序列-结构关系,引入了结构引导设计,与以前的方法相比显著提高了序列恢复率。最近,BindCraft将AlphaFold2梯度整合到端到端可微分优化管道中,实现了序列的直接优化,以实现与目标蛋白质的高亲和力结合。
深度学习还显著增强了作者生成新型蛋白质主链的能力。传统方法依赖于基于片段的组装和能量最小化,这对结构多样性施加了限制。RFdiffusion引入了基于深度学习的替代方法,它采用去噪扩散概率模型(DDPM)从噪声中迭代构建蛋白质主链,实现不受现有模板限制的从头折叠生成。这种方法促进了具有新型拓扑结构和功能性支架的蛋白质设计。随后的方法如RFdiffusion All-Atom扩展了这种方法,以生成预测能够与预先指定的小分子结合的蛋白质主链。
最近,有一些进展旨在在统一的生成框架内联合建模序列和结构。ESM3引入了一个基于多模态transformer的框架,联合建模序列、结构和功能,使设计的序列具有隐式的结构和功能偏好。Pinal通过加入自然语言输入作为额外约束调整了这个概念,使得可以通过基于文本的提示来指定功能特性。
随着新模型和方法的发展,现有范式将被完善、扩展或替代。以下章节将按时间顺序检视一些塑造现代蛋白质设计的当前最先进模型,以反映该领域的演变。
蛋白质结构预测
从氨基酸序列预测蛋白质结构长期以来一直是计算生物学中的核心挑战,因为序列和三维构型之间存在复杂的关系。准确的结构预测提供了对蛋白质功能、稳定性和相互作用的见解,使药物发现、蛋白质工程和合成生物学应用成为可能。传统方法严重依赖同源建模和基于物理的模拟,虽然在某些情况下有效,但受到同源结构的可用性和计算复杂性的限制。深度学习的最新进展已经彻底改变了这一领域,使模型能够以前所未有的精度和规模捕捉序列-结构关系。在这些进展中,AlphaFold2标志着一个变革性的飞跃,在直接从序列预测单链蛋白质结构方面达到了接近实验的精度。
AlphaFold2通过整合多序列比对(MSAs;进化相关序列的结构化比对,可揭示保守残基和共进化模式)、结构模板、称为Evoformer的迭代注意力网络架构以及利用不变点注意力(IPA)机制来模拟空间关系的结构模块,能够以高精度从序列预测蛋白质三维结构。
AlphaFold2从UniRef90、BFD、Uniclust30和MGnify数据库获取同源序列并编译成MSAs。在可用的情况下,蛋白质数据库(PDB)的结构模板会与查询序列对齐,提供显式的空间先验来指导模型。
对于给定序列中的每对残基,AlphaFold2通过整合相对位置嵌入与MSA衍生信息和结构模板(如果可用)来初始化成对特征。位置嵌入对序列中两个残基之间的分箱距离进行编码,使模型能够捕捉基于序列接近度的依赖关系。MSA衍生的共进化信息使用外积平均操作提取(即,对于每对残基,在MSA中所有同源序列上计算其嵌入向量的外积,然后在MSA上平均以形成单一共进化矩阵)。在可用时,模板衍生的结构先验从对齐的PDB结构提供明确的成对距离和方向,提供空间约束,这对MSA覆盖稀疏的区域特别有价值。
ESMFold利用ESM-2直接从ESM-2派生的序列嵌入预测3D蛋白质结构,绕过了对MSAs的需求。此外,ESMFold完全省略了模板信息,而是使用ESM-2派生的注意力图,这些图已被证明能隐式捕获结构约束。与传统的依赖MSA和模板的模型相比,这些调整使推理速度显著提高并且更具可扩展性。
与由Evoformer和结构模块组成的AlphaFold2架构(第4.1节)相比,ESMFold用一个称为折叠块(Folding Block)的更简单的替代方案替换了Evoformer,同时保留了相同的结构模块。由于ESM-2派生的特征是一维的,而不是二维MSAs,折叠块在一维特征空间上执行标准的自注意力,而不是AlphaFold2的Evoformer中使用的轴向注意力。折叠块的其余操作镜像了Evoformer的操作,使ESMFold能够保持结构预测性能,同时提高计算效率。
多链和复杂组装结构预测
生物功能通常来源于生物分子复合物或大分子组装体,这些结构涉及蛋白质、核酸和小分子。准确预测这些复合物的结构,无论是蛋白质-蛋白质、蛋白质-配体,还是蛋白质-核酸复合物,都需要能够捕获分子间进化信号、界面动态以及每种复合物特有的结构约束的建模方法。
像AlphaFold2这样的单链模型在预测单个蛋白质结构方面表现出色,但缺乏对生物分子复合物进行建模的能力。为了解决这一限制,已经开发了新的架构来高精度预测多组分生物分子结构。
AlphaFold 3扩展了AlphaFold2(第4.1节)的能力,通过引入架构修改,使其能够预测基于蛋白质的多组分生物分子复合物结构。与AlphaFold2相比,AlphaFold 3用Pairformer网络替换了Evoformer,该网络保留了序列和配对表示,但移除了MSA表示。这种策略使模型即使在同源序列有限的情况下也能准确预测结构。
此外,AlphaFold2中的确定性结构模块被基于扩散的生成模型所取代,该模型以噪声扰动的原子坐标作为输入,并通过多个去噪步骤迭代地改进它们以预测最终的3D结构。SE(3)等变性通过在训练过程中对输入坐标应用随机旋转和平移来实现,确保预测结果对全局方向和位置保持不变。AlphaFold 3架构中基于扩散的组件由30个顺序注意力块组成;三个局部注意力块捕获原子级相互作用,接着是24个建模长程残基-残基相互作用的全局注意力块,最后以三个在产生最终输出前优化原子位置的局部注意力块结束。
为了实现准确的置信度估计,AlphaFold 3在训练期间采用扩散展开。尽管是在单步去噪而非完整的端到端生成上训练的,但在扩散展开过程中,模型通过从噪声扰动结构开始的20个迭代去噪步骤模拟完整的扩散过程。然后将得到的预测结构与真实结构进行比较,得出用于训练置信度头以预测每个残基置信度分数的性能指标。
AlphaFold 3预测多样化生物分子复合物和大分子组装体结构的能力显著推进了结构生物学中的深度学习,启发后续模型效仿其方法。
Boltz-1是AlphaFold 3的开源版本,对架构进行了有针对性的改进。具体来说,该模型修改了MSA模块(即处理并将配对表示传递给Pairformer的模块)中的操作流程,以改善单一表示和配对表示之间的相互作用。此外,Boltz-1在扩散模块的注意力层中引入了残差连接并重新排序操作,以改进梯度流、反向传播效率和层内信息传递。Boltz-1以MIT许可证发布,为研究社区提供了更大的可访问性,促进了进一步的开发和适应。
从头结构生成
从头蛋白质结构生成旨在独立于进化约束设计全新的蛋白质。传统方法依赖于基于片段的组装和基于物理的能量最小化,但这些方法对设计灵活性和结构多样性施加了显著的限制。深度生成模型,特别是去噪扩散概率模型(DDPMs),通过直接学习结构分布并迭代地将随机初始化精炼成稳定的蛋白质折叠,实现了从头主链生成。这种能力使得创建超越自然界现有范围的新型拓扑结构和功能性支架成为可能。
RFdiffusion是通过微调RoseTTAFold(RF)开发的一种用于从头蛋白质主链设计的DDPM。该模型遵循标准的前向(加噪)-反向(去噪)扩散框架。在前向过程中,源自PDB的蛋白质结构在200个步骤中逐渐受到扰动,通过在Cα坐标上添加高斯噪声进行平移,并在旋转矩阵流形上使用布朗运动来改变残基方向。在反向过程中,神经网络通过从噪声坐标和方向预测每个时间步施加的噪声,学习迭代地对蛋白质主链进行去噪。为确保去噪时间步之间的轨迹连贯性,RFdiffusion在每个时间步上对前一时间步预测进行自我条件化噪声预测。在推理过程中,模型从完全随机的残基框架开始,最终生成蛋白质主链结构。
除了从噪声无条件生成蛋白质主链外,RFdiffusion还能够进行有条件生成,明确控制设计约束,包括功能基序(例如,酶活性位点、病毒表位)支架构建、拓扑结构(例如,TIM桶)条件设计,以及对蛋白质结合剂设计的热点残基进行条件控制。为了实现蛋白质结合剂设计,RFdiffusion在来自PDB的多链蛋白质和蛋白质-蛋白质复合物上进行微调,学习在给定目标蛋白质的热点残基的情况下生成结合蛋白。在这些情况下,RFdiffusion都展示了显著的成功案例。
随后,通过微调RoseTTAFold All-Atom(第5.2节)开发了RFdiffusion All-Atom(RFdiffusionAA),并扩展了RFdiffusion的能力,可以生成以指定小分子为条件的新型蛋白质主链结构,使生成的蛋白质能够与这些小分子结合。
从头序列生成和优化
生成能够可靠折叠成功能性结构的新型蛋白质序列是蛋白质设计中的一个基本挑战。传统方法依赖于基于能量的模型和启发式搜索方法,这些方法通常计算成本高昂,在探索广阔的序列空间方面能力有限。深度学习的最新进展引入了能够从头设计蛋白质序列的生成模型,这些序列超越了通过自然选择产生的序列空间,并能优化序列以获得所需特性(如增强的稳定性、表达、结合亲和力和/或催化活性)。
ProteinMPNN是一种基于图的消息传递神经网络(MPNN),用于蛋白质序列设计,包含一个主链结构编码器和序列解码器,用于预测能折叠成特定主链结构的氨基酸序列。与Rosetta等基于能量的方法不同,Rosetta依赖计算密集的蒙特卡洛优化来为固定主链设计低能量序列,而ProteinMPNN使用学习到的序列-结构关系预测最佳序列,从而在序列恢复率(52.4%,相比Rosetta的32.9%)和设计准确性方面实现显著改进。
该模型将蛋白质主链结构表示为图,其中节点对应于Cα原子,边存在于每个节点与其在3D空间中由欧几里得距离确定的30个最近邻之间。边编码了成对的几何特征,包括残基间的Cα-Cα距离、N、Cα、C、O和虚拟Cβ原子之间的残基内成对距离、主链二面角和局部框架方向。这个图由编码器处理,编码器是一个同时更新节点和边的MPNN。然后,编码的特征由一个与顺序无关的自回归解码器处理,该解码器预测序列中每个位置上氨基酸的概率分布。通过随机化解码顺序实现与顺序无关的解码。
ProteinMPNN在来自PDB的19,700个高分辨率单链结构上进行训练。模型在训练过程中的目标是在给定蛋白质主链结构作为输入的情况下,最小化预测序列与天然序列之间的每个残基交叉熵损失。为了增强泛化能力,特别是对预测结构的泛化,模型在训练期间用高斯噪声(标准偏差为0.02 Å)扰动主链坐标。
ProteinMPNN成功应用于挽救失败的Rosetta设计的蛋白质结合剂,并产生了通过X射线晶体学和冷冻电镜验证的稳定、可表达的蛋白质。
尽管ProteinMPNN最初是为固定主链序列设计而开发的,但它被重新用于优化蛋白质序列以改善表达、稳定性和功能。这是通过以下方式实现的:固定对功能至关重要的残基(例如,配体结合位点),利用进化保守性来维持结构或功能上重要的位置,使用ProteinMPNN生成预测能够折叠成特定主链结构的序列变体,并使用AlphaFold2过滤设计的序列以确保高置信度的折叠和与输入主链结构的结构一致性。这种方法成功应用于肌红蛋白,以增强其在高温下的稳定性和功能,以及应用于TEV蛋白酶以增强其稳定性和催化效率。
编译|黄海涛
审稿|王梓旭
参考资料
Kyro, G. W., Qiu, T., & Batista, V. S. (2025). A Model-Centric Review of Deep Learning for Protein Design. arXiv preprint arXiv:2502.19173.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢