DRUGAI
今天为大家介绍的是来自清华大学张学工教授所带领的团队发表的一篇论文。细胞调控是一个复杂的系统,涉及从单个基因的调控到多个基因之间的相互作用等多个层面。虽然目前已有一些神经网络模型可以将分子变化与细胞特征联系起来,但这些模型在设计上缺乏对调控机制的考虑,难以解释细胞状态转换等关键生命活动背后的调控机制。为解决这一问题,研究团队开发了一个名为regX(Regulatory Network)的深度神经网络。这个模型的特点是同时考虑了基因层面的调控和基因之间的相互作用机制,能够找出推动细胞状态改变的关键调控因子,并对其作用机制进行解释。研究人员将regX应用于2型糖尿病和毛囊发育的单细胞多组学数据分析中。结果表明,该模型能够准确识别出影响细胞状态变化的关键转录因子和重要的DNA调控区域。更重要的是,研究发现的一些调控因子为疾病治疗提供了新的靶点,同时也为已有药物的新用途开发和致病基因变异(单核苷酸多态性)的鉴定提供了线索。这项研究展示了如何通过设计可解释的神经网络模型,来更好地理解和揭示生物系统的运作机制。

细胞是一个复杂的调控系统,包含多个层次的调控过程。在单个基因层面,有表观遗传调控(Epigenetic Regulation)、转录调控(Transcriptional Regulation)和翻译调控(Translational Regulation);在更高层面,还存在基因之间的相互作用和信号通路。开发能够模拟这种多层次细胞系统的计算方法,对于深入理解细胞功能的分子机制、细胞命运决定以及细胞调控的可能性具有重要意义。近年来,多个研究团队开发了具有生物学意义的神经网络模型,用于研究多层次生物系统。这些模型在科学发现方面取得了显著成果。然而,现有模型都忽略了一个重要方面:它们没有考虑DNA包装结构变化(染色质重塑)和蛋白质与DNA的结合(转录因子结合)等基础调控过程,而这些过程对于细胞发育和疾病进展过程中的变化至关重要。
为此,研究团队开发了regX(Regulatory Network)深度神经网络模型。这个模型的独特之处在于它同时考虑了基因层面的调控和基因之间的相互作用机制。研究人员将该模型应用于两个研究案例:2型糖尿病的疾病进展和毛囊发育的过程,分析它们的单细胞多组学数据。研究结果显示,regX能够可靠地识别出关键的转录因子(TF)和重要的DNA调控区域(cCRE)。特别是在2型糖尿病研究中,模型发现的一些重要调控因子不仅提供了新的治疗靶点和药物开发思路,还帮助发现了一些可能导致疾病的基因变异位点。这些发现为解读全基因组关联研究(GWAS)的结果提供了新的视角,这也是当前基因组研究领域的一个重要课题。此外,在毛囊发育研究中也获得了一些有价值的生物学发现。
模型架构
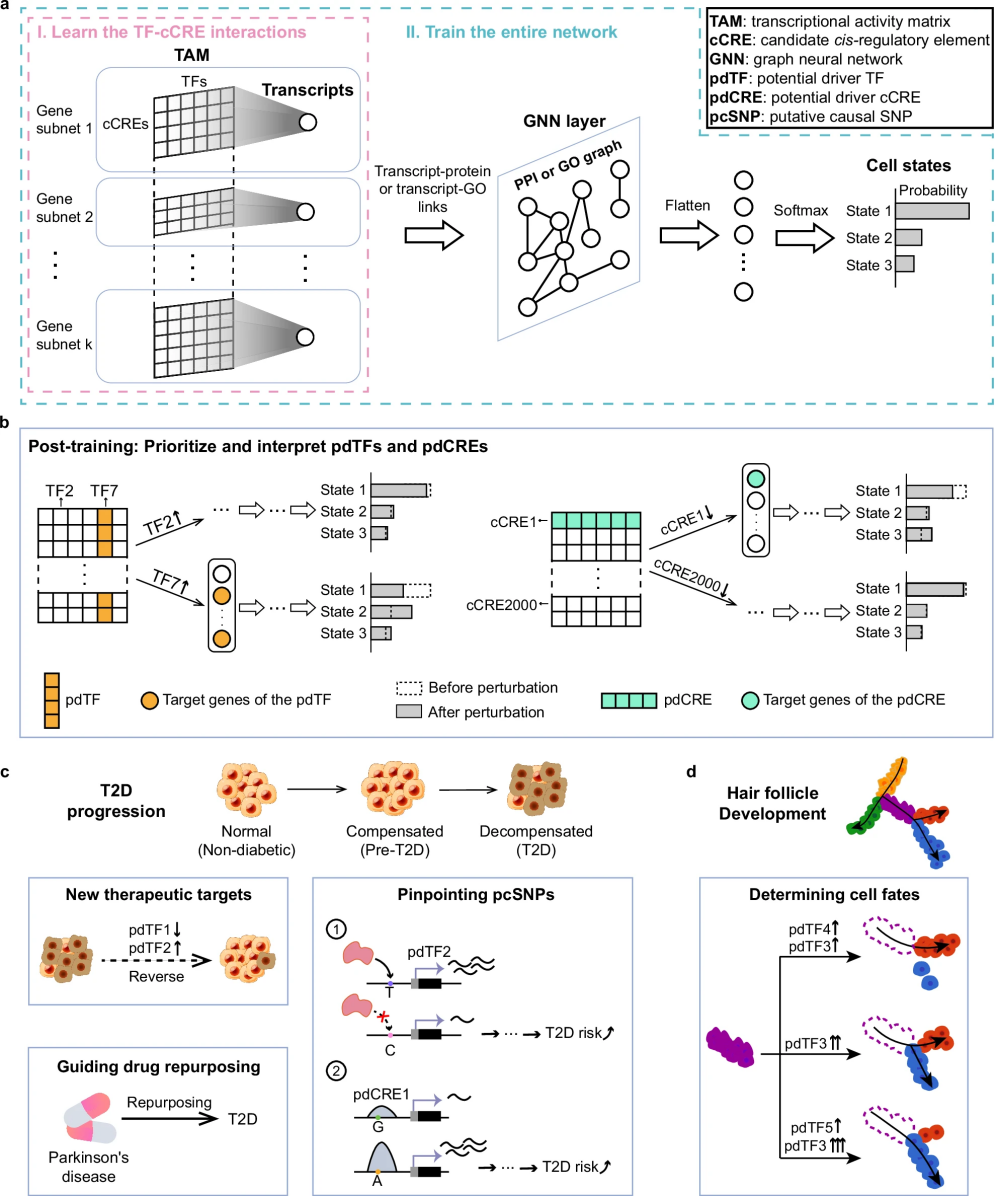
图 1
研究团队设计了一个基于生物学机制的神经网络,用于预测细胞状态。如图1a所示,该网络通过分析DNA调控区域(cCRE)和转录因子(TF)的数据来实现预测。在网络的隐藏层中,研究人员嵌入了目标基因、蛋白质和功能信息,作为调控细胞状态的重要途径。
为了模拟基因表达过程,研究团队创新性地开发了一个可学习的转录活性矩阵(Transcriptional Activity Matrix, TAM)。这个矩阵通过计算转录因子表达量、DNA可及性区域的开放程度,以及从数据中学习得到的转录因子-DNA相互作用三者的乘积,来描述转录因子如何通过与基因周围的开放染色质区域相互作用来调控基因表达。为了模拟基因间的相互作用,研究人员使用了图神经网络(Graph Neural Network, GNN)来整合蛋白质之间的相互作用网络或基因功能分类(Gene Ontology, GO)之间的关系。这种设计理念认为,蛋白质相互作用网络和基因功能分类图谱都是从不同角度对基因间真实关系的合理抽象。
如图1b所示,研究人员通过计算机模拟扰动(in-silico perturbation)的方式,来识别在细胞状态转换过程中起关键作用的转录因子和DNA调控区域。具体方法是比较在给定扰动前后,细胞状态概率的变化程度。得益于基于机制的设计,研究人员还能够通过分析目标基因表达水平的变化,来确定这些关键调控因子的作用靶点。
如图1c和图1d所示,研究团队将regX应用于两个单细胞多组学数据集的分析。第一个是使用10x multiome技术对人类胰腺β细胞进行测序的2型糖尿病数据集,样本来自非糖尿病、糖尿病前期和糖尿病患者。第二个是使用SHARE-seq技术对小鼠毛囊发育过程中的皮肤细胞进行测序的数据集。考虑到人类数据在后续分析中有更丰富的资源可用,研究重点使用2型糖尿病数据集来展示regX在识别关键调控因子方面的特点和能力,以及这些发现的机制和治疗意义。而毛囊发育数据集则用于展示regX在结构上的灵活性和通用性,以及其在量化调控因子对细胞命运决定影响方面的优势。
数据驱动的转录调控模型捕获了TF-cCRE的相互作用
研究团队创新性地设计了转录活性矩阵(TAM),这是整个神经网络的基石。不同于简单地将转录因子表达量和DNA可及性数据拼接在一起,TAM通过一种全新的方式整合了这些信息。这种设计基于一个重要认识:转录因子的浓度(通过表达水平体现)、DNA调控区域的开放程度,以及转录因子与DNA之间的相互作用强度,这三个要素在生物学过程中是密不可分的。
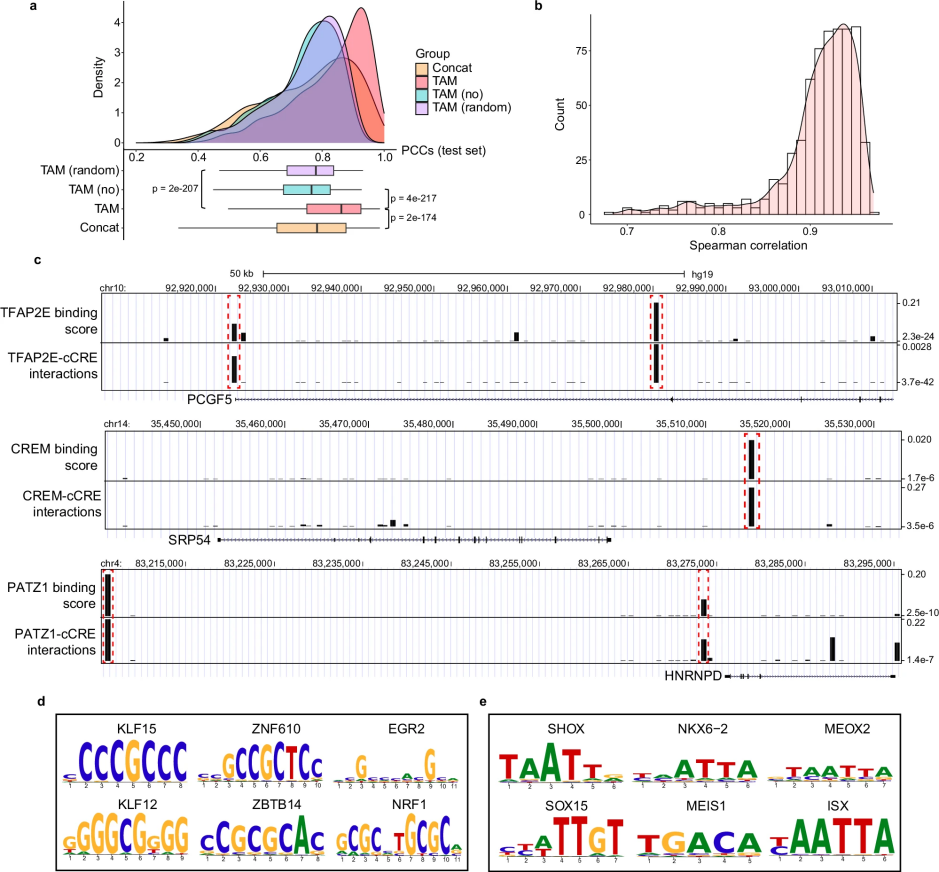
图 2
如图2a所示,研究人员通过预测667个差异表达基因的表达水平来评估TAM的性能。使用皮尔逊相关系数(Pearson Correlation Coefficient, PCC)进行评估,TAM模型达到了0.83的平均相关系数,显著优于其他特征组合方法。图2b的结果表明,模型学习到的转录因子与DNA调控区域之间的相互作用在重复实验中表现出很好的稳定性。
更有趣的是,如图2c-e所示,研究人员比较了模型学习到的相互作用强度与基于DNA序列预测的结合能力。结果发现,对于每个目标基因,平均有6.1(±2.5)%的转录因子表现出与序列预测一致的结合模式。特别是,那些具有较长DNA识别序列和较高GC碱基含量的转录因子,往往表现出更强的序列特异性结合能力。这可能是因为这类转录因子在结构上更稳定,与DNA的结合更依赖于特定的序列特征。相反,具有较短识别序列和较低GC含量的转录因子则可能倾向于以蛋白质复合物的形式间接与DNA相互作用。
为了进一步验证模型的可靠性,研究人员使用了三个外部单细胞多组学数据集(SNARE-seq、PBMC 10x multiome和DPCL)进行测试,并与现有的领先方法(SCENIC+和LINGER)进行比较。虽然TAM的设计初衷不是构建完整的基因调控网络,而是识别在特定数据中起重要作用的调控关系,但评估结果显示,TAM在准确性指标(F1 score)上仍然显著优于现有方法。特别是在识别最重要的转录因子-DNA调控区域相互作用方面,TAM表现出更高的精确度。
regX优先考虑潜在驱动β细胞过渡的TFs
研究团队使用regX模型分析了2型糖尿病发展过程中胰腺β细胞的状态变化。模型在细胞状态预测方面表现出色,准确率评分(F1 score)达到0.960±0.012。
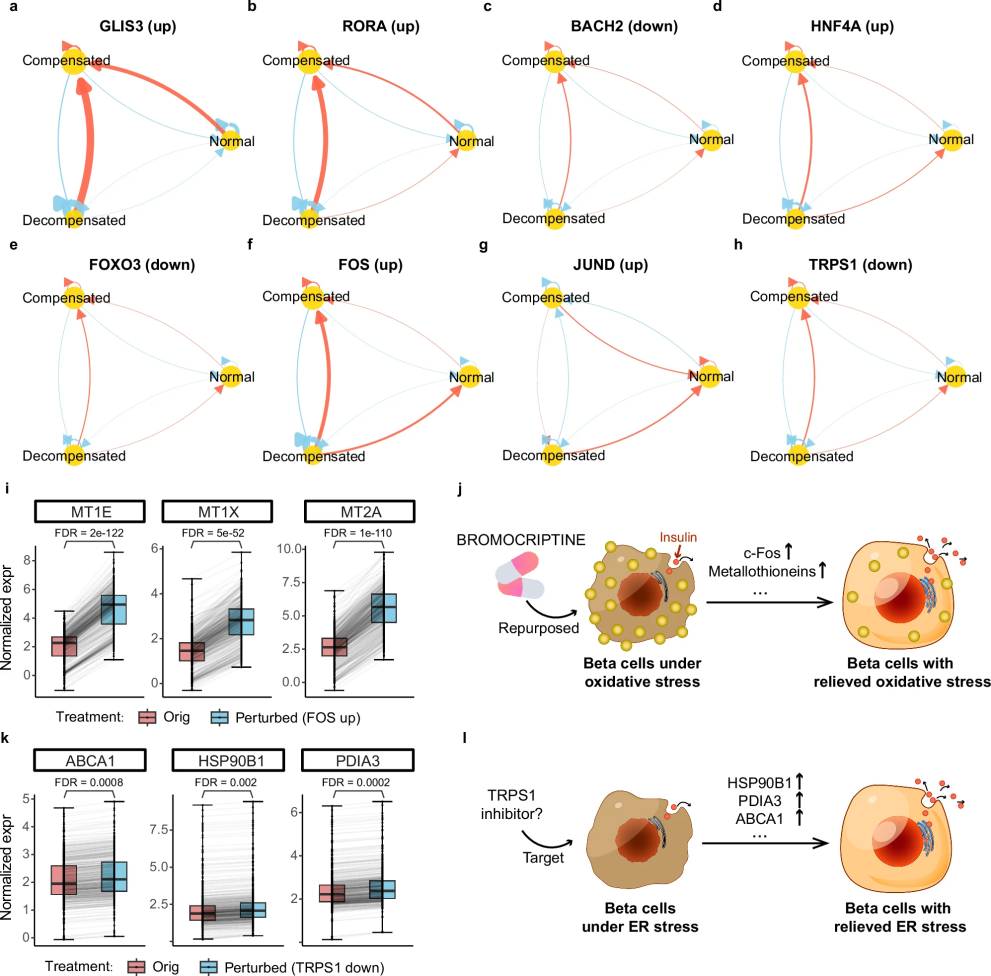
图 3
如图3a-h所示,通过计算机模拟实验,研究人员识别出23个可能驱动细胞状态转换的关键转录因子。这些因子中,21个在先前研究中已被证实与β细胞功能或2型糖尿病有关,其中10个是36种处方药、研究性药物或膳食补充剂的直接或间接作用靶点。研究发现了两个特别值得关注的发现:
首先,如图3i-j所示,研究揭示了一个有趣的药物重新利用案例。溴隐亭(Bromocriptine)原本用于治疗泌乳素过高、肢端肥大症和帕金森病,后来被发现可以用于辅助治疗2型糖尿病,但其作用机制一直不明确。regX模型的分析表明,溴隐亭可能通过与FOS基因相互作用,激活金属硫蛋白(Metallothionein)等抗氧化基因,从而减轻细胞的氧化应激,帮助恢复胰岛素的产生。
其次,如图3k-l所示,研究发现了一个潜在的新型治疗靶点TRPS1。模型预测,抑制TRPS1可以促进多个重要基因的表达,这些基因能够帮助细胞应对蛋白质折叠压力(内质网应激),并促进胰岛素分泌。虽然这些基因表达的变化幅度相对较小,但多个基因的协同作用可能产生显著的治疗效果。
表1:23个潜在驱动转录因子(pdTFs)在计算机模拟下调和上调实验中的信息
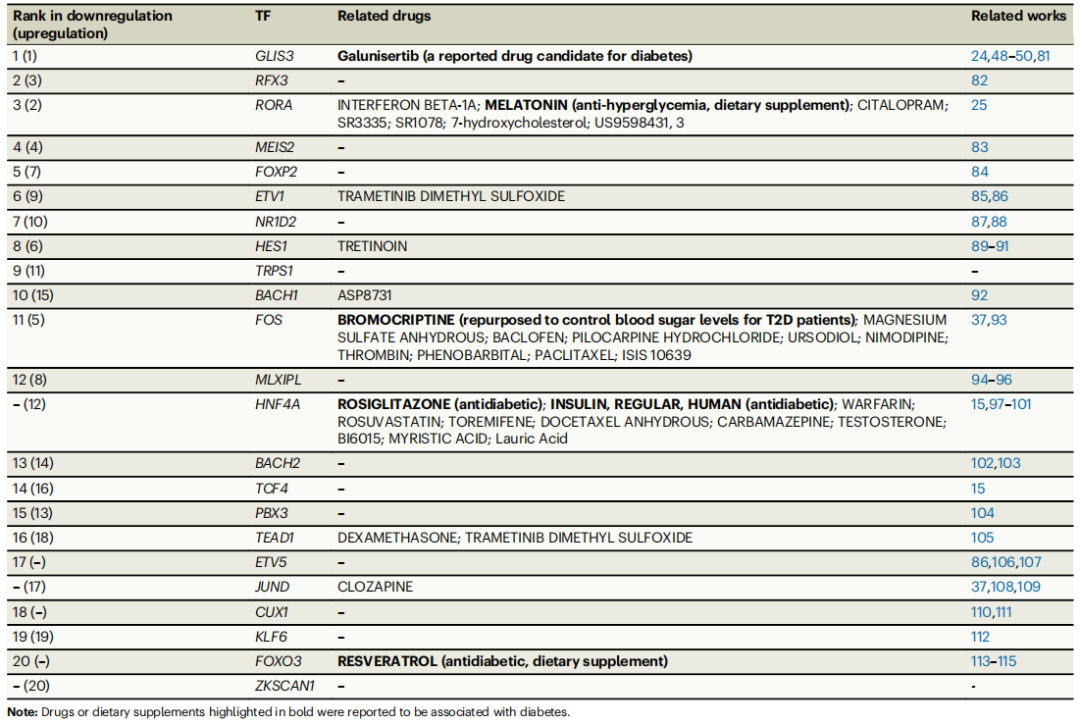
表1总结了这23个关键转录因子与现有药物和疾病的关联。这些发现不仅帮助解释了某些药物的作用机制,还为开发新的治疗方案提供了方向。特别是,那些已知作用于这些转录因子的药物可能具有治疗2型糖尿病的潜力,值得进一步研究。不过,对于新发现的治疗靶点如TRPS1,还需要更多的实验验证来评估其临床应用价值。
排名靠前的调控因子揭示2型糖尿病两类潜在致病SNPs
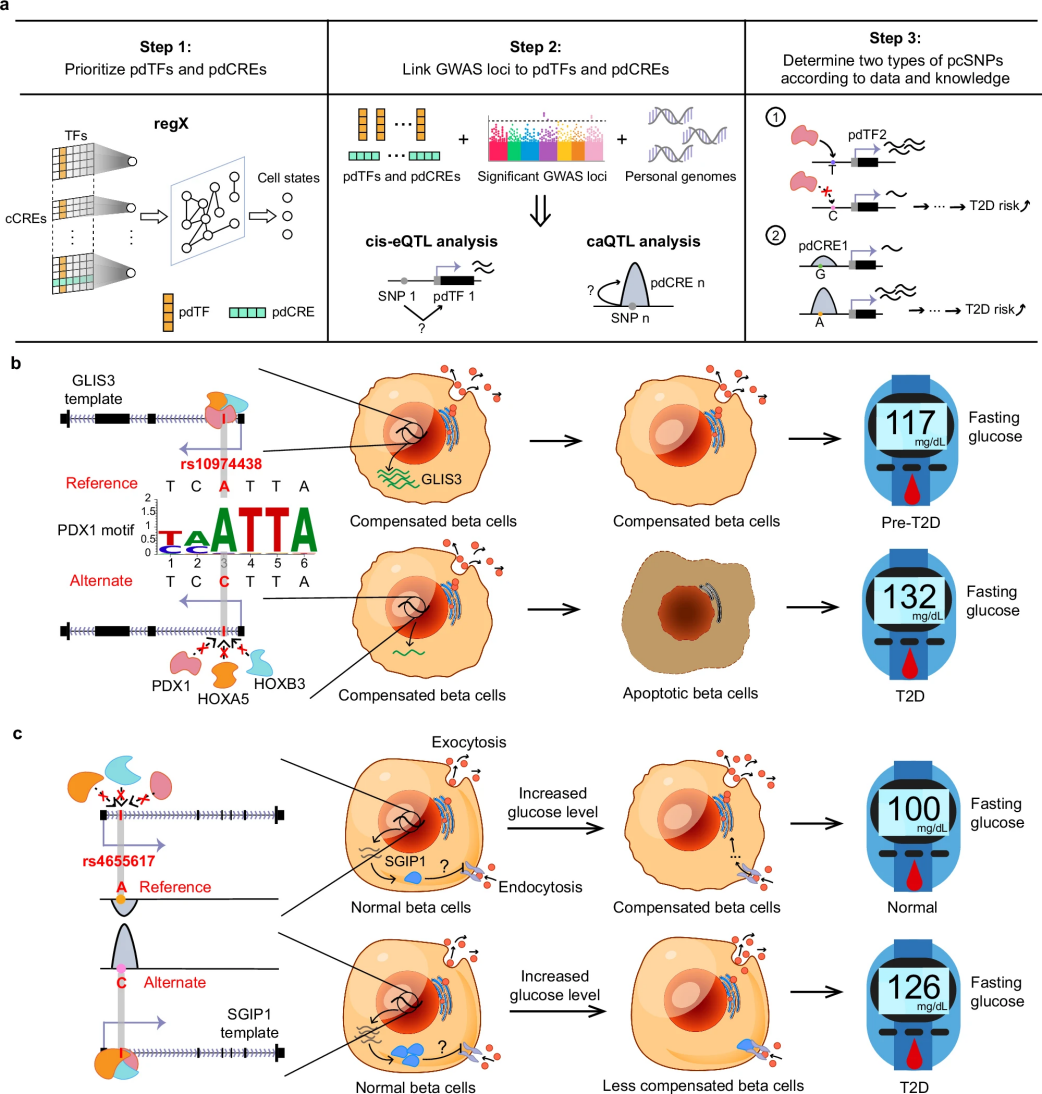
图 4
在全基因组关联研究(Genome-Wide Association Study, GWAS)领域,区分基因变异与疾病之间的因果关系一直是一个重要问题。如图4a所示,研究团队提出假设:那些影响疾病关键调控因子的基因变异位点很可能是导致疾病的原因。基于这一假设,研究人员利用regX模型发现了两类可能导致2型糖尿病的DNA变异位点(putative causal SNPs, pcSNPs)。研究团队分析了2,441个与2型糖尿病相关的基因变异位点,最终确定了5个此前未被报道的重要pcSNPs。这些DNA变异可以分为两类:
第一类是通过影响关键转录因子表达的pcSNPs。如图4b所示,以rs10974438为例,当这个位点发生从A到C的变异时,会降低多个转录激活因子与DNA的结合能力,导致GLIS3基因表达降低。GLIS3表达的减少会促进胰腺β细胞的死亡并减少胰岛素分泌,最终增加2型糖尿病的风险。
第二类是通过改变DNA的可及性来发挥作用的pcSNPs。如图4c所示,以rs4655617为例,当这个位点从A变为C时,会增加特定DNA区域的开放程度,从而提高SGIP1基因的表达。研究推测,SGIP1蛋白可能通过影响细胞的物质摄取过程(内吞作用),干扰胰岛素的正常分泌。当血糖升高时,SGIP1表达增加的细胞可能无法正常应对,增加了发生2型糖尿病的风险。
这些发现不仅帮助解释了基因变异如何导致2型糖尿病,还为开发新的治疗方法提供了方向。特别是对SGIP1的新发现,为进一步研究2型糖尿病的发病机制提供了新的视角。这些结果也证明了regX模型在揭示生物学机制方面的优势和可靠性。
regX识别毛囊发育过程中决定细胞命运的关键转录因子
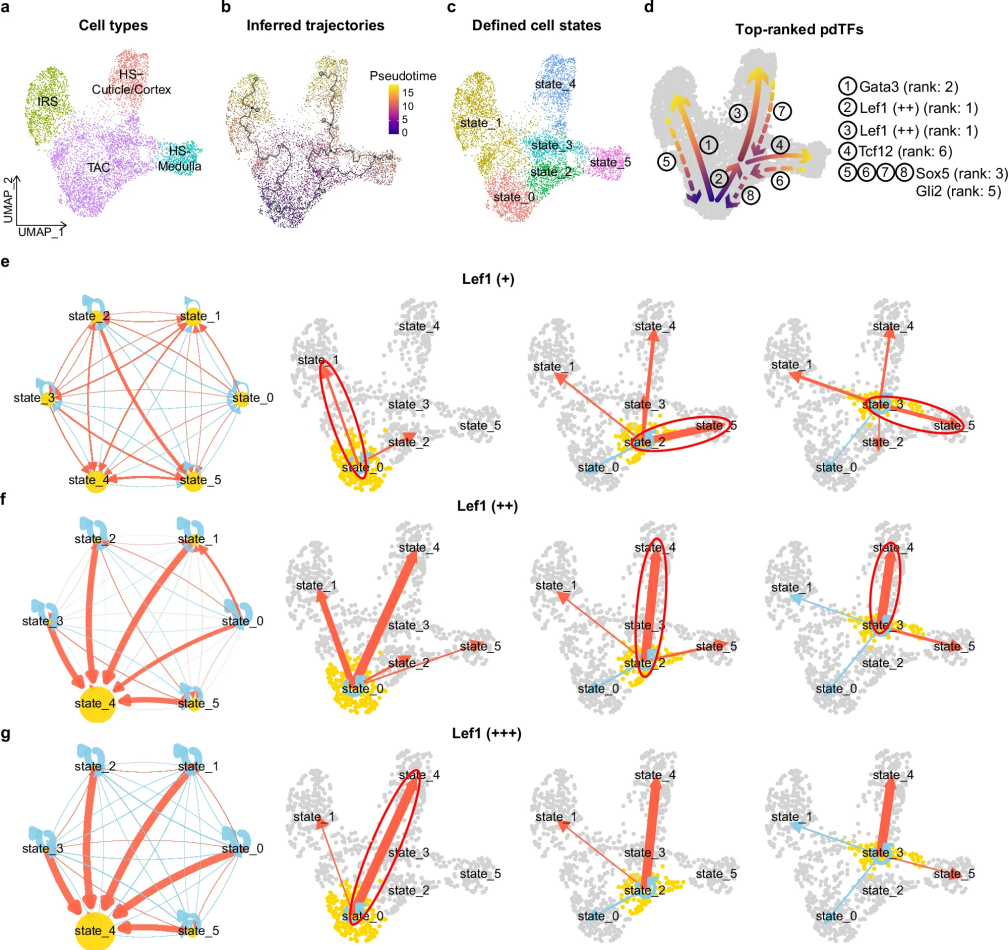
图 5
研究团队将regX模型应用于毛囊发育研究。如图5a所示,研究关注了毛囊发育过程中的几种关键细胞类型:瞬时扩增细胞(Transit-Amplifying Cells, TACs)、内根鞘细胞(Inner Root Sheath, IRS)、毛干角质层/皮质层细胞和髓质细胞。这个数据集与之前的2型糖尿病数据集有几个显著区别:它来自小鼠样本,使用了不同的测序技术,并且研究重点是发育过程而非疾病。研究团队创新性地利用基因本体论(Gene Ontology, GO)数据库中与毛囊发育相关的12个功能类别,构建了一个有向无环图来描述这些功能之间的关系。
如图5b-c所示,研究人员对模型结构进行了优化,引入了图注意力层(Graph Attention Layer, GAT),这种结构能够更好地捕捉基因之间的复杂关系。模型不仅可以预测细胞的离散状态,还能预测细胞发育的连续时间进程。评估结果显示,这种改进的模型结构在预测准确性上显著优于传统方法。通过计算机模拟实验,研究团队识别出了控制毛囊发育的关键转录因子。如图5d-g所示,其中最重要的发现是关于Lef1和Gata3这两个转录因子的作用机制:
Gata3的表达增加会明确地引导瞬时扩增细胞向内根鞘细胞发育;
Lef1的作用则更为复杂:
只有在高度激活时才能引导瞬时扩增细胞向毛干细胞发育;
不同的激活水平会导致细胞发育成不同类型的毛干细胞:高水平促进角质层/皮质层细胞的形成,低水平则倾向于形成髓质细胞。
这些发现突显了regX模型的独特优势:它不仅能识别关键的调控因子,还能定量分析这些因子的表达水平如何影响细胞的发育命运。这种深入的机制解析对于理解毛囊发育过程和开发相关治疗方法具有重要意义。
讨论
regX是一个创新的深度学习模型,通过整合多层次的调控过程来研究细胞系统。该模型将转录因子表达、染色质可及性和蛋白质相互作用等生物学机制融入网络设计,可以预测细胞状态转换并识别关键调控因子。研究团队将regX应用于2型糖尿病和毛囊发育研究,不仅发现了新的治疗靶点和药物重新利用的可能性,还揭示了基因变异导致疾病的潜在机制。尽管模型仍有改进空间,但这项工作为分析单细胞多组学数据和理解复杂生物过程提供了新的研究范式。
编译|于洲
审稿|王梓旭
参考资料
Xi X, Li J, Jia J, et al. A mechanism-informed deep neural network enables prioritization of regulators that drive cell state transitions[J]. Nature Communications, 2025, 16(1): 1284.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢