DRUGAI
高通量组学技术的快速发展带来了生物数据的指数级增长,这一速度往往超过了研究人员提取分子层面洞见的能力。在自然语言处理领域,大语言模型通过整合海量数据集构建统一模型,并在多个下游任务中展现出强大能力,为应对数据洪流提供了新思路。借鉴这一理念,研究人员设想构建多模态基础模型,并在包括基因组学、转录组学、表观基因组学、蛋白质组学、代谢组学以及空间组学在内的多类组学数据上进行预训练。此类模型有望以前所未有的方式表征细胞的分子状态,描绘出细胞、基因与组织的整体图谱。
特定情境下应用这些基础模型的迁移学习能力,将赋能一系列关键任务,包括新型细胞类型识别、生物标志物发现、基因调控机制推断乃至虚拟干预实验的开展。这一新范式或将开启人工智能赋能的分子细胞生物学分析新时代,不仅有助于实验设计,也将深刻拓展研究人员对生命科学的理解。

分子细胞生物学的核心目标之一,是揭示和表征DNA、RNA、蛋白质和代谢物之间的动态相互作用与调控机制。这种全面理解将为捕捉、模拟并预测细胞发育与状态变化奠定基础。长期以来,研究人员围绕“全细胞建模”或“虚拟细胞”等概念不断探索。传统模型多由规则驱动的子模块或常微分方程构成,每个子模块负责模拟一个生物过程。例如,早期的全细胞模型用于模拟支原体的细胞过程,采用了一组相互连接的微分方程。但这类方法常因动态过于简化或模型不稳定而受限,难以处理大规模、非线性、多样化的细胞环境,尤其难以应用于复杂的人类组织系统中。
近年来,技术进步带来了新机遇。一方面,测序、单细胞技术、冷冻电镜及质谱等手段推动了多组学数据的快速积累;另一方面,大规模机器学习模型的兴起也为生物数据的整合与理解提供了新的计算工具。目前,来自基因组学、转录组学、蛋白质组学等多源数据的生成速度正呈指数级增长,为研究人员解析正常与病理状态下的分子功能带来了前所未有的可能。同时,多个全球科研项目已汇聚了来自数百万细胞的多模态数据。此外,近年涌现的多组学联合测量手段,允许在同一细胞中获取两种甚至三种模态的数据,进一步凸显了整合建模的紧迫性。
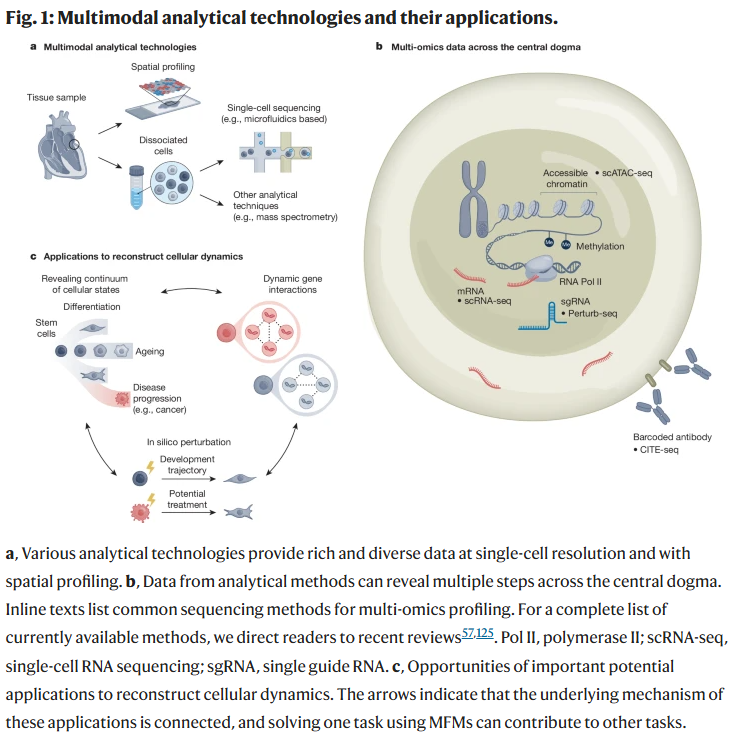
此背景下,研究人员提出构建多模态基础模型(MFMs)作为应对这一挑战的新策略。其核心思想是在多模态数据上以自监督方式进行预训练,使模型能够自动学习基础分子知识,并具备跨任务能力。这类模型可接受不同类型的输入数据,并用于多种任务,如表征细胞状态、预测基因功能,或推演健康与疾病条件下的动态变化。
接下来的章节将深入介绍MFMs的结构与能力:“多模态基础模型概述”将说明其在加速“实验-计算-反馈”闭环中的潜力;“MFMs的应用前景”将探讨其在组织异质性、基因功能预测和虚拟干预等方面的价值;“构建MFMs的路径”将讨论实现这类模型所需的计算架构与数据基础;“面临的挑战与局限”则将概述当前开发与应用中仍需解决的关键问题。
多模态基础模型概述
基础模型的理念
基础模型是一类通过自监督学习在大规模数据集上训练的深度神经网络模型,能够通过迁移学习广泛适用于各种下游任务。在自然语言处理领域,以Transformer为基础的模型(如GPT和LLaMA系列)已在海量文本上训练,并能通过微调或上下文学习快速适应多种任务。近期,这类模型的成功也拓展到了图像、视频等领域,并展现出跨模态生成能力。

分子细胞生物学中,基础模型为统一理解多种生物过程提供了强大手段。其关键优势在于能够学习并表征细胞系统中复杂而互联的关系。通过多组学数据的训练,模型能够挖掘出传统单模态实验中难以察觉的规律与联系,甚至揭示一些普适的生物学原理。
多模态基础模型的特性与架构
多模态基础模型(MFMs)应具备处理多种类型数据(如bulk与单细胞测序)的能力,涵盖转录组、蛋白质组、代谢组和表观组等多种模态。其训练流程包括两个阶段:
首先,模型在大量多模态数据上以自监督方式进行统一预训练,捕捉基因、蛋白、通路等生物过程的潜在特征;
然后,通过迁移学习(如微调或上下文学习)适配至具体任务,实现如细胞状态追踪、新型细胞类型识别、扰动响应预测等应用。
Transformer架构因其内部注意力机制,已成为当前最主流的基础模型框架,并在建模语言、图像语义方面表现出色。在生物科学中,多个具有里程碑意义的研究也采用Transformer来预测蛋白结构、生成新型蛋白,或表征基因表达和染色质状态,进一步验证了其在模拟生物分子交互中的潜力。近年来,包括scGPT、GeneFormer、scBERT等模型也开始应用Transformer于单细胞组学领域,用于学习细胞与基因的表示。
数据驱动的新范式:实验反馈闭环
多模态基础模型的提出正在推动分子细胞生物学的范式转变。传统的研究流程以假设为驱动:识别现象、提出假设、设计实验验证、再修正理论。这种方式强调专科化,例如研究癌细胞以理解癌症,研究心肌细胞以理解心脏病,其隐含假设是不同生物背景之间知识不能通用。
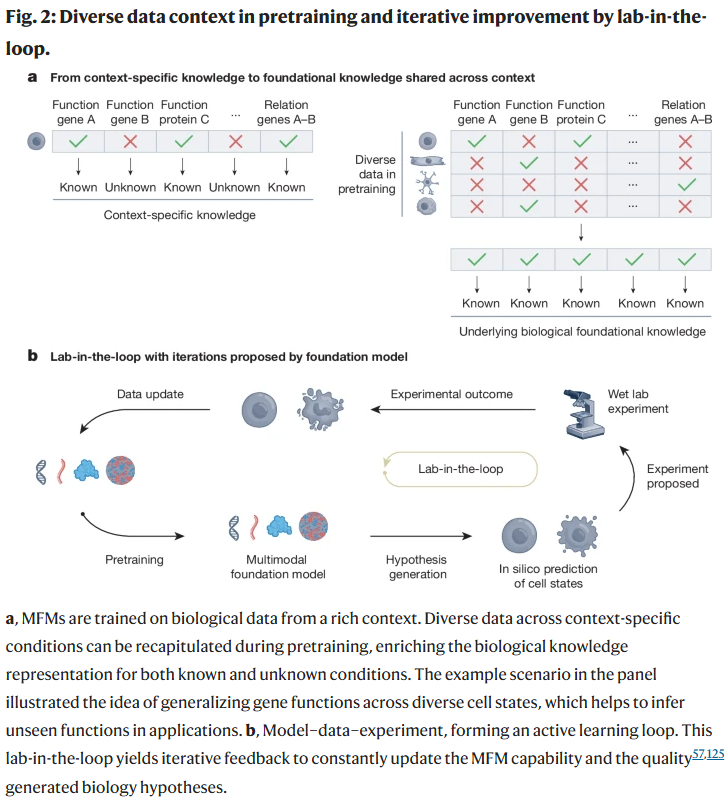
如今,MFMs 提供了一种全新的数据驱动流程:研究人员先在大规模、高维度、无假设的数据基础上训练模型,再通过模型抽象出有生物学意义的表示。模型若能有效再现真实系统(例如可复现实验数据),便可用于探索系统规律、推演潜在机制。这种流程不仅提升了建模速度与精度,也使模型具备在不同背景下迁移应用的能力。
以癌症与心血管研究为例,通过在涵盖多种组织与状态的大规模数据中训练模型,研究人员可捕捉影响细胞行为的共性机制,实现跨学科、跨系统的知识迁移。
实验-计算闭环(lab-in-the-loop)
这一数据驱动流程的核心,是“实验-计算闭环”策略:实验与计算团队协同进行,模型帮助优化实验设计,实验反过来又用于改进模型性能。训练完成的基础模型可预测潜在结果,引导下一轮更具信息量的实验。例如,模型可预测某药物对不同细胞系的效果,并优先建议对预测不确定性高的细胞系进行验证。这些实验结果再反馈至模型训练中,最终形成一个具备模拟能力的“分子细胞模拟器”,为后续实验设计与理论研究提供指导。
多模态基础模型的应用前景
通过整合多组学测量结果,多模态基础模型(MFMs)可以构建覆盖从基因到转录本再到蛋白质的全流程表达,全面表征特定基因、细胞类型及其在动态环境下的调控关系。以下是几个MFMs尤具潜力的重要应用方向。
表征组织异质性
单细胞组学技术的进展,使研究人员得以识别超越传统表面标志物的细胞亚群,尤其在肿瘤等复杂组织中揭示细胞的异质性。例如,单细胞RNA测序揭示了胶质母细胞瘤中的表达差异,而表观组和蛋白质组数据则进一步描绘了其功能多样性。将多模态数据整合于同一细胞层面,有助于更细致地描绘细胞状态的过渡与谱系关系。
MFMs 能够突破传统方法仅聚焦离散细胞类型的限制,定义连续的细胞状态谱,从而推测细胞在刺激前后的变化过程。其能力主要体现在三个方面:
上下文化细胞状态:通过训练模型整合多组学数据,MFMs 可将细胞嵌入一个连续的表达空间,映射其在发育轨迹或疾病进程中的位置。
比较细胞状态:MFMs 支持跨样本、跨模态的整合分析,有助于比较健康与疾病条件下的细胞特征。
补全细胞状态:面对实验中常见的组学缺失问题,MFMs 可通过学习获得的生成能力补全缺失模态,实现对完整细胞图谱的重建,尤其适用于临床样本难以进行多模态测量的情况。
预测基因功能与调控机制
MFMs 可在大规模异质性疾病数据中学习潜在模式,从而识别关键基因模块、蛋白标志物或代谢特征。研究人员已展示了仅基于基因序列预测功能的可行性,而加入表观信息如染色质开放性或甲基化状态,可进一步提升推理精度。
除了功能预测,MFMs 还可用于重建特定情境下的基因调控网络(GRNs)。这源于两个核心优势:
多组学整合能力:传统的调控网络构建依赖于表达共现或已知数据库,而MFMs 可将转录调控、DNA结合、RNA剪接与蛋白修饰等事件纳入统一建模,获得更完整的调控视角。
情境适应能力:基因调控高度依赖于细胞类型与生理状态,MFMs 可通过迁移学习灵活适应不同组织、时间或疾病环境下的网络结构,进而解析复杂生物系统的动态调控逻辑。
此外,研究人员也正在探索将已有调控知识融入MFMs,以更准确地复现发育过程和细胞时间序列中的调控关系。
虚拟干预模拟(In silico perturbation)
MFMs 有望模拟基因突变或药物作用对细胞状态的影响,为新药开发与机制研究提供高效路径。已有多种模型初步展示了在细胞嵌入空间进行扰动并预测转录响应的能力,而MFMs 可将应用拓展至表观组、蛋白组等多个层面。
具体来说,MFMs 可:
在整合表达、表观、蛋白信息的基础上,构建完整细胞状态嵌入;
在特定细胞背景下施加扰动,模拟其响应;
借助空间与时间数据,追踪扰动在组织与发育过程中的传播路径;
利用已学习的通路知识与调控网络,预测复杂下游效应。
随着CRISPR结合单细胞组学的实验数据不断积累,例如Perturb-seq等方法,MFMs 可以在训练后预测任意基因组合扰动的响应,显著加速基因功能研究与治疗靶点的发现。
构建分子细胞生物学中的多模态基础模型
为了实现前述应用,多模态基础模型(MFMs)需要具备若干关键的技术能力。本节将从数据需求与计算架构两个方面探讨其构建思路。
模型训练所需的数据资源
预训练高效的MFMs需依赖大规模、异质化的多组学数据,涵盖bulk测序、单细胞测序、空间转录组、染色质可及性与蛋白质组等。尽管已有如HuBMAP、ENCODE、IHEC 和 HCA 等公开数据库,但目前具备跨模态配对数据的资源仍较为稀缺。这类配对数据通过近年发展出的技术如10X Multiome、CITE-seq、ASAP-seq等获得,可同时测量单细胞中多种组学模态,对模型学习跨模态联系至关重要。
尤其是单细胞数据在训练MFMs中扮演重要角色,它揭示了bulk数据中难以观测的个体异质性。例如CellxGENE数据库中已收录超过9000万个细胞,体量在一年内翻了三倍。尽管RNA测序数据目前构成主要训练集,其他模态如ATAC-seq或蛋白组数据的样本数量仍有限,未来仍需持续扩展多模态数据规模与覆盖范围。
此外,统一整合来自不同项目的海量数据也非常关键。包括元数据标签标准化、质量控制和归一化等步骤,甚至可以通过MFMs自身辅助完成这些前处理工作。
核心计算模块设计
统一的多模态数据编码方式
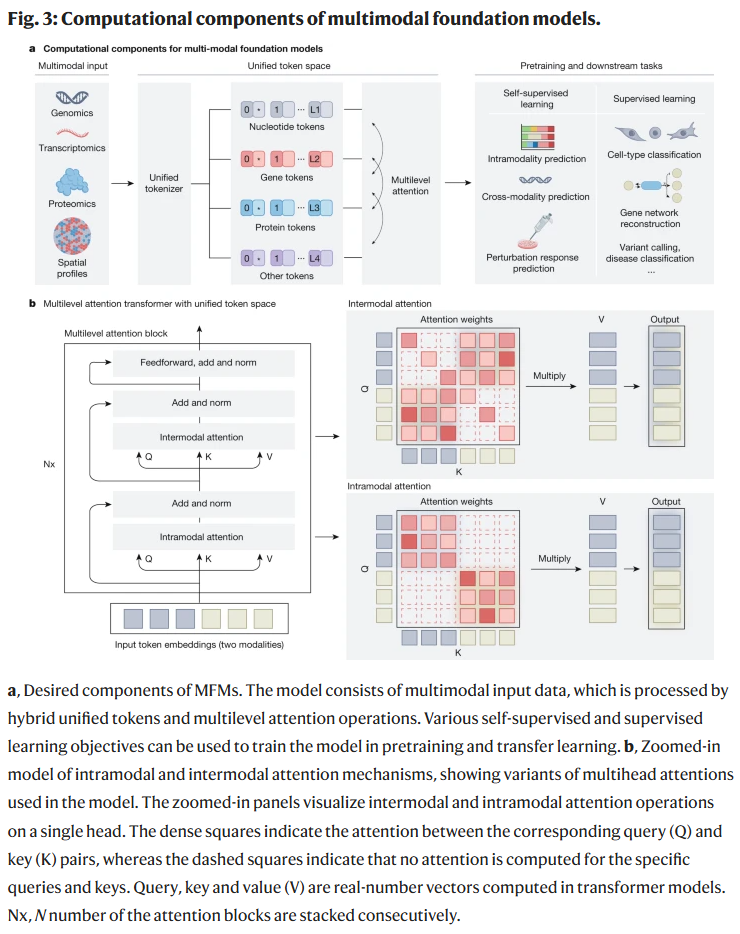
多组学数据具有模态复杂、粒度不一的特点,从单个核苷酸到整个蛋白质跨度较大。一个可行方案是借鉴自然语言模型的思路,通过统一的token化机制将不同模态的基本单位编码至共享的向量空间中。例如,将DNA序列中的k-mer、蛋白质中的氨基酸片段,甚至是整个基因都视为不同层级的token,可按需生成低、中、高分辨率的表示。
多层级注意力机制
分子数据天然具备多层次结构,如核苷酸-基因-通路等层级。研究人员提出采用混合型注意力机制,在模型中引入局部(同层)与全局(跨层)自注意力机制,实现细粒度信息建模与整体系统推理的协同。例如,局部注意力可建模基因内部的调控关系,而全局注意力则连接基因与蛋白之间的交互,进而构建完整的生物通路图景。
自监督任务与任务引导(Prompt)
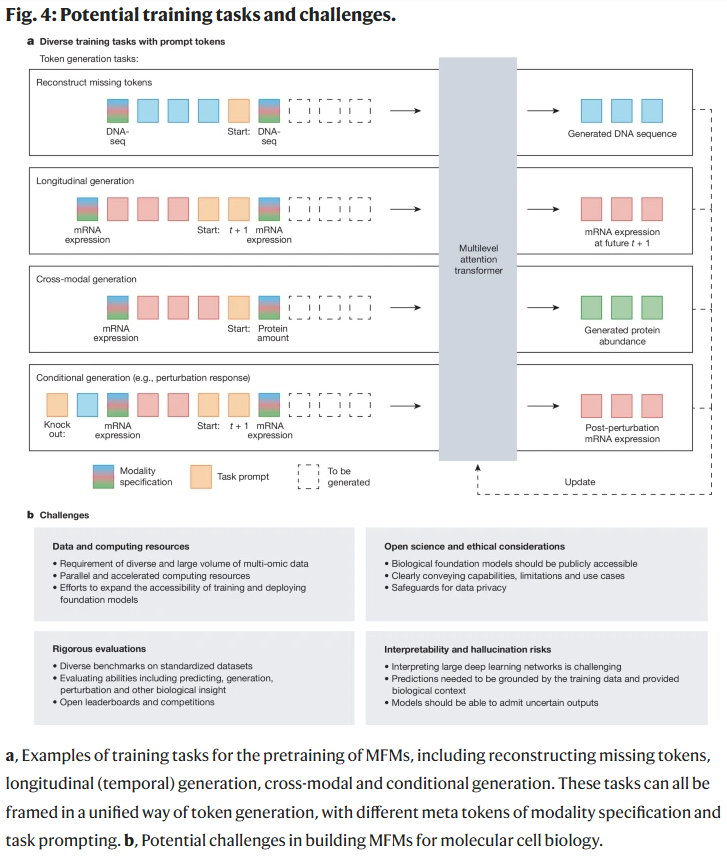
MFMs 可在无标签多组学数据上进行自监督训练,任务包括遮蔽表达恢复、模态缺失填补、扰动响应预测等。这些任务可分为模态内与跨模态两类:
模态内任务:如预测被随机遮蔽的转录数据,或根据初始细胞状态预测扰动后的表达变化。
跨模态任务:如对同一细胞不同模态的正负样本对进行对比学习,或引入“任务token”来引导模型完成特定跨模态预测(如mRNA到蛋白表达)。
此外,研究人员还提出用统一的“Prompt驱动生成框架”,通过少量指令token(如“<转录组>”“”“<开始生成>”)即可灵活控制不同任务,实现模型参数的高效共享。
值得注意的是,MFMs 在训练中可利用医学背景信息,如年龄、性别、疾病状态等结构化标签,作为监督信号加入训练,这也是生物学场景区别于通用语言模型的特点。
人类知识的整合策略
为了增强模型的生物学归纳能力,研究人员也探索如何将结构化与非结构化知识引入模型预训练:
结构化知识:如基因通路、GO术语、蛋白互作网络等,可转化为知识图谱,通过图嵌入引导token初始化,从而将已有调控关系纳入模型表示。
非结构化知识:如生物医学文献中的知识,可借助大型语言模型(如BioGPT、Med-PaLM)提取文献向量嵌入,再与实验数据联合训练,实现对实验和文献知识的双重学习。
这种“双源知识融合”的策略,正在成为推动生物基础模型能力跃升的重要方向。
多模态基础模型的挑战与局限
尽管多模态基础模型(MFMs)在分子细胞生物学中展现出巨大潜力,其广泛应用仍面临诸多技术与监管挑战。以下是当前亟需关注的几个核心问题。
数据与算力资源
预训练MFMs需依赖配对且对齐的多组学数据,理想状态下应包含空间信息与时间序列样本。然而此类数据目前仍较稀缺且分散,全球范围内的协作共享将是推动数据整合与算法开发的关键。
与此同时,大规模模型的训练需耗费大量算力资源(如高端GPU),限制了模型的可及性,也增加了能耗负担。低资源建模技术(如LORA或Adapter Transformer)或将成为发展绿色AI与推动MFMs普及的突破口。
此外,合成数据作为真实数据的补充,也可用于填补数据缺口,特别是在模态不完整或样本稀缺的情况下,为模型训练提供多样性支持。
严谨的评估体系
MFMs的广泛应用依赖于严格且系统的评估方法。需要构建多维度基准任务,如细胞类型识别、疾病建模、in silico扰动预测等。然而,当前评估多依赖人工注释,这可能掩盖模型识别罕见类型或新模式的能力。例如,当模型识别出文献未标注的新细胞亚群时,反而可能被误判为错误预测。
因此,研究人员呼吁开发更客观、去人为偏见的评估指标,以公正反映模型的探索能力。公开竞赛与排行榜机制(如OpenProblems与DREAM挑战)已在推动多组学方法标准化方面发挥重要作用,未来应进一步拓展规模与覆盖范围。
可解释性与“幻觉”风险
大型深度学习模型的“黑箱”问题使得其可解释性备受关注。MFMs可预测基因表达、细胞类型等复杂表型,但为何预测某基因上调,或为何识别某类调控网络,仍难以追溯。近期如Kolmogorov–Arnold网络等方法为模型决策过程提供了符号解释的可能性,有望提升生物模型的透明度。
“幻觉”问题,即模型生成看似合理但不真实的输出,是另一个隐忧。MFMs在缺乏事实支持或上下文不符时可能给出虚假建议,尤其在临床应用场景中尤为危险。因此,需开发不确定性评估机制,在模型无法可靠预测时,主动表明“不确定”状态,避免误导下游使用者。
开放科学与伦理审视
MFMs的发展需在透明与开放的基础上进行。训练数据、模型能力、适用范围与局限都应明确披露,以建立信任。类比自然语言模型的发展路径,MFMs亦应接受多维度审视,包括数据来源、方法公开、伦理风险与公平性等方面。
此外,涉及患者数据的模型部署必须严格保障隐私与安全。模型应确保各类人群的代表性,避免偏倚和不平等预测。特别是在医疗推荐场景中,必须保证预测结果的准确性,并提供合理解释,以支撑实际应用决策。
促进模型与数据的开放获取,将是推动整个领域包容性与持续创新的关键。
面向未来的共创生态
整合多组学的MFMs有望重塑分子生物学的研究范式,实现对生命系统前所未有的理解深度与广度。这一过程需要生物学家、数据科学家、人工智能研究者与伦理专家的通力合作,共同推动数据标准化、模型优化与公平共享。
未来,MFMs有望在个性化治疗、疾病建模、新药开发等领域发挥关键作用,正如人类细胞图谱等项目已在医学研究中取得的变革性影响。可以预见,分子生命科学的未来,将由一个开放、协同、充满愿景的科研生态系统共同推动。
整理 | WJM
参考资料
Cui, H., Tejada-Lapuerta, A., Brbić, M. et al. Towards multimodal foundation models in molecular cell biology. Nature 640, 623–633 (2025).
https://doi.org/10.1038/s41586-025-08710-y
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢