
图|Bo Wang,多伦多大学医学生物物理学助理教授
随着 AlphaFold 在蛋白质折叠上的突破,以及 AI for Science 领域的不断创新,人工智能(AI)正势不可挡地重构生命科学的研究范式。
高通量组学技术的快速发展,使生物学数据量呈指数级增长,远远超出了我们从中提取分子层面信息的能力。大语言模型(LLM)通过整合海量数据并实现多任务应用,为解决海量数据处理问题提供了思路。
受此启发,华裔学者、多伦多大学医学生物物理学助理教授 Bo Wang 团队及其合作者提出了“开发面向分子细胞生物学的多模态基础模型(MFM)”的构想,这类模型在基因组学、转录组学、表观基因组学、蛋白质组学、代谢组学和空间剖析进行预训练,能够表征细胞分子状态,构建细胞、基因和组织的整体图谱。
相关观点文章以“Towards multimodal foundation models in molecular cell biology”为题,已发布在国际权威科学期刊 Nature 上。

文章链接:
https://www.nature.com/articles/s41586-025-08710-y
研究团队表示,通过迁移学习,MFM 可以应用于多种下游任务,例如新型细胞类型识别、生物标志物发现、基因调控推断和虚拟扰动等,有望开启 AI 赋能的生物学分析新时代,揭示分子细胞生物学的复杂机制,支持实验设计,并扩展我们对生命科学的理解。
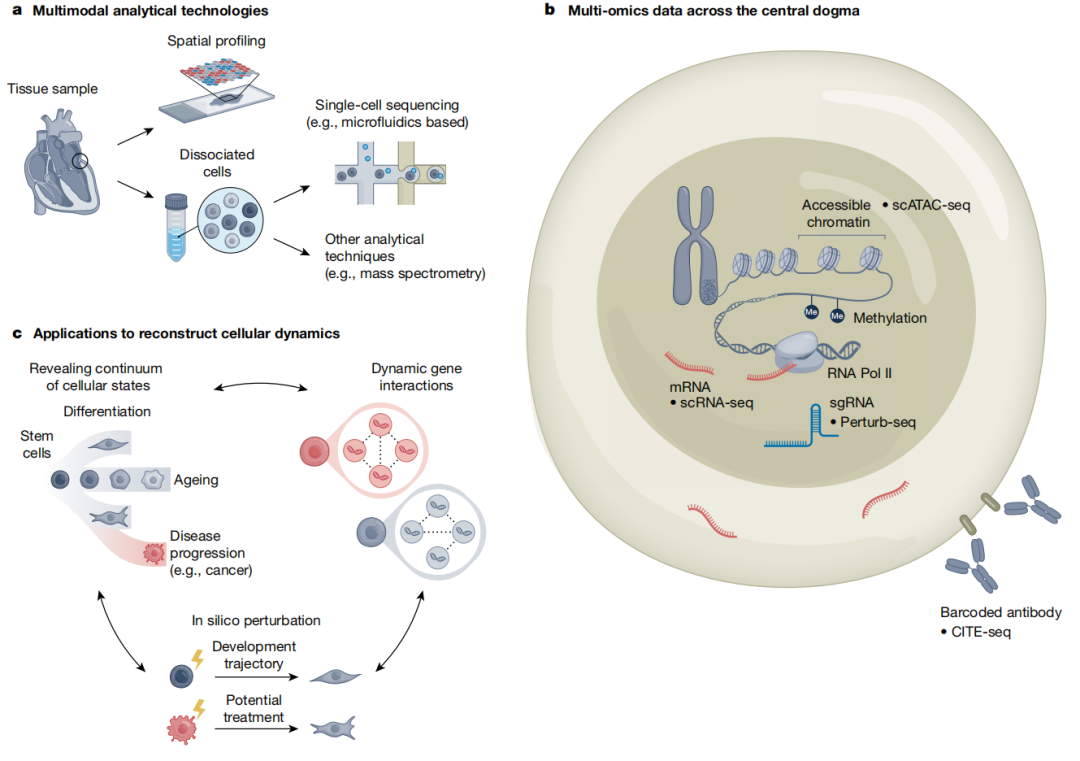
图|多模态分析技术及其应用。A. 各种分析技术可提供丰富多样的单细胞分辨率和空间剖析数据;B. 来自分析方法的数据可揭示跨越中心法则的多个步骤;C. 重建细胞动力学的重要潜在应用机会。箭头表示这些应用的基本机制是相互关联的,使用 MFM 解决一项任务可以促进其他任务的完成。
MFM 与分子细胞生物学:Lab-in-the-loop
基础模型是通过对海量数据集进行自监督学习训练的深度神经网络计算模型,因此通过迁移学习在广泛的下游任务中展现出强大的能力。
在自然语言处理领域,基于 Transformer 的基础模型,如 GPT 和 Llama 系列,在庞大的文本语料库上进行训练,可以通过微调或上下文学习快速适应各种下游任务。基础模型也已扩展到了自然图像和视频,并具备了语言与图像之间的跨模态生成能力。
在分子细胞生物学领域,基础模型为整合多样生物过程的认知提供了一种方法。生物基础模型的核心优势在于其能够学习并表征细胞系统复杂的相互关联特性。通过在多组学数据上进行训练,这些模型能够揭示孤立实验或单一模态分析中不易察觉的细微模式与关联,可能揭示出在更狭窄研究中被掩盖的普遍生物学原理。
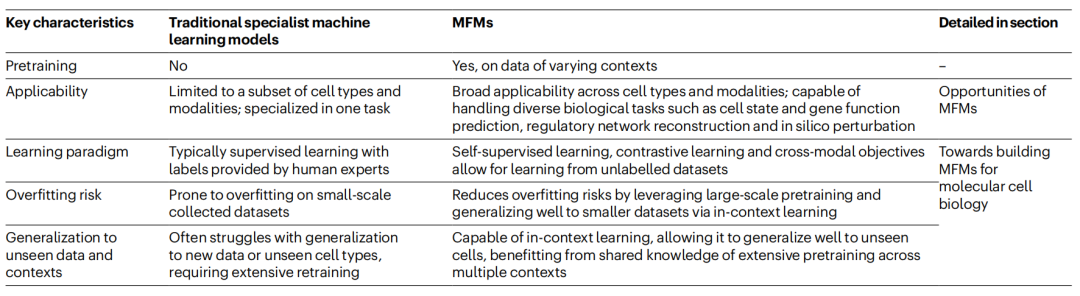
图|传统机器学习模型与分子细胞生物学 MFM 的比较
MFM 通过自监督学习在海量多组学数据上进行预训练,能够捕捉生物分子间隐秘的交互模式。例如,基于 Transformer 架构的 MFM 利用注意力机制模拟 DNA 序列到基因表达的动态过程,其核心优势在于打破单一模态分析的局限,揭示跨组学数据的深层关联。这种能力使得 MFM 在下游任务中展现出惊人潜力:从重建细胞发育轨迹,到预测基因扰动响应,再到发现新型生物标志物,均能提供超越传统方法的精准洞察。
研究团队特别强调了 Lab-in-the-loop 的创新工作流程。在这种模式下,实验设计与计算模拟形成闭环反馈:MFM 通过预测未知细胞系的药物敏感性指导实验方向,实验结果又反哺模型训练,形成知识迭代。这种数据驱动的跨领域知识迁移,突破了传统假设驱动研究的局限,为复杂生物系统建模提供了全新思路。
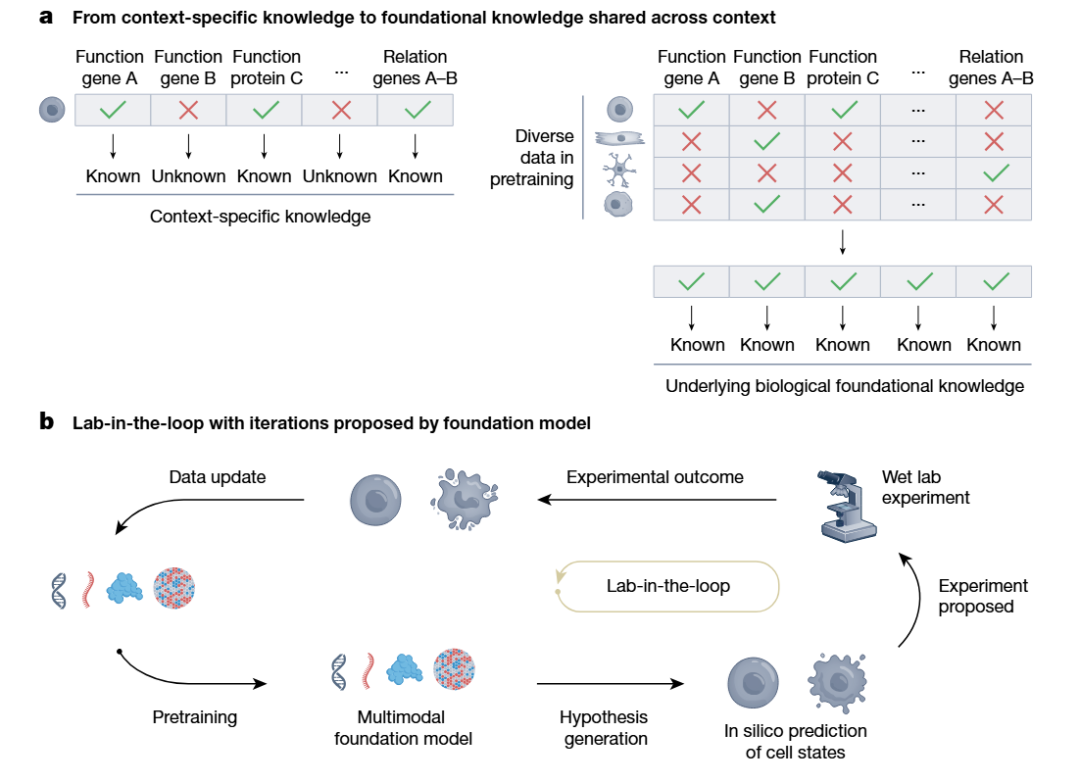
图|不同数据背景下的预训练和 Lab-in-the-loop 迭代改进。a. MFM 在来自丰富背景的生物数据上进行训练。在预训练期间,可以概括来自特定上下文条件的多样化数据,丰富已知和未知条件下的生物学知识表示。面板中的示例场景说明了在不同细胞状态下概括基因功能的想法,这有助于在应用中推断出未见过的功能;b. 模型 - 数据 - 实验,形成一个主动学习循环。Lab-in-the-loop 模式产生迭代反馈,以不断提升多模态基础模型的能力和生成的生物学假设的质量。
机遇
通过整合多模态数据,MFM 在表征细胞状态、预测基因功能以及重建基因调控网络等方面展示出了独特的优势。
在表征细胞状态方面,MFM 能够通过整合不同组学数据,更全面地理解细胞状态的连续性,从而更准确地比较不同细胞状态,并补全缺失的组学数据,例如在临床样本中预测代谢组学数据。
在预测基因功能和调控方面,MFM 能够学习多组学数据中的统一模式,从而预测基因功能,并重建特定环境下的基因调控网络,例如结合转录组和染色质可及性数据,揭示重要的调控因子。
在虚拟扰动方面,MFM 能够预测遗传或化学扰动对细胞状态的影响,从而加速基因调控理解和新治疗方法发现,例如预测药物在未知细胞系上的疗效,并指导实验验证。
为了实现这些潜在应用,研究团队指出了分子细胞生物学 MFM 应具备的一些关键技术特性。
首先,MFM 的训练需要大规模、多样化的多组学数据,包括单细胞测序、空间转录组学和纵向样本等,这些数据可以从全球细胞图谱等资源中获得,但需要进一步整合和标准化。研究团队表示,为了解决数据量不足的问题,可以考虑利用合成数据作为补充。
其次,研究团队提出了 MFM 的计算组件,包括统一的多模态数据表示、混合多层注意力机制、提示驱动的训练任务和人类知识的整合。
为了应对不同尺度的生物分子相互作用,MFM 需要构建统一 token,实现早期融合,并采用混合多层注意力机制,区分局部(单模态)和全局(跨模态)注意力。为了实现多种下游任务,MFM 需要设计提示 token 控制的统一框架,并包含单模态和跨模态的自监督学习任务,例如掩码语言模型、对比学习、跨模态预测和条件生成等。
此外,研究团队认为,将人类知识融入 MFM 预训练过程十分重要,例如将通路、基因本体、蛋白质相互作用网络和文献等知识以图嵌入或向量嵌入的形式加入模型,从而提供有用的归纳偏差,增强模型的预测能力。
挑战和展望
然而,在推广应用 MFM 的过程中,仍然存在技术和监管方面的挑战和限制。尽管在构建分子细胞生物学 MFM 时遇到的这些挑战与一般领域的基础模型有一些相似之处,但研究团队发现,该领域的具体要求和潜在解决方案往往独特。他们强调了以下几个问题:
数据和计算资源:需要多样化和大量的多原子数据;并行和加速计算资源;努力扩大训练和部署基础模型。
开放科学与伦理考虑:生物基础模型应向公众开放;明确传达能力、局限性和使用案例;保障数据隐私。
严格的评估:标准化数据集上的各种基准;评估包括预测、生成、扰动和其他生物洞察力在内的能力;公开的排行榜和竞赛。
可解释性和幻觉风险:解读大型深度学习网络具有挑战性;预测需要以训练数据为基础,并提供生物背景;模型应能接受不确定的输出结果。
将 MFM 应用于整合多种组学数据,有望以前所未有的规模和精度,推动分子生物学的变革。要实现这一构想,需要生物学家、数据科学家、人工智能研究人员和伦理学家通力合作,以生成高质量数据、完善模型并确保可访问性。
展望未来,将 MFM 融入医学领域,可推动个性化治疗、疾病建模和药物发现等领域的创新。这与细胞图谱(如 HCA)在医学研究中已经发挥的变革性作用如出一辙。从本质上讲,分子发现的未来将由一个充满活力、具有共同愿景的合作生态系统来孕育,使科学界有能力解决生物学和医学中一些最紧迫的挑战。
作者:锦鲤
如需转载或投稿,请直接在公众号内留言
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢