
数字病理图像作为一种全切片病理图像 (WSIs),能够对组织切片的高分辨率数字化呈现,全面展示细胞形态、结构和空间分布特征。与传统玻璃切片相比,WSIs 不仅便于存储和分析,还能在多个尺度上提供更直观的组织视图,因而在病理诊断与生物医学研究中应用日益广泛。通过对这些图像的分析,科研人员可以探索细胞空间组织与基因表达之间的内在联系,从而揭示多细胞系统中复杂的转录调控机制。
近年来,空间转录组学 (Spatial Transcriptomics, ST) 作为单细胞 RNA 测序的空间延伸技术,已成为研究细胞亚型分布、互作关系和分子机制的重要工具。然而,受限于其高昂的设备和试剂成本,ST 技术在实际应用中仍面临普及难题。相较之下,WSIs 更具经济性和可及性,在临床应用上更为经济且易于获取。因此,如何借助深度学习手段,从 WSIs 中低成本重建 ST 图谱,成为一个备受关注的研究方向。
现有方法多将 ST 预测问题视为传统的回归问题,使用单级图像 - 标签对进行训练。这使得它们只能对最大放大倍数图像的基因表达关系进行建模,浪费了 WSIs 固有的多尺度信息。
基于这一问题,中国浙江大学的林兰芬教授研究团队联合浙江杭州之江实验室以及日本立命馆大学共同提出了 M2OST,这是一种多对一回归 Transformer 模型,旨在利用不同层次的病理图像共同预测基因表达。通过整合 WSIs 中的采样点视觉信息和多尺度特征,该模型能够生成更准确的 ST 图谱。此外,研究团队还将多对一的多层特征提取过程解耦为层内特征提取和跨层特征提取,在不影响模型性能的情况下大大降低了计算成本,优化了计算效率。
相关成果以「M2OST: Many-to-one Regression for Predicting Spatial Transcriptomics from Digital Pathology Images」为题,入选 AAAI 2025。
研究亮点:
- 将 ST 预测问题概念化为多对一建模问题,利用分层结构 WSIs 中嵌入的多尺度信息和点间特征,联合预测 ST 图谱
- 提出基于多对一回归的 Transformer 模型 M2OST,对不同序列长度的输入集具有鲁棒性
- 将 M2OST 中的多尺度特征提取过程解耦为层内特征提取和跨层特征提取,在不影响模型性能的情况下显著提高了计算效率
- 对提出的 M2OST 方法进行了全面的实验,并在 3 个公开的 ST 数据集上证明了其有效性

论文地址:https://arxiv.org/abs/2409.15092
关注「HyperAl超神经」公众号,后台回复「M2OST」获取完整 PDF
开源项目「awesome-ai4s」汇集了 200 余篇 AI4S 论文解读,并提供海量数据集与工具:
https://github.com/hyperai/awesome-ai4s
数据集:使用 3 个 ST 数据集证明其有效性
研究团队使用了 3 个公开的 ST 数据集来评估所提出的 M2OST 模型的性能:
*人类乳腺癌数据集 (HBC):包含 68 个 WSI 中的 30,612 个点位,每个点位至多有 26,949 个不同的基因。该数据集中的点直径为 100μm,以 200μm 的中心距排列成网格。
*人类阳性乳腺肿瘤数据集 (HER2):由 36 个病理图像和 13,594 个点位组成,每个点位包含 15,045 个已记录的基因表达数据。该数据集中的 ST 数据每个捕获点之间的中心距为 200μm,每个点的直径为 100μm。
*人类皮肤鳞状细胞癌数据集 (cSCC):包括 12 个 WSI 和 8,671 个点位。该数据集中的每个点位都对 16,959 个基因进行了分析。所有点的直径为 110μm,排列成中矩形点阵,中心距为 150μm。
M2OST 模型:多对一回归结构,多层次病理图像共同预测基因表达
近年来,从全切片病理图像 (WSIs) 中预测空间转录组 (ST) 图谱成为当前数字病理学领域中的研究热点。早期方法如 ST-Net 和 DeepSpaCE 基于卷积神经网络 (CNN) 进行图像块级别的 ST 预测。近期发布的双模态嵌入框架 BLEEP 引入对比学习策略,将 WSI 图像块特征与 ST 点嵌入对齐,并引入 K 近邻算法缓解推理阶段的批次效应问题。
随着基于 Transformer 的模型兴起,其性能已超越传统 CNN。深度学习模型 HisToGene 首次将 Transformer 引入基因表达预测,实现玻片级建模,提升了效率但仍受限于计算资源。Hist2ST 模型在此基础上融合 CNN、Transformer 与图神经网络,进一步捕捉长距离依赖,但其复杂的模型结构也导致过拟合风险上升。
与主流关注采样点间相关性的思路不同,基于分层图像特征提取的方法 iStar,强调采样点内的基因表达仅与其对应的图像块区域相关,采用预训练的 HIPT 进行特征提取,并通过 MLP 映射至表达值,性能优越,但由于特征不可学习,仍存在进一步优化空间。
研究团队受此启发,M2OST 同样采用了图像块级方案,一次预测一个采样点,确保每个预测的独立性与准确性。研究团队还进一步拓展了 iStar 的思路,设计了一套可学习的多尺度特征提取与融合模块,通过对局部区域的精细建模和跨尺度信息整合,提升模型在复杂组织结构下的预测能力。
如下图所示,来自不同全切片病理图像 (WSIs) 层级的 3 个图像块序列被输入到模型中,以共同预测相应位点的基因表达。
在接收到来自 3 个不同层次的病理图像块后,首先,M2OST 会将它们送入可变形图像块嵌入层 (DPE),以实现自适应 token 生成。DPE 不仅能从每张图像中提取基础病理图像块,还能在高层次的病理图像中引入更大尺寸的图像块,从而捕捉更广泛的上下文信息。
同时,DPE 通过生成细粒度的点内 token 和粗粒度的周围 token,以强化模型对采样点中心区域特征的关注,从而在多对一的建模过程中突出采样点间特征 (inter-spot features),为后续的表达预测提供更精细、结构化的特征表示。
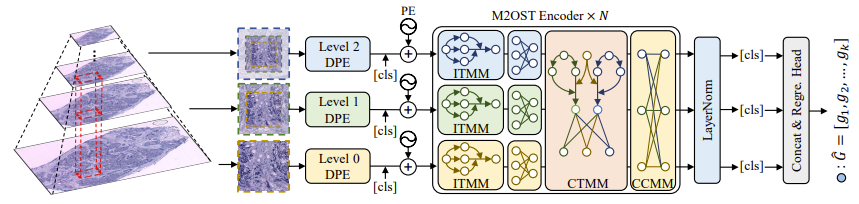
M2OST 模型示意图
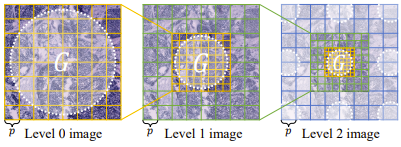
在 M2OST 中使用的 DPE
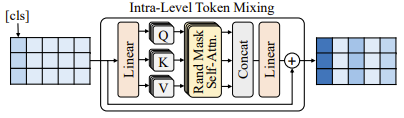
而后,在每个序列中添加 cls token,并如图中 PE 所示,引入可学习的位置编码,M2OST 使用内层 token 混合模块 (ITMM) 对每个序列进行层内特征提取。ITMM 基于 Vision Transformer 架构构建,并引入随机掩码自注意力机制 (Rand Mask Self-Attn),以增强模型在图像建模过程中的泛化能力。
在层内特征提取完成后,M2OST 引入跨层 token 混合模块 (CTMM),用于促进多层序列之间的跨层信息交互。由于多尺度输入序列的长度存在差异,CTMM 通过引入全连接跨层注意力机制以避免直接融合造成的信息失真,同时保持每个尺度分支参数的相对独立性。随后,为了增强通道级的跨尺度信息交换能力,M2OST 在 CTMM 之后引入了跨层通道混合模块 (CCMM)。
CCMM 采用对序列长度不敏感的结构设计,CTMM 根据不同层之间的注意力相似度和可学习权重,动态整合跨尺度的上下文信息,输出同形状的多层序列。首先对每个层次的序列进行全局平均池化 (Global Avg Pooling),将其序列信息压缩为一个 token 表示,然后将不同层次的 token 组合在一起,并结合挤压激励机制 (Squeeze & Excitation) 计算跨层通道注意力分数。这些分数随后被映射回各自的输入序列,完成通道级的跨尺度信息交换。
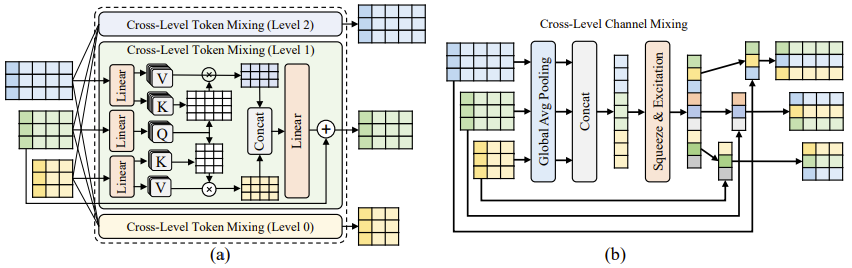
(a) CTMM 的网络结构。(b) CCMM 的网络结构。
该多尺度特征建模过程整体构成 M2OST 的编码器模块,并在整个网络中迭代 N 次,以逐步丰富空间转录组预测所需的多层次、高表达力的图像表征。最后,将 3 个 cls token 连接起来,送入线性回归头部进行 ST 点预测。
实验结果:多维度评估证明 M2OST 模型有效性
研究团队全面比较了 M2OST 与多种主流方法在多个数据集上的表现。实验结果如下表所示,M2OST 在更少的参数量和更少的 FLOPs 下,实现了更为优越的性能。与 ST-Net 相比,M2OST 的参数量减少了 0.40M,FLOPs 降低了 0.63G,而 M2OST 在 HER2+ 和 cSCC 数据集上的皮尔逊相关系数 (PCC) 分别提升了 1.16% 和 1.13%。
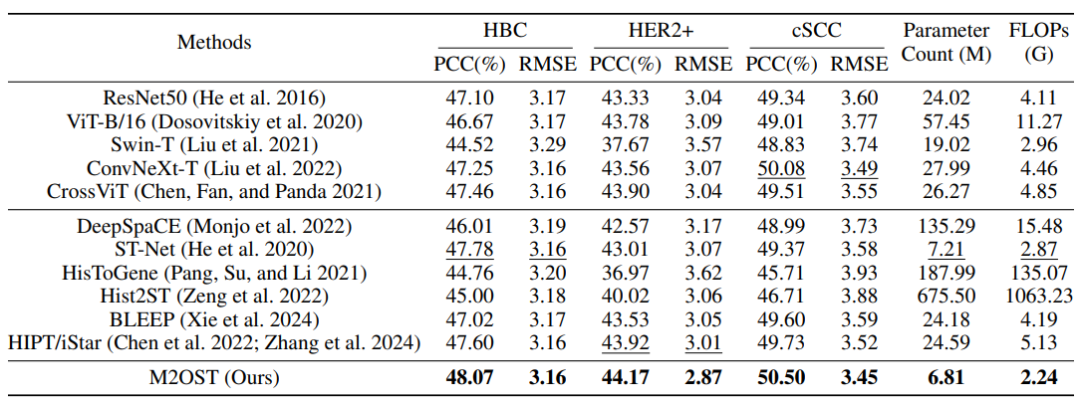
M2OST 与其他方法的比较实验结果
M2OST 与一对一多尺度方法的比较:
研究团队还将 M2OST 与普通的一对一多尺度方法进行了比较,如 CrossViT 和 HIPT/iStar。相较于标准 ViT,CrossViT 展现出更强的 ST 回归能力,证实了在该任务中整合多尺度信息具有显著优势。然而,CrossViT 在点内信息建模方面存在一定限制,其整体性能仍不及 M2OST。
此外,iStar 在 ST 预测准确性上表现出色,证明了 HIPT 架构在从 WSI 中提取多尺度特征方面的有效性。然而,为了节约计算成本,iStar 采用固定的 HIPT 权重来生成用于 ST 预测的 WSI 特征,限制了其特征提取能力。同时,在推理效率方面,iStar 的逐块、逐尺度的提取流程显著增加了处理时间。研究结果表明,当在相同的 GPU 内存限制下运行,M2OST 的推理速度比 iStar 快约 100 倍,且性能仍优于后者,充分展示了端到端训练在 ST 回归任务中的潜力和 M2OST 模型的有效性。
图像块级和玻片级 ST 方法的比较:
实验结果显示,玻片级方法在 3 个数据集上的表现普遍不及图像块级方法。尽管 Hist2ST 相较于 HisToGene 展现出更强性能,但其大量参数和高 FLOPs 使得这种性能的提升意义不大。与 ST-Net 等基线图像块级方法相比,Hist2ST 在 3 个数据集上的 PCC 分别降低 2.78%、2.99% 和 2.66%。这表明一个点的基因表达主要与其对应的组织区域相关,引入点间相关性并未显著提升预测准确性。尽管如此,玻片级方法在生成完整 ST 图谱方面仍具优有更高的效率,未来通过优化网络设计,仍有潜力实现具有竞争力的回归精度。
可视化分析:
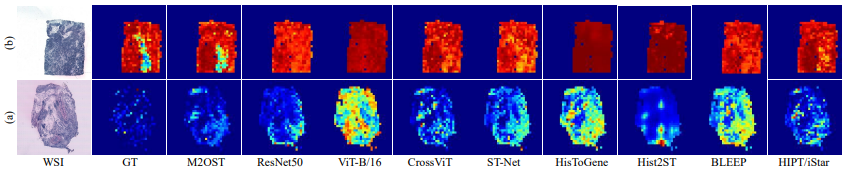
(a) 主成分分析 (PCA) 之后的空间转录组 (ST) 图谱的可视化结果。 (b) DDX5 基因空间分布的可视化结果。
研究团队对不同方法在 ST 图谱预测中的可视化结果进行了分析对比,结果显示玻片级方法(如 HisToGene 和 Hist2ST)通常能够生成更平滑的图谱,而图像块级方法则保留了更清晰的局部结构特征。
值得注意的是,M2OST 始终能够生成更准确的 ST 图谱,呈现出更高的预测精度。研究团队进一步对关键基因 DDX5 的表达进行了可视化,DDX5 通过激活 β - catenin 信号通路,在非小细胞癌细胞的增殖和肿瘤发生中起关键作用。结果表明 M2OST 在该基因的预测中表现最优,优于所有对比方法,验证了 M2OST 模型在单基因表达预测水平上的准确性。
空间转录组学的突破性进展与跨领域应用
空间转录组学作为连接细胞功能与组织结构的桥梁,能够解析单个细胞在时间和空间维度上的基因表达模式,并揭示细胞类群的空间位置及其生物学特征,正推动生物医学研究向更深层次发展。
在这一领域,2025 年 4 月,日本东京大学医科学研究所的研究团队开发了基于图像辅助的图对比学习进行空间转录组学分析的深度学习框架 STAIG。该框架能够整合基因表达、空间数据和组织学图像,且不需要对齐数据,从而克服了传统方法在消除批次效应和识别空间区域上的局限性。STAIG 通过自监督学习,从苏木精和伊红 (H&E) 染色图像中提取特征,无需依赖大规模数据集进行预训练。
在训练过程中,STAIG 动态调整图结构,并通过组织学图像选择性排除无关的负样本,减少了偏差。最终,STAIG 通过局部对比分析基因表达的共性,成功实现了批次整合,避免了手动坐标对齐的复杂性,显著减少了批次效应。研究表明,STAIG 在多个数据集上表现出色,特别是在空间区域识别方面,能够揭示肿瘤微环境中的详细基因和空间信息,展现出其解析空间生物学复杂性的重要潜力。
点击查看详细报道:无需预对齐即可消除批次效应,东京大学团队开发深度学习框架STAIG,揭示肿瘤微环境中的详细基因信息
与此同时,中国上海临港实验室魏武研究团队也在空间转录组学领域取得了显著进展。2024 年 11 月,团队在 Briefings in Bioinformatics 期刊上发表了题为「MCGAE: unraveling tumor invasion through integrated multimodal spatial transcriptomics」的研究论文。该研究开发了专为空间转录组数据分析设计的深度学习框架 MCGAE (Multi-View Contrastive Graph Autoencoder),该框架通过结合基因表达、空间坐标和图像特征,创建多模态、多视图的生物表征,显著提升了空间域识别的准确性。在肿瘤数据中展现了对肿瘤区域的精确识别与分子调控特征的深度解析,为复杂组织、疾病机制研究和药物靶点发现提供了强有力的工具。
论文原文:
https://academic.oup.com/bib/article-pdf/26/1/bbae608/60786360/bbae608.pdf
此外,空间转录组学在农业领域的应用也展现出巨大潜力。2025 年 4 月,北京大学现代农业研究院的研究团队在 Genome Biology 上发表了一项题为「Spatiotemporal tranomics reveals key gene regulation for grain yield and quality in wheat」的重要研究,利用空间转录组技术构建了小麦籽粒发育早期不同时间段的高分辨率基因表达图谱,揭示了小麦籽粒发育过程中的基因表达特征。这一研究不仅为小麦的分子设计育种与产量提高提供了重要的理论支持,也为全球粮食安全提供了有力保障。
论文原文:
https://www.biorxiv.org/content/biorxiv/early/2024/06/03/2024.06.02.596756.full.pdf
未来,随着空间转录组数据的不断积累和数字病理图像获取手段的持续优化,人工智能与组学技术的深度融合将推动深度学习模型在多种组织类型和疾病背景中的广泛应用,助力精准医疗的发展。M2OST 的提出为构建高效、低成本、高精度的空间基因表达预测框架奠定了坚实基础,预示着人工智能与多组学数据融合分析在生物医学领域的深远前景。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢