DRUGAI
今天为大家介绍的是来自纽约大学Aristotelis Tsirigos团队的一篇论文。自监督学习(SSL)可以自动从未标注的苏木精-伊红染色的全扫描切片(WSIs)中提取和解释病理组织学特征。我们在来自癌症基因组图谱的435个结肠腺癌WSI上训练了一个SSL Barlow Twins编码器,用于从小型图像块中提取特征。利用Leiden社区检测方法将这些特征分组为组织形态学表型簇(histomorphological phenotype clusters,HPCs)。一项独立的临床试验(N = 1213 WSIs)确认了HPC的可重复性和对总体生存率的预测能力。这种无偏的图谱有47个HPCs,展示出独特且共享的临床显著组织形态学特征,突出了组织类型、数量和结构,特别是在肿瘤基质的背景下。通过对这些HPCs的深入分析,包括免疫景观和基因集富集分析,以及与临床结果的关联,作者阐明了影响生存和对标准辅助化疗和实验治疗反应的因素。对HPCs的进一步探索可能会揭示额外的见解,并有助于结肠癌患者的决策和个性化治疗。

传统上,结肠癌的诊断是由病理学家通过苏木精和伊红染色(H&E)切片对切除标本进行显微镜检查来确认的。对每位患者,根据包括肿瘤-淋巴结-转移(TNM)分类和其他生物标志物(biomarker)在内的临床病理特征进行风险评估的指南,通过多学科会议制定个性化治疗策略。然而,由于人口老龄化和生物标志物研究的增加,诊断和预测结肠癌患者的预后可能很耗时,或者复杂且资源需求大,特别是在整合突变变异筛查时。
在现代数字病理学中,将H&E切片扫描成高分辨率全扫描切片(WSIs)使深度学习(DL)的应用成为可能。特别是深度卷积神经网络最初通过最小化评价者间的分歧和工作量,使诊断过程受益。在结直肠癌中,有监督的DL模型也显示出预测分子通路(即突变密度、微卫星不稳定性[MSI]、染色体不稳定性)和关键突变如BRAF和KRAS的能力。DL甚至在预测患者生存等复杂预后结果方面具有令人惊讶的潜力。此外,将多组学数据与相关的H&E切片整合,即多模态数据整合,改善了大多数癌症类型总体生存率(OS)的预后预测。
以往的深度学习研究主要集中在训练模型,在可能广泛且耗时的人工派生标注(幻灯片或像素级别)的监督下从WSIs中提取特征,即有监督学习。另一方面,自监督学习(SSL)因其能够自动从未标记数据中捕获图像特征的能力而受到越来越多的关注。SSL模型的应用,包括通用基础模型,在各种下游癌症分类、生存和分子表型预测任务中展示了比传统有监督学习模型更优越的性能。Barlow Twins,一种设计用于学习非冗余图像特征的SSL模型,相比其他SSL学习模型(如对比学习模型)具有几个优势,包括不需要大批量大小,也不需要网络"孪生体"之间的不对称性。
使用多机构TCGA数据集对WSI特征进行自监督学习
作者使用仅来自TCGA结肠腺癌(TCGA-COAD)集的数据训练了自监督算法,消除了对病理学家标注的需求(图1)。从TCGA-COAD数据集获得的共435个WSIs(428名患者)(首先被分割成较小的图像块(224×224像素),也称为图像瓦片(tile),放大倍数为10倍(图1a)。为了识别这些图像块上的特征,作者使用随机子集的瓦片(N = 250,000个图像瓦片)训练了SSL Barlow Twins特征提取器(图1a)
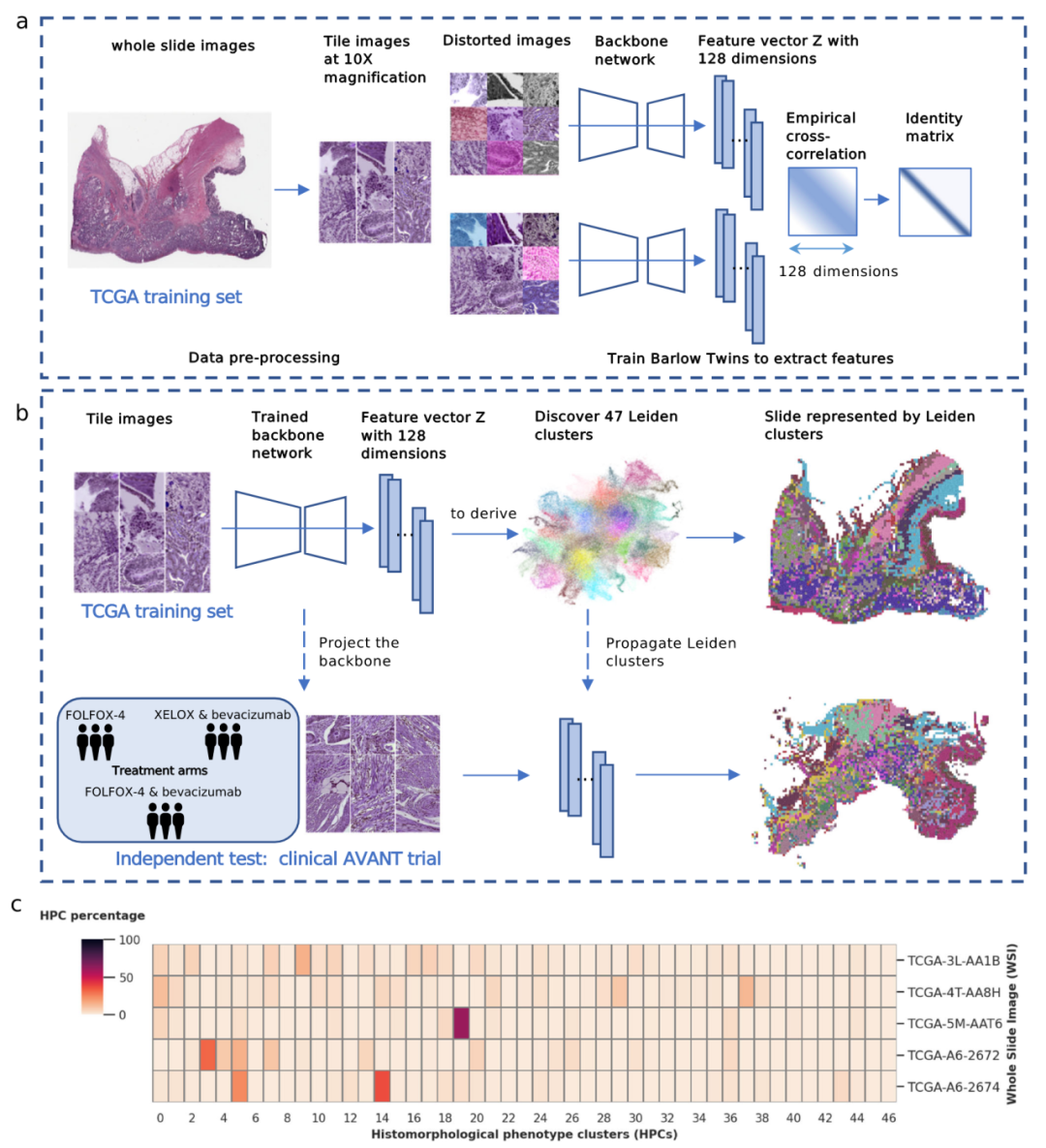
图 1
Barlow Twins的训练目标函数是评估两个相同骨干网络嵌入(特征向量z)之间的交叉相关矩阵,这两个网络被输入同一批图像瓦片的变形版本。目标函数通过最小化交叉相关矩阵与单位矩阵之间的偏差来优化。这增加了变形样本版本的z向量嵌入之间的相似性,同时减少了这些向量的各个组件之间的冗余性。因此,每个瓦片被描述为一个128个提取特征的向量,随后可用于按相似性将瓦片分组成簇。
通过社区检测构建无偏的组织学模式图谱
作者应用Leiden社区检测算法来导出HPCs,即具有相似组织学模式的簇(图1b)。该过程首先通过将训练好的Barlow Twins投影到整个TCGA-COAD数据集上开始,为每个图像瓦片提取128维特征表示。随后,作者利用Leiden社区检测算法处理从这些瓦片向量表示构建的最近邻图(图1b)。具有相似向量表示的瓦片被聚类成一个组,并分配特定的HPC ID号。Leiden配置的优化是通过无监督过程实现的,最终识别出总共47个HPCs,这些HPCs在降维图(UMAP图)中可视化表示(图2a)。
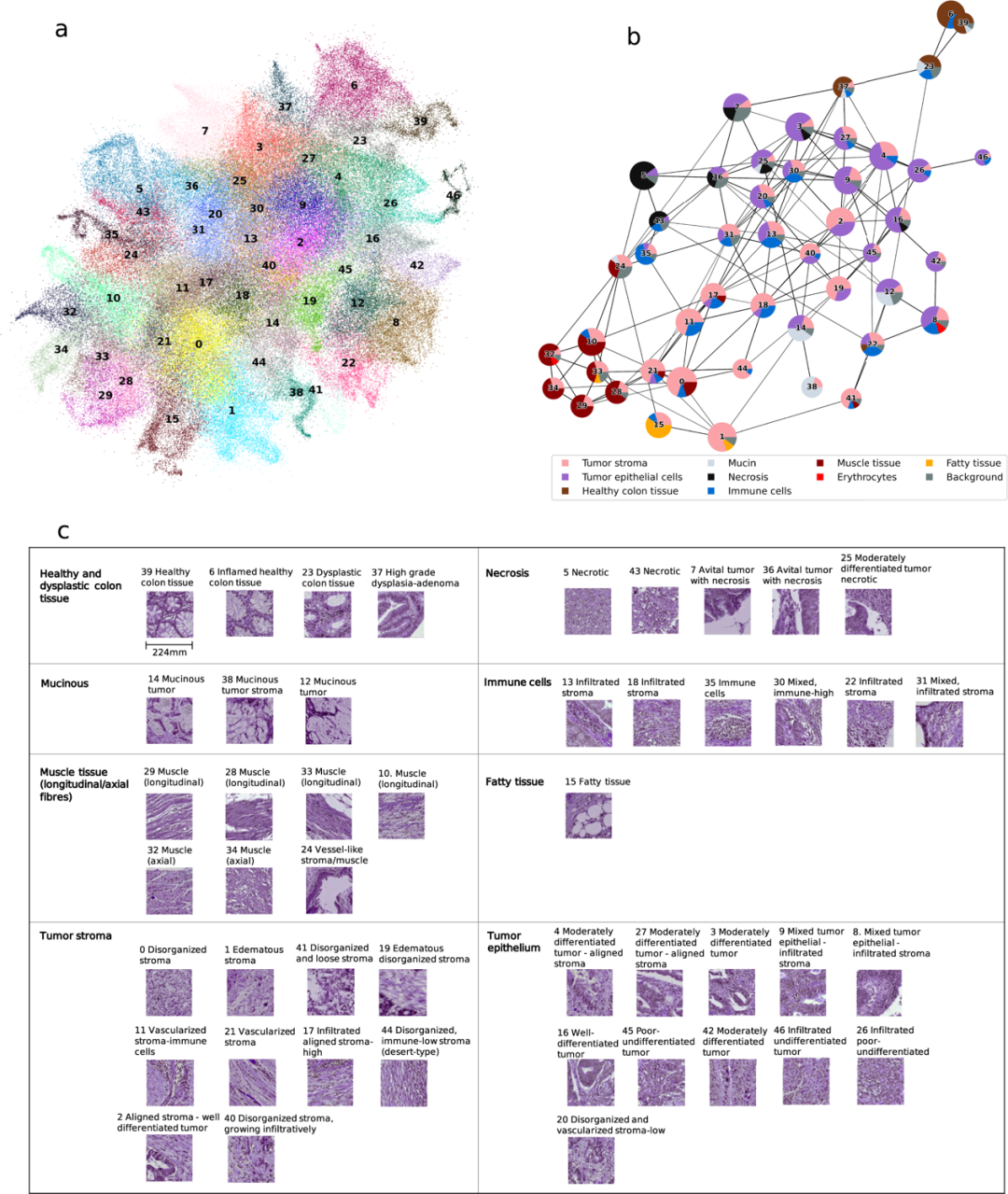
图 2
作为外部数据集,作者分析了共1213名结肠癌患者的诊断病理H&E WSIs(每位患者一个WSI),这是临床AVANT试验的一个子集。作者利用优化的SSL Barlow Twins模型生成未见过的AVANT WSI瓦片的嵌入。未见过的AVANT中识别的HPCs的分配是使用K-最近邻方法实现的。AVANT数据中每个瓦片的HPC标签是根据TCGA训练集中其K个最近邻(K = 250)的多数投票确定的(图1b)。因此,作者获得了WSIs的综合视觉表示,其中WSI瓦片按其相应的HPC着色(图1b)。此外,作者能够使用从HPCs导出的构成数据捕获WSIs的特征和异质性,即WSI上每个HPC覆盖区域的百分比,从而促进下游分析和建模(图1c)。
组织病理学评估和HPCs的特征描述
每个HPC在TCGA内随机选择的32个瓦片集上进行了组织病理学分析,由两名病理学家(ASLPC和JHJMvK)和一名研究员(MP)独立评估。在瓦片上观察到的组织类型,特别关注肿瘤上皮、肿瘤基质和免疫细胞进行了描述。其他独特的组织病理学特征或模式,如肿瘤分化程度和基质组织结构,也被记录下来。所有存在的组织类型都以百分比计分并使用饼图显示(图2b)。作者使用基于分区的图抽象(PAGA)绘制了47个HPCs的相互联系,饼图反映了它们的组织构成(图2b)。有趣的是,根据组织构成的相似性、HPCs在PAGA图中的相互连接性和拓扑结构,可以观察到明显的更大的簇群或"超级簇"(图2b)。
总共,作者识别了八个超级簇:(1) 健康和异型性结肠组织,(2) 坏死,(3) 黏液区域,(4) 免疫细胞,(5) 肌肉组织(纵向/轴向),(6) 脂肪组织,(7) 肿瘤基质,和 (8) 肿瘤上皮,无特定顺序,由图2c中显示的HPC组形成。在每个指定的超级簇内的HPCs中注意到了共同的组织病理学特征,而包含跨越多个超级簇的各种组织类型的HPCs通常位于它们的交叉处。例如,不仅含有肿瘤上皮而且含有黏液性肿瘤的HPC 12,在PAGA图中位于属于黏液性肿瘤和肿瘤上皮两个超级簇的HPCs之间。此外,以异型性结肠组织为特征的HPC 23,在健康结肠组织HPC 39和含有肿瘤上皮的超级簇HPCs(如HPCs 4、26、46)之间形成了桥梁,暗示潜在的时间病理发生。总之,派生的HPCs显示出独特的组织病理学特征。此外,基于提取的特征在UMAP和PAGA图上位置接近的HPCs展示了共同特征,暗示潜在的模式关系、混合表型或病理轨迹。
评估TCGA和AVANT队列内部和跨队列的HPC一致性
尽管SSL方法最近已应用于组织病理学,但通常没有对这些方法在数据集内部和跨数据集发现的组织学模式一致性进行系统分析。在这里,作者通过纳入几种定性和定量评估来解决这一潜在陷阱。
首先,进行了定性评估,以评估TCGA-COAD中派生的47个HPCs的簇内和簇间异质性。基于从TCGA-COAD中每个HPC随机选择的32个瓦片,三位专家(ASLPC、JHJMvK和MP)通过比较组织类型数量和结构独立评估了每个HPC(上述组织病理学评估程序)。总体而言,所有评价者达成一般共识,认为存在显著的簇内形态相似性和47个HPCs之间的显著多样性(图3 [a–i]),尽管表型相似性在HPCs之间有所不同,暗示有些HPCs可能比其他HPCs更相似。
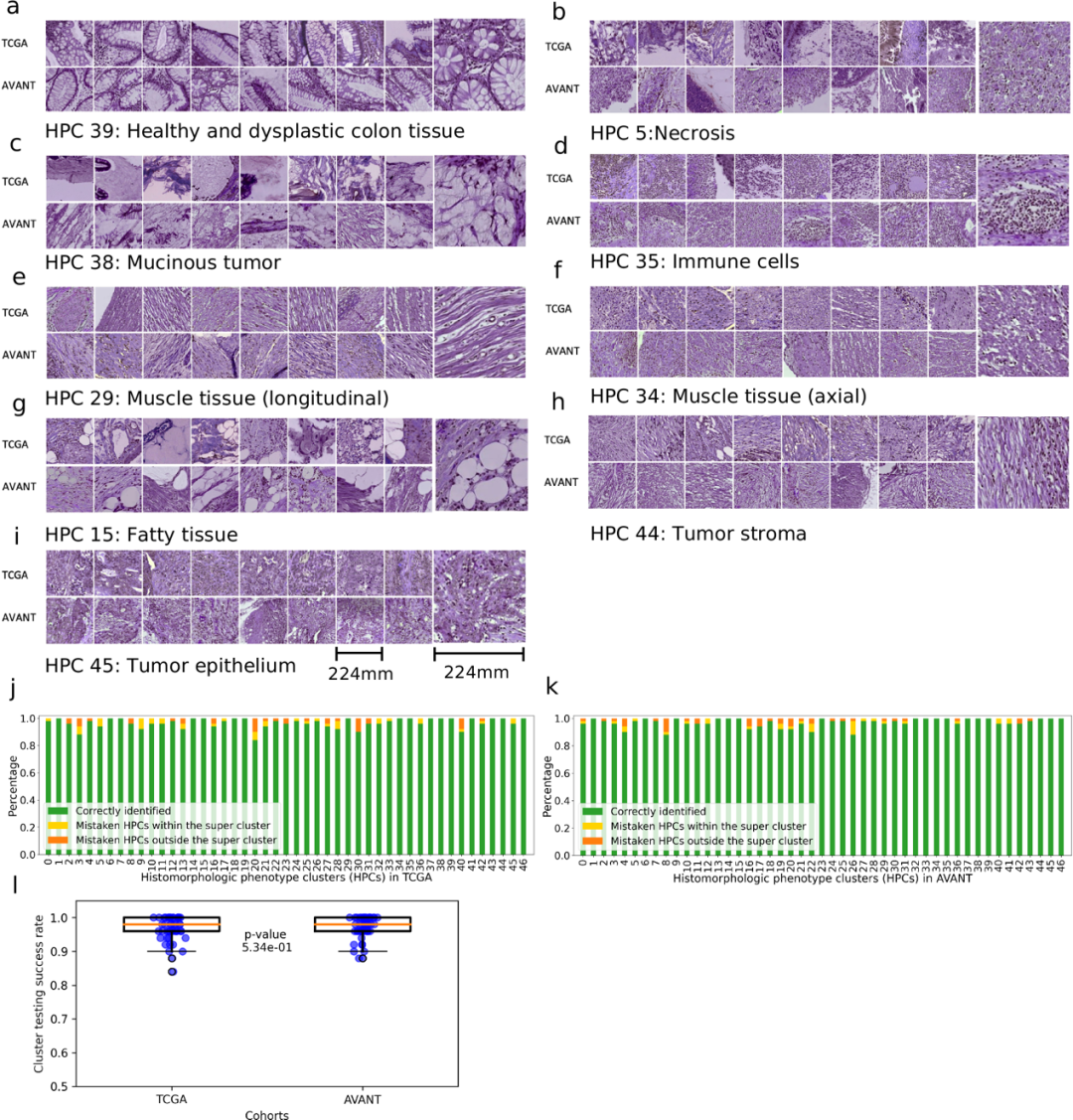
图 3
为了更深入地探讨簇内和簇间异质性,作者分别在TCGA和AVANT瓦片内进行了定量客观盲测。这是为了确定每个HPC识别的形态学模式是否也能被人类专家识别。在这个测试中,评估者(MP)被展示了三组图像瓦片,每组包含五个瓦片。两组来自同一HPC,第三组来自随机选择的另一个HPC,也称为"异常HPC"。评估者需要识别"异常HPC"。47个HPCs中的每一个都进行了50次测试,以确定成功率。在TCGA内,作者发现47个HPCs中有17个达到了100%的识别率,而其余30个HPCs的正确识别率在84%到98%之间。同样,在AVANT内,47个HPCs中有17个达到了完美准确率,而其余的准确率在88%到98%之间(图3j)。一般来说,在PAGA图中彼此靠近或属于同一超级簇的HPCs更容易被错误分配。
为了评估从TCGA转移形态学模式到外部AVANT测试集的有效性,三位专家(ASLPC、JHJMvK和MP)独立审查了从TCGA-COAD子集随机选择的32个瓦片集和从AVANT试验随机选择的另外32个瓦片。这种定性比较得出结论,在各自的HPCs内,TCGA和AVANT瓦片之间存在显著的相似性(图3 [a–i])。在比较TCGA和AVANT的客观测试结果时,作者发现两个数据集之间错误分类的HPCs有80%重叠,正确分类的HPCs有65%重叠(图3j, k)。这些结果表明,从训练集提取的稳健形态学特征可以有效地转移到独立的未见过的测试集。
编译|黄海涛
审稿|王梓旭
参考资料
Liu, B., Polack, M., Coudray, N., Claudio Quiros, A., Sakellaropoulos, T., Le, H., ... & Tsirigos, A. (2025). Self-supervised learning reveals clinically relevant histomorphological patterns for therapeutic strategies in colon cancer. Nature Communications, 16(1), 2328.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢