DRUGAI
今天为大家介绍的是来自伦敦癌症研究所癌症生物学系Chris Bakal教授带领团队发表的一篇论文。这项研究介绍了一个名为MorphoMIL的创新计算方法,它结合了几何深度学习(geometric deep learning)和基于注意力的多实例学习(attention-based multiple-instance learning),用于分析细胞及其细胞核的三维形态特征。研究团队采用3D点云数据作为输入,不仅能在单个细胞层面捕捉形态特征,还能分析整个细胞群体的特征,同时考虑了细胞群体中个体差异(表型异质性)。他们对超过95,000个黑色素瘤细胞进行了实验,这些细胞分别接受了具有临床意义的药物处理、基因改造,以及影响细胞内部支架结构的处理。该方法不仅能准确预测药物对细胞的影响和细胞状态,还能发现药物带来的细微形态变化,找出与细胞信号活动相关的关键形状特征,并帮助我们理解为什么同一群细胞会表现出不同状态。MorphoMIL不仅表现出色,而且适用于各种不同类型的数据集,这为未来在药物研发中进行大规模、高效的细胞形态分析开辟了新途径。

细胞的形状是如何形成的?这要从细胞内部成分说起。细胞中的蛋白质、代谢物和脂质不断与周围环境互动,最终决定了细胞的形状。细胞形态的异常往往预示着疾病的发生,因此研究细胞形状可以帮助我们了解细胞的状态。传统研究主要关注细胞的二维形态,使用面积、周长等简单的几何特征来描述细胞。但这种方法存在明显局限:首先,它依赖预设的形状测量标准;其次,真实生物体内的细胞是三维的,仅用二维图像难以完整描述细胞形态。随着技术进步,特别是3D显微技术的发展(如斜面显微镜技术),科学家们终于能够观察到细胞的真实三维结构。然而,如何定量分析这些3D形态数据仍然是一个挑战。为此,研究团队开发了一个创新的分析框架——MorphoMIL。这个框架采用了点云数据(即在3D空间中分散的点的集合)来表示细胞形状,并结合了几何深度学习和基于注意力的多实例学习技术。MorphoMIL的应用效果令人惊喜:它不仅能够通过细胞的3D形状预测重要的生化活性(如细胞信号通路MEK-ERK的活性),还能通过定量形态特征(quantitative morphological signatures, QMS)预测基因功能和蛋白质之间的相互作用。这些发现表明,几何深度学习不仅能够识别3D细胞形状的特征,当与注意力机制的多实例学习结合时,还能识别不同处理条件下的特征表型,预测细胞状态,为药物研发提供新的研究思路。
这项研究取得了四个重要突破:
几何深度学习技术能够在各种实验条件下,准确捕捉细胞的3D形状特征;
通过多实例学习技术分析细胞形态的变化规律,成功预测药物的处理效果;
通过分析MIL的注意力权重,研究人员发现了药物处理会导致细胞产生特定的形态特征;
研究团队开发的3D细胞形状分析模型,成功将细胞的形态特征与其内部的信号传导状态和蛋白质互作联系起来。
一种使用GDL对三维细胞形状进行分析的自动化方法
研究团队开发了一种创新的方法,用于自动分析细胞的3D形态特征。他们首先使用一种特殊的显微镜技术——阶段扫描斜面显微镜(stage-scanning OPM)对实验样本进行观察。如图1A所示,研究人员将超过65,000个WM266-4黑色素瘤细胞培养在模拟人体组织环境的胶原蛋白水凝胶中。这些细胞被特殊处理,能够发出荧光信号来显示细胞膜、细胞核的位置,以及细胞内ERK激酶的活动情况。
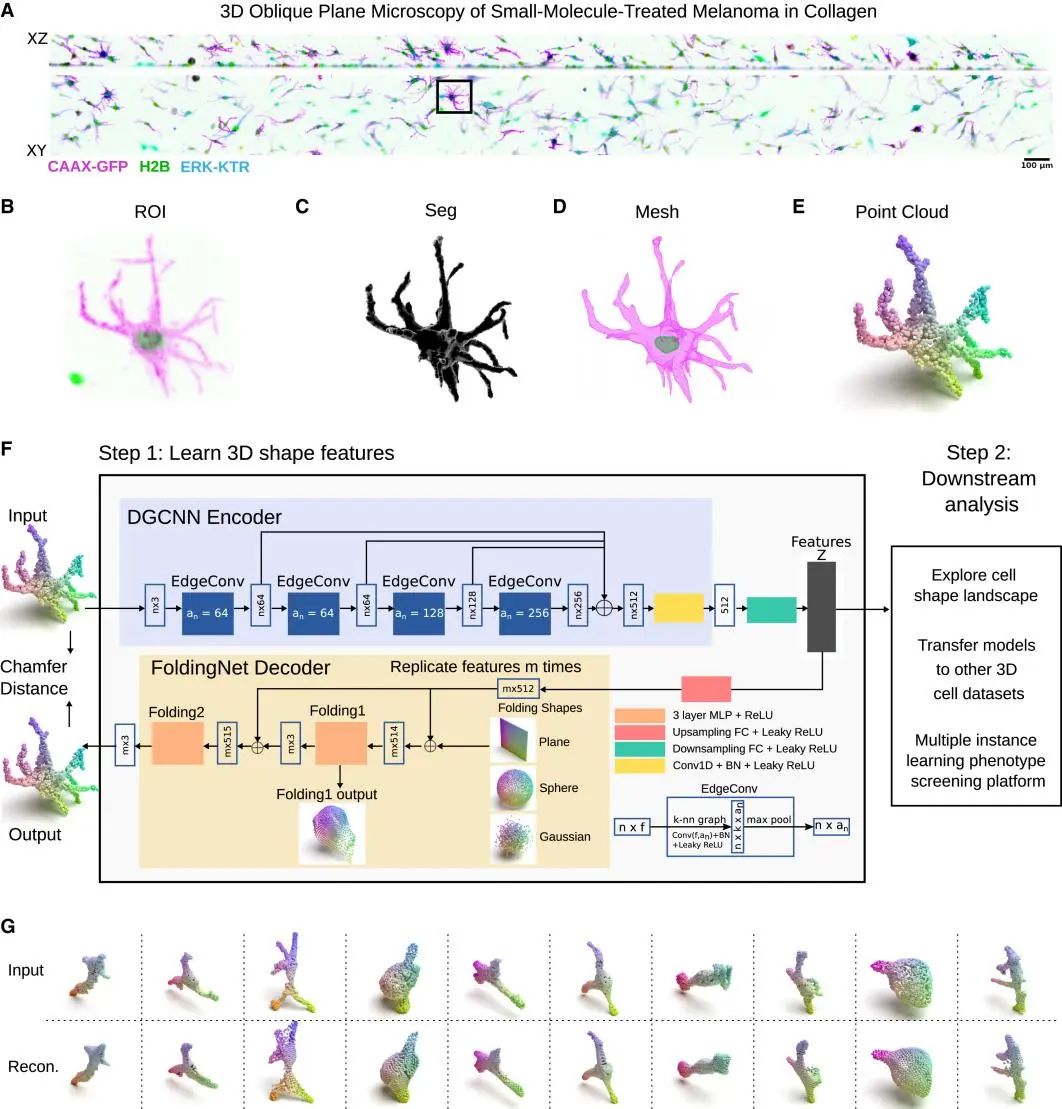
图 1
为了研究不同条件下细胞的形态变化,研究人员使用了多种临床相关的药物处理细胞,这些药物可以影响细胞骨架的组织和细胞信号通路。如图1B-E所示,研究团队开发了一套完整的数据处理流程:首先对细胞和细胞核进行分割(图1B, C),然后将3D图像数据转换为网格结构(图1D),最后生成点云表示(图1E)。
为了准确量化单个细胞的3D形状特征,研究人员开发了一个智能的计算模型——动态FoldingNet(DFN)。如图1F所示,这个模型通过结合动态图卷积神经网络(DGCNN)技术,能够同时学习细胞的局部特征(如突起等)和整体形态特征。如图1G所示,DFN通过“自编码器”的方式工作:先将输入的点云数据压缩成更简单的特征表示,然后尝试从一个基本形状重建原始点云。这种方法使得研究人员能够提取出细胞3D形状的关键特征,为后续分析奠定基础。
细胞3D形态分布反映其所处环境以及受到的干扰
研究人员首先使用UMAP技术(一种数据可视化工具)将复杂的3D细胞形态数据简化成二维图像(图2A)。这个可视化结果显示,细胞形态的主要差异体现在两个方面:细胞的大小(图2B)和形状的规则程度(偏心率)(图2C)。较小且圆形的细胞会聚集在一起(图2D, E),而较大且形状不规则的细胞则会出现在图的其他区域。
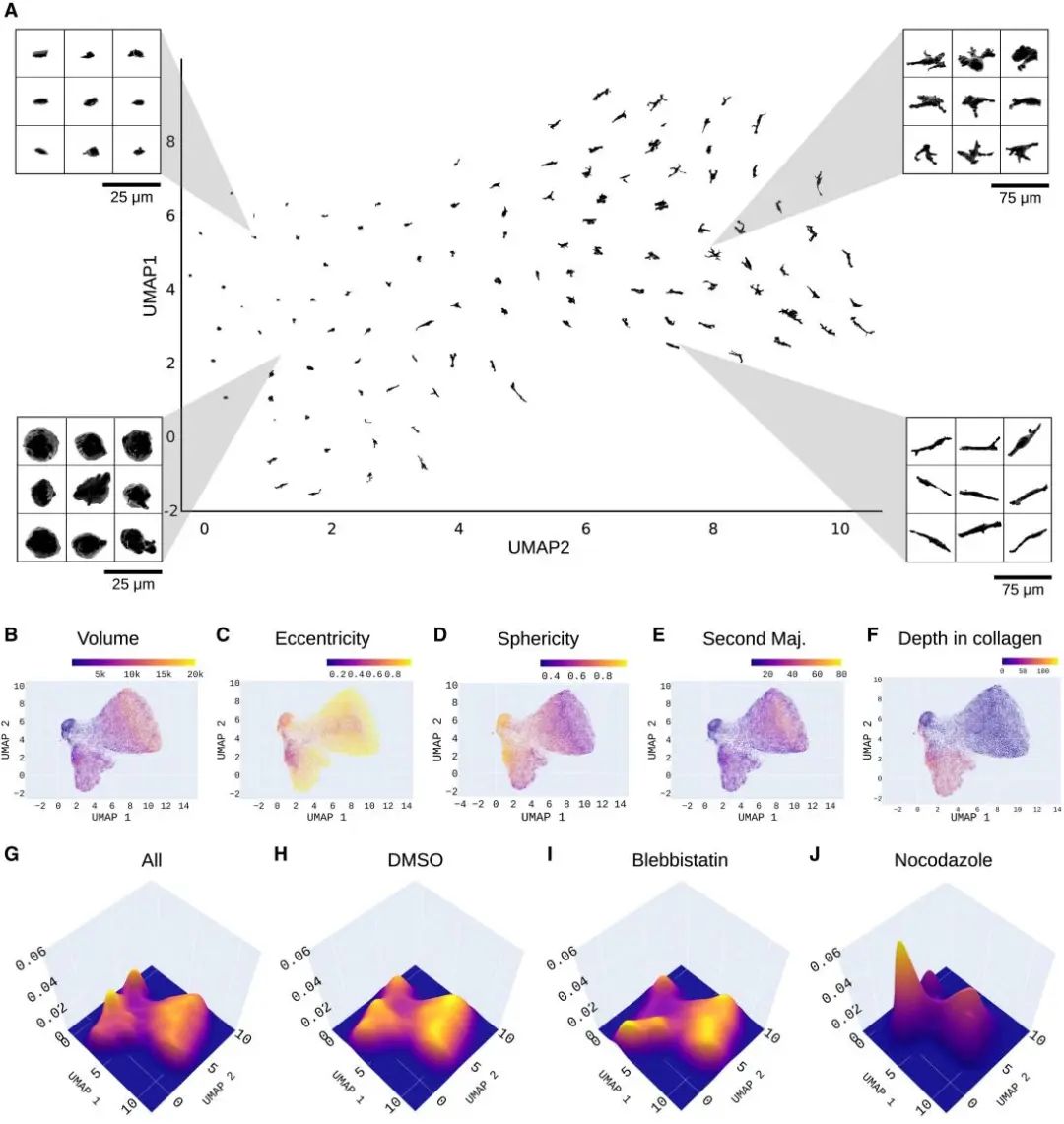
图 2
研究还发现,细胞在胶原蛋白基质中的位置会显著影响其形态。如图2F所示,研究人员根据细胞核心距离盖玻片的距离,将细胞分为两类:距离小于7微米的“近端”细胞和距离大于7微米的“远端”细胞。这个发现表明,3D细胞形态能够反映细胞所处的物理环境。更有趣的是,不同药物处理会导致细胞呈现不同的形态特征。如图2G-J所示,研究人员比较了对照组(DMSO处理)和不同药物处理组的细胞形态分布。例如,blebbistatin处理的细胞往往会形成更多突起且形状不规则(图2I),而nocodazole处理的细胞则更趋向于圆形且突起较少(图2J)。
这项研究的一个重要发现是:药物处理通常不会让细胞产生全新的形状,而是改变了细胞群体中不同形态的分布比例。这个发现提示我们,在研究药物如何影响细胞形态时,需要同时考虑细胞群体内部的差异和不同细胞群体之间的相似性。这种深入理解有助于我们更好地评估药物的作用机制和效果。
3D细胞形状反映干扰程度
研究团队开发的3D细胞形态分析方法究竟准确吗?为了回答这个问题,他们设计了一系列验证实验。首先,研究人员使用一种叫做支持向量机(Support Vector Machine, SVM)的人工智能算法来识别不同药物处理的细胞。他们采用两个指标来评估模型的表现:平衡准确率(衡量模型正确分类的比例)和AUC值(反映模型区分不同类别的能力)。
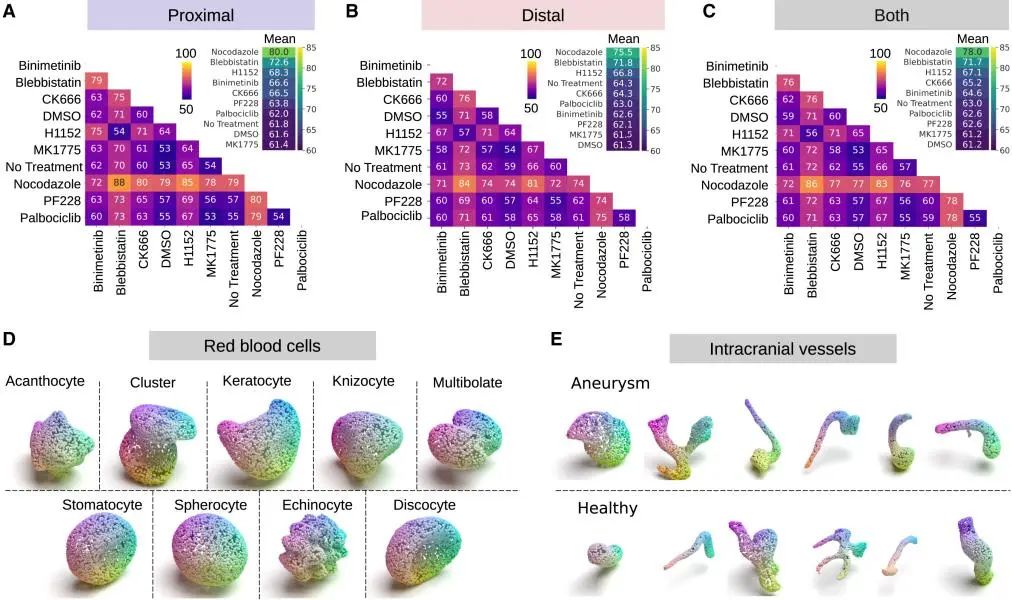
图 3
如图3A和3B所示,模型在识别靠近盖玻片的近端细胞时表现最好。特别是对于三种药物处理:破坏微管动态的nocodazole、抑制肌球蛋白-2的blebbistatin,以及抑制ROCK蛋白的H1152,模型表现出极高的识别准确率。图3C全面展示了模型在不同条件下的分类效果。研究还发现一个有趣的现象:虽然单独使用细胞形状或细胞核形状的特征都能进行预测,但结合两者的信息效果最佳。这说明细胞的整体形态和细胞核的变化都携带着重要的生物学信息。更令人惊讶的是,仅凭细胞的3D形状特征,模型就能以84.9%的高准确率判断出细胞在胶原蛋白基质中的位置(是处于15-60微米还是大于70微米的深度)。这个发现表明,细胞的形态特征与其所处的微环境密切相关,为我们理解细胞如何适应和响应周围环境提供了新的视角。研究团队首先验证了他们开发的DFN方法的普适性。他们不仅在自己的黑色素瘤细胞数据上进行了测试,还将方法应用到了两个公开数据集:825个3D红细胞图像(图3D)和1,908个3D脑血管图像(图3E)。结果显示,DFN在所有数据集上都优于其他方法,证明了这种方法具有良好的通用性。
一个基于注意力的多实例框架
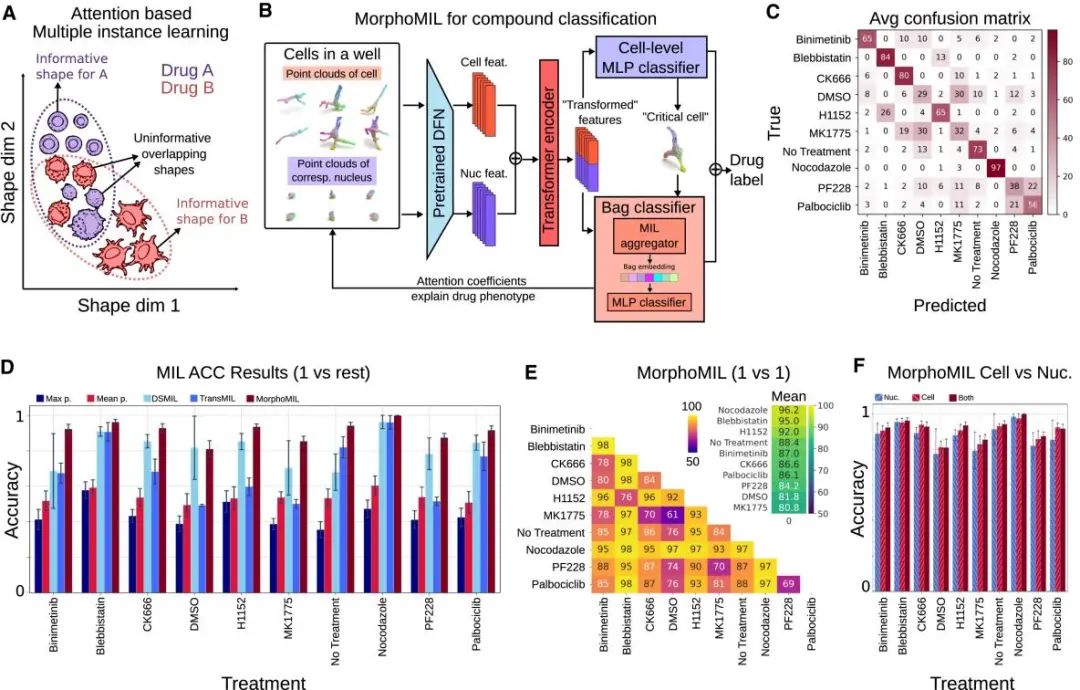
图 4
然而,研究中遇到了一个关键问题:即使是相同的药物处理,不同细胞的形态反应也可能大不相同。为了解决这个问题,研究团队开发了一个创新的分析框架——MorphoMIL(图4B)。这个框架就像一个智能的药物侦探,它不仅能观察单个细胞的变化,还能通过分析细胞群体的整体特征来识别药物处理的效果。如图4C-E所示,它能以极高的准确率(最高达99.3%)识别不同的药物处理。特别值得注意的是,即使是作用机制相似的药物,如blebbistatin(抑制非肌肉肌球蛋白-2)和H1152(抑制ROCK),MorphoMIL也能准确区分(准确率76.2%)。这说明细胞的3D形态变化能够反映出药物作用的细微差异。研究还发现了一个有趣的现象:在大多数情况下,同时分析细胞和细胞核的形态特征能获得最佳的识别效果(图4F)。这就像观察犯罪现场时,需要同时考虑多个线索才能得出最准确的结论。
MorphoMIL可推广到其他细胞类型和成像方式
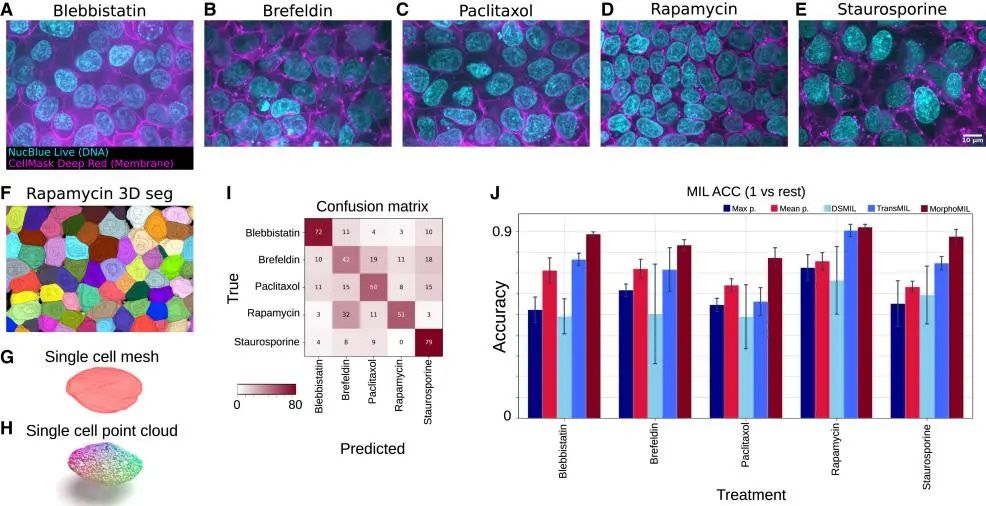
图 5
为了验证MorphoMIL方法的通用性,研究团队选择了一个具有挑战性的测试对象——人诱导多能干细胞(hiPSC)。这种细胞具有特殊的分化潜能,被广泛应用于药物开发和疾病建模研究。如图5所示,研究人员分析了五种不同药物(blebbistatin、brefeldin、paclitaxel、rapamycin和staurosporine)处理的hiPSC细胞(图5A-E)。他们首先使用CellPose技术对3D图像中的单个细胞进行分割(图5F),然后创建网格模型(图5G)并提取点云数据(图5H)。有趣的是,即使是在完全不同的细胞类型上,MorphoMIL仍然展现出优异的分类性能(图5I和5J)。
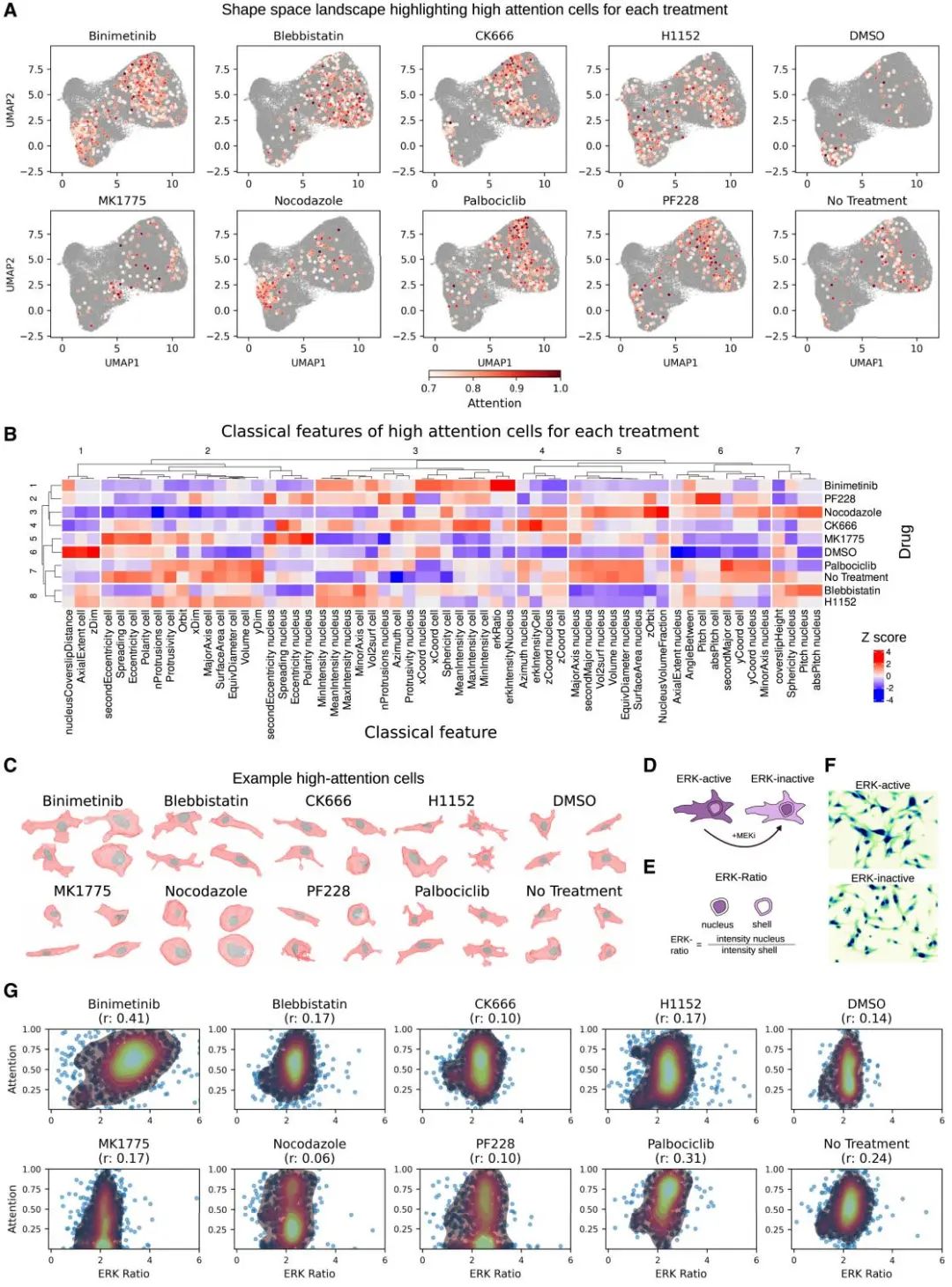
图 6
MorphoMIL的一个独特优势是它能够“解释”自己的决策过程。通过分析模型赋予每个细胞的“注意力权重”(类似于模型对不同细胞特征的关注程度),研究人员发现了一些有趣的规律。如图6A所示,不同药物处理导致的典型细胞形态在UMAP图上呈现出独特的分布模式。进一步分析高注意力细胞(即模型认为最具代表性的细胞)的特征(图6B)揭示了不同药物的独特“形态签名”:
nocodazole处理的细胞呈现明显的圆形特征,球形度达到0.71(远高于平均值0.63);
blebbistatin和H1152处理的细胞则表现出扁平(扩展度分别为0.93和0.91)和不规则(偏心率分别为0.97和0.94)的特征。
研究团队也探讨了高注意力细胞与信号活性的关系。如图6D-G所示,研究人员使用ERK-KTR报告基因来测量MEK活性:当ERK活跃时,荧光信号在细胞质中增强;当ERK被抑制时,荧光信号在细胞核中增强。ERK比率(细胞核与核周区域的ERK强度比)越高表示ERK活性越低。研究发现,binimetinib分类器的注意力得分与ERK比率呈现显著相关,这一相关性明显高于其他药物和对照组(0.14和0.24)。这表明仅基于3D细胞和细胞核形状的模型能够学习到MEK抑制和活性的特征形态。
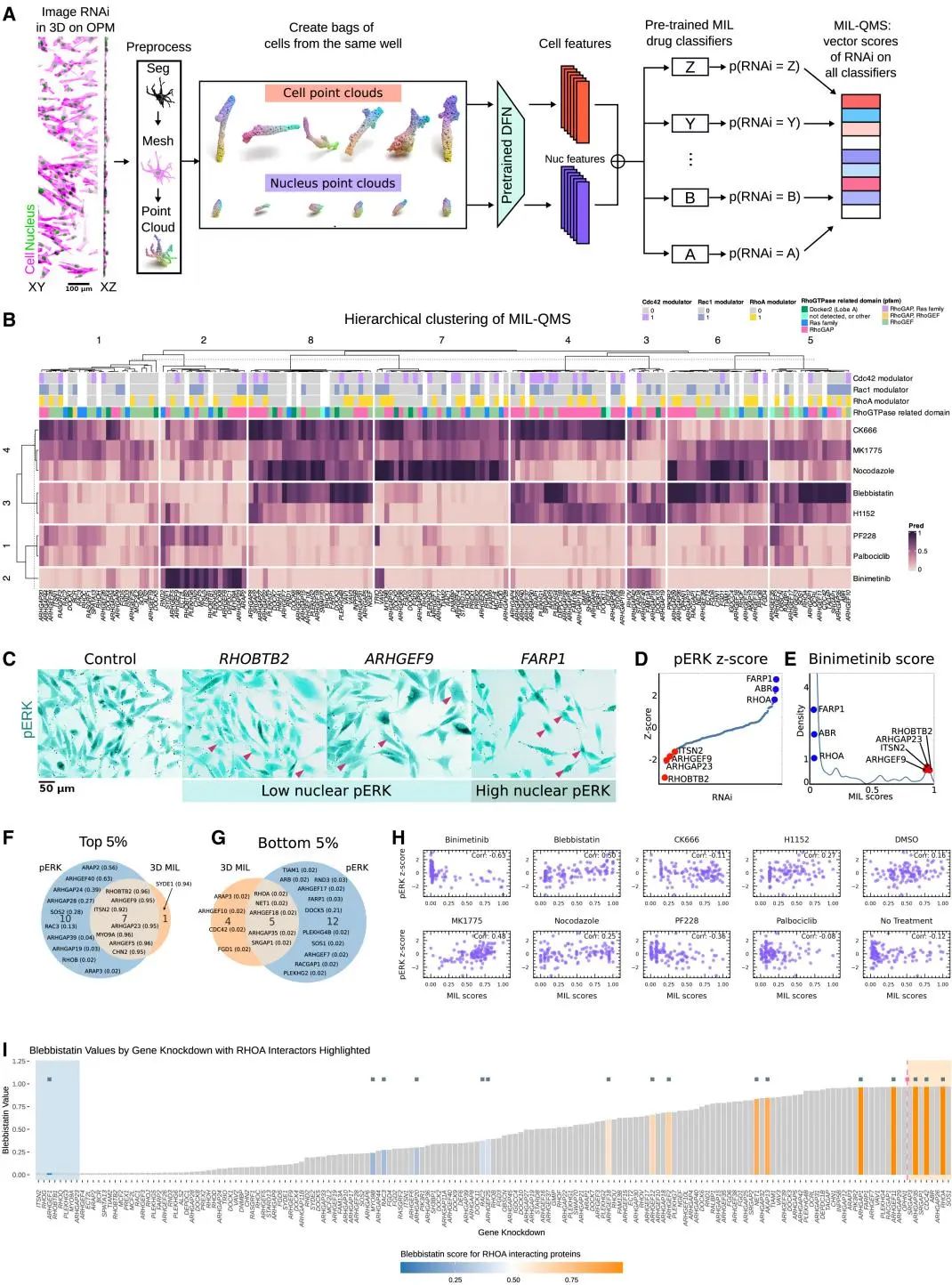
随后,研究团队使用MorphoMIL分析了167个基因(包括RhoGEFs、RhoGAPs和Rho家族GTPases)敲低后的细胞形态变化。如图7所示,他们为每个基因构建了8维特征向量(MIL-QMS),并将其分为8个簇(图7B)。研究发现MorphoMIL的预测分数与激酶活性相关:binimetinib分类器的得分与pERK水平呈显著相关。此外,通过分析RHOA-ROCK-NM2通路中的基因,研究团队发现3D细胞形态可以预测蛋白质之间的物理相互作用。
结论
本文介绍并验证了两种新颖的流程:DFN(特征提取)和MorphoMIL(包含分类功能)。这些流程旨在利用3D细胞形态来确定潜在的细胞状态,模拟受治疗干扰的细胞群体的异质性,并提高可解释性。这项工作强调了结合GDL(图深度学习)和MIL(多实例学习)进行3D形态分析的潜力,以推动高通量药物发现工作,并为探索基于形状的细胞状态预测开辟了新途径。作者的所有算法都打包为开源Python包,以便更广泛的研究社区使用。
作者已经证明,先进的3D成像技术结合深度学习和多实例学习的模型是一种强大的方法,可以建立对遗传或化学扰动效应的机制性洞察。作者的方法快速、成本效益高且可扩展,使其成为同时查询多个信号通路活动的高效工具。因此,这种方法在小分子药物、生物制剂以及小干扰RNA(siRNA)/CRISPR文库的大规模筛选中具有相当大的潜力。此外,3D形状分析的应用在临床样本的诊断评估中显示出巨大的前景。
编译|于洲
审稿|王梓旭
参考资料
De Vries M, Dent L G, Curry N, et al. Geometric deep learning and multiple-instance learning for 3D cell-shape profiling[J]. Cell Systems, 2025, 16(3).
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢