DRUGAI
本文介绍一篇由浙江大学侯廷军教授、谢昌谕教授及碳硅智慧近期在化学领域国际知名期刊Chemical Science上发表题为《TRAP: A Contrastive Learning-Enhanced Framework for Robust TCR-pMHC Binding Prediction with Improved Generalizability》的研究论文,第一作者为浙江大学博士研究生戈婧萱。

T细胞受体(TCR)与肽-MHC I(pMHC)复合物的特异性结合在触发针对潜在健康威胁的适应性免疫应答中至关重要。精准预测TCR与pMHC之间的结合对于加速免疫治疗领域的研究、开发新型免疫疗法具有重要意义。然而,现有用于TCR-pMHC结合预测的模型在面对未见过的抗原表位时,往往预测性能欠佳,这极大地制约了这些模型在实际临床应用中的广泛推广与适用性。本文作者开发了名为TRAP的预测模型,利用对比学习来提高模型的性能,并通过映射结构和序列特征以提升模型在处理不可见表位时的预测能力。此外,TRAP能够精准识别相似抗原表位间TCR交叉反应性预测中的潜在混淆因素,有效解决了传统模型在这一复杂场景下的性能瓶颈。这种高度稳健的性能使TRAP模型成为现实世界中大规模TCR-pMHC结合预测任务的理想工具。为验证其实际效果,研究团队开展了一项具体的案例研究。结果表明,TRAP能够精准识别出与参考实验数据具有相当结合自由能的TCR分子(即“命中TCR”),凸显了TRAP的实际应用潜力,更为基于TCR的个性化免疫治疗开发提供了强有力的技术支撑。
TRAP模型架构总览
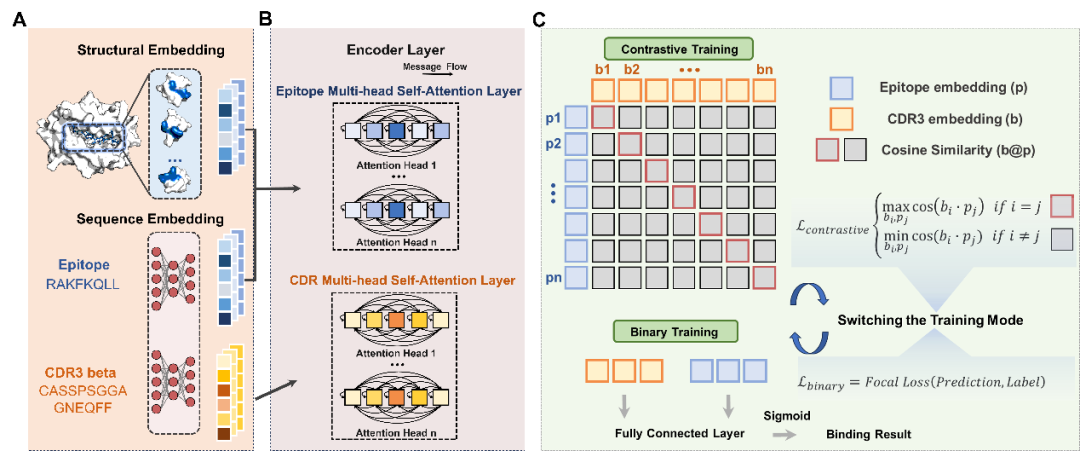
图1. TRAP架构总览
TCR和pMHC的识别涉及跨多个位点的复杂残基间相互作用。除依赖序列信息外,TRAP还整合了反映各种MHC表位构象的关键结构信息。具体而言,作者聚焦于以每个表位残基为中心、半径为特定截止距离的局部结构邻域,最大限度地减少了来自整个 MHC 结构的外部噪声。随后,CDR3β和表位的嵌入被传递到编码器进行特征编码。
TRAP 的另一个创新之处在于其运用了对比学习机制。以往的研究通常通过错配阳性对来创建阴性样本以训练模型,但这种策略完全忽略了交叉反应性,而交叉反应性在免疫系统中普遍存在,尤其在数据集中TCR或表位存在高度相似性时更为显著。为了解决这个问题,TRAP 在阳性样本中运用了对比学习,最大限度地利用了阳性数据。TRAP 将阳性结合对分离为CDR3β和表位,然后使用编码器生成的表征计算每个CDR3β和表位之间的余弦相似度构建相似度矩阵,旨在最大化阳性样本对的余弦相似度将CDR3β的表征与表位对齐。然后,TRAP切换到二分类训练,添加通过统一频率的负样本策略生成的负结合对。该策略根据pMHC的频率错配结合对以生成负样本。在每个训练周期中,TRAP会在这两种训练模式之间交替。
TRAP具有高度准确且可推广的预测能力
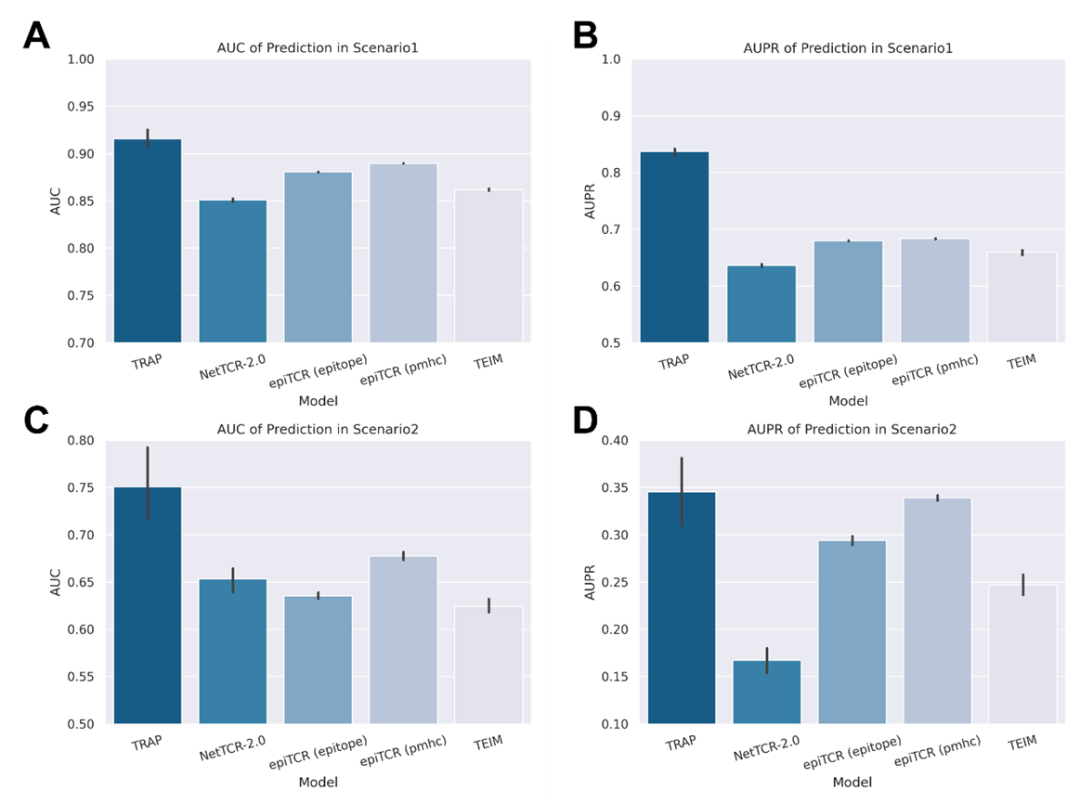
图2. 不同模型的分类任务指标的结果
为系统验证TRAP模型在真实免疫学研究场景中的实用效能,作者设置了两种模拟应用场景:在场景1中,将阳性样本和阴性样本以9:1的比例随机分成训练集和测试集,模拟预测数据库中存在的TCR-pMHC相互作用的常见情况;在场景2中,训练集包含数据集中记录了5个以上阳性结合CDR3β的表位对,测试集包含训练集中剩余的表位对,这些表位尚未出现在训练集中,这意味着没有表位同时出现在训练集和测试集中。
在场景1中,TRAP的AUC达到0.92,AUPR达到0.84,比排名第二的模型(包含pMHC信息的epiTCR)的AUPR高出22.4%。在场景2中,对于未知表位对的预测,TRAP展现出良好的泛化能力,并且在AUC上分别比包含pMHC和表位信息的epiTCR高出10.8%和18.1%。
结构和序列联合知情决策:对交叉反应有更好的预测能力
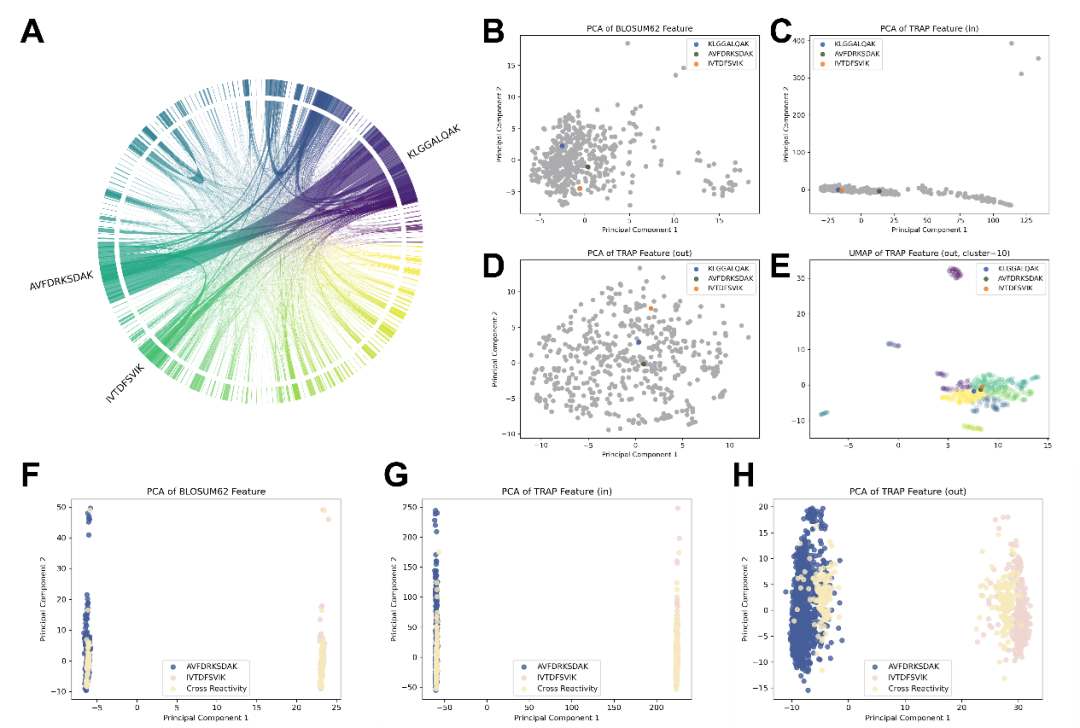
图3. (A) 展示了交叉反应性表位对的数量(高亮排名前三的表位)。 (B-D) 不同表位特征的降维表示。(E) 对表位的TRAP输出特征进行聚类和降维。(F-H) 表位-CDR3β对特征的降维表示。
TCR与抗原表位的相互作用机制与小分子药物-靶点结合存在本质相似性——TCR并非严格遵循"一对一"识别模式,而是能够与多个结构相似或功能相关的表位发生特异性结合,这一现象被称为TCR交叉反应性。在基于TCR的肿瘤免疫治疗、自身免疫病干预等临床场景中,交叉反应性可能引发严重安全隐患:当TCR与脱靶表位结合时,可能触发非预期的免疫激活,导致自身免疫毒性或疗效衰减。值得关注的是,公开免疫数据库中已积累大量TCR交叉反应实例,其复杂程度远超传统药物研发中的脱靶效应评估。本研究创新性选取三种交叉反应频率最高的表位作为代表性样本,系统验证了TRAP模型在解析交叉反应网络中的核心优势。作者通过t-SNE算法对不同的特征表示(BLOSUM62、TRAP的输入和输出特征)进行降维,比较了这三种表位在所有表位空间中的分布。实验结果显示,在使用BLOSUM62矩阵和TRAP输入特征时,表位似乎聚集在一起,难以区分;当使用TRAP模型输入特征时,不同类型的表位显示分散分布,从而降低了区分它们的难度。此外,具有交叉反应性的三种表位仍然相对接近,这表明TRAP确实有效地捕捉了三种表位之间的相似性。此外,虽然某些表位在序列水平上可能难以区分,但TRAP提取的特征可以很好地区分它们。
作者接下来分析了与HLA-A*11:01 AVFDRKSDAK和HLA-A*11:01 IVTDFSVIK相关的结合对,因为它们在表位特征空间中距离较近。作者发现,在由BLOSUM62和TRAP输入组成的特征空间中,结合对呈现出难以区分的聚类方式。在TRAP输出的特征空间中,结合对分散且易于区分,根据表位形成两个不同的聚类,而具有交叉反应性的结合对远离聚类中心,倾向于另一个聚类。这凸显了TRAP清晰区分结合对和捕捉对水平交叉反应本质的能力。
有效的负样本采样策略以规避学习捷径
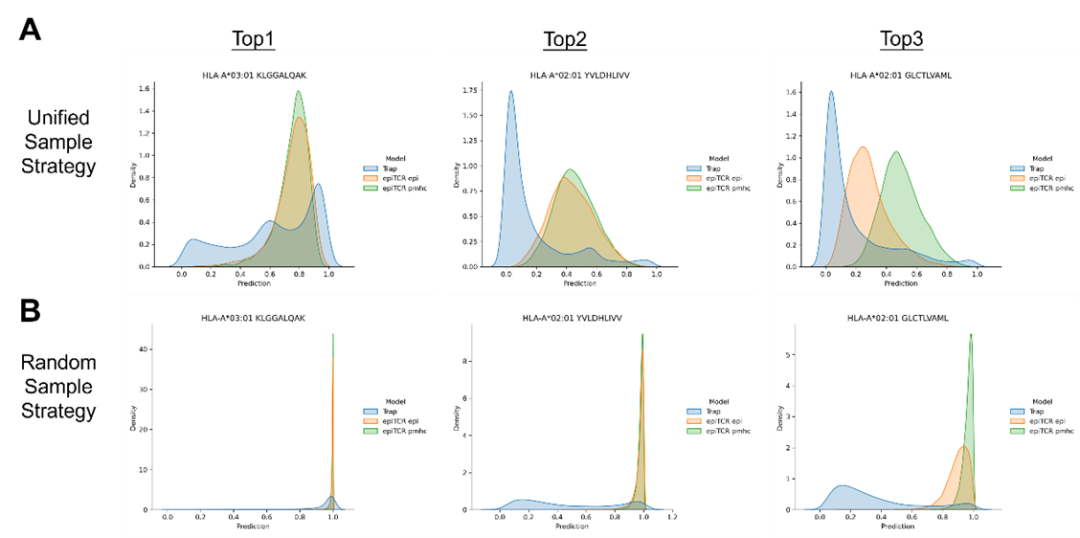
图4. 来自真实阴性数据集的表位-CDR3β对的预测结果的分布。
TCR-pMHC结合数据呈现明显的长尾分布特征,为每个表位固定配置等量阴性CDR3β样本,对于阳性CDR3β频率较高的表位,阳性CDR3β的数量将明显超过阴性,反之亦然。这种失衡数据分布将诱导模型学习到数据偏差而非真实生物学规律,具体表现为模型可能简单地将高频表位预测为阳性,而无法准确识别低频表位的特异性结合模式。为了避免这种不平衡,作者参考了Jiang等人提出的统一负样本策略,确保每个表位都具有相似的阳性与阴性TCR比例。
作者建立了一个真阴性数据集,其中包含来自健康供体TCR库的CDR3β,并假设这些CDR3β缺乏与任何表位的结合能力。作者选择了结合对中出现次数最多的三个表位,并将它们与真阴性数据集中的CDR3β配对。理想情况下,所有这些配对都应该被预测为阴性结合,并根据获得的假阳性结果来衡量成功率。为此,作者使用了TRAP和epiTCR来测试真阴性数据集。如图4A和B所示,使用随机采样数据训练的模型的假阳性率显著高于使用统一采样训练的模型,这揭示了该采样策略在解决TCR-pMHC结合问题方面的优势。此外,TRAP对所有三种pMHC阴性样本的预测精度均优于epiTCR,进一步验证了TRAP的卓越性能。
利用TRAP的筛选案例研究
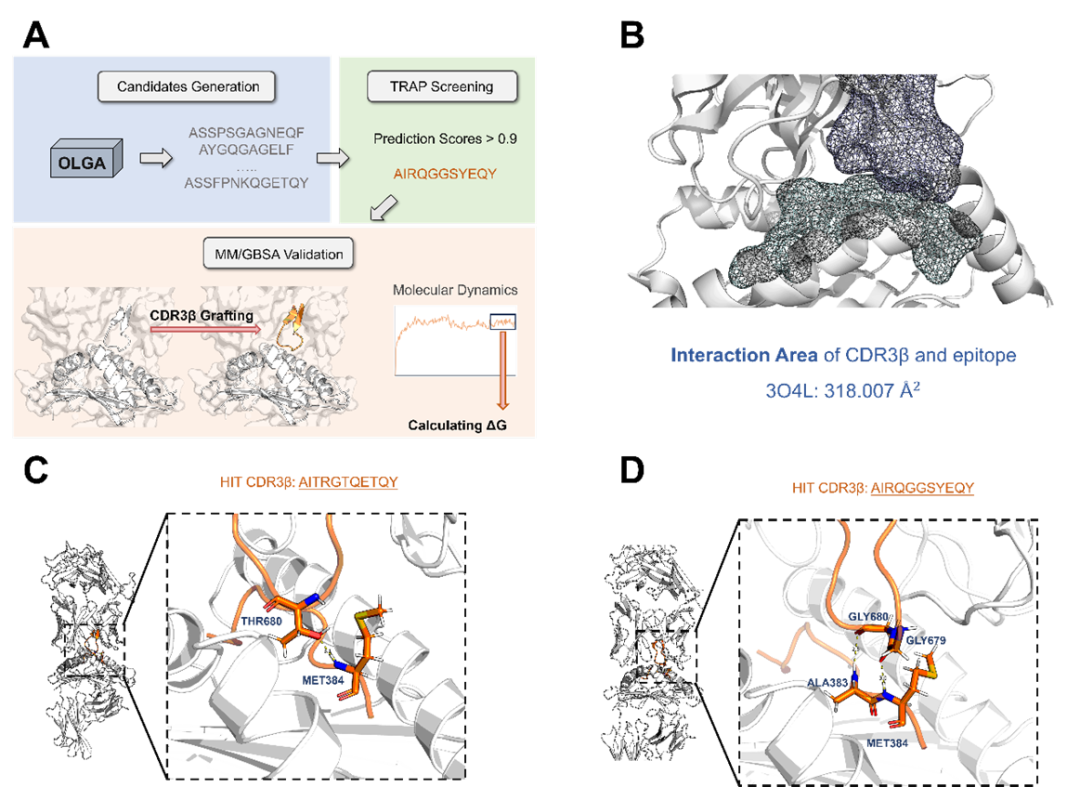
图5. 利用TRAP筛选案例研究的工作流程
作者将TRAP应用于现实世界的生物实验场景,设计了一个用于筛选案例研究的工作流程。使用OLGA随机生成CDR3β序列,并通过从TRAP 训练数据集中剔除重复序列来确保其唯一性。当一个序列的预测得分超过设定阈值时,即被指定为候选CDR3β。通过利用分子动力学模拟以及MM/GBSA自由能预测方法,作者验证了该工作流程用于计算机模拟筛选的实用性和巨大的应用潜力。
总结展望
TRAP 采用对比学习技术实现TCR与表位表征空间的对齐,并通过结构特征增强模型对复杂变异的辨识能力,使其能够捕捉未知表位场景下的关键差异,从而取得预测精度的突破。实验验证表明,基于健康个体TCR数据集的测试中,优化的负采样策略有效抑制了模型分数虚高现象。值得注意的是,TRAP 不仅能够精准区分不同表位,还可同时捕获TCR的交叉反应性与结合特异性。案例研究进一步证实,TRAP可有效筛选出与已知晶体结构具有相似结合模式的TCR靶标,凸显了其在TCR相关治疗开发中的潜力。
参考资料
J. Ge, J. Wang, Q. Ye, L. Pan, Y. Kang, C. Shen, Y. Deng, C. Hsieh and T. Hou, Chem. Sci., 2025, DOI: 10.1039/D4SC08141B.
https://doi.org/10.1039/D4SC08141B
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢