DRUGAI
尽管蛋白–蛋白相互作用图谱已取得显著进展,其在不同组织中的特异性仍研究不足。在本研究中,研究人员基于蛋白质共丰度可预测功能关联的前提,整合并分析了来自11个人体组织的7,811个蛋白质组学样本的蛋白丰度数据,构建了一个组织特异性的蛋白相互作用图谱。研究人员发现,该方法不仅能复现已知的蛋白复合物,还能揭示细胞的更大结构组织。稳定蛋白复合物的相互作用在各组织中高度保守,而细胞类型特异性的结构(如突触成分)则是组织间差异的重要驱动因素。
研究结果显示,超过25%的蛋白关联具有组织特异性,其中不到7%可归因于基因表达差异。针对脑组织,研究人员通过突触体的共分级实验、脑源拉下数据的人工整理以及AlphaFold2结构建模,对蛋白相互作用进行了验证。进一步地,研究人员还构建了与精神分裂症相关基因的脑组织相互作用网络,表明该方法可用于在与脑疾病相关的基因座中功能优先筛选候选致病基因。

蛋白–蛋白相互作用决定了细胞的物理结构和功能,其异常可能导致疾病。尽管近年来质谱技术和高通量筛选方法的进步推动了蛋白互作的研究,现有数据库多为不具上下文的全局互作信息,而实际互作在不同组织或细胞状态中具有高度特异性,且不到一半的蛋白可在所有组织中检测到。
为理解细胞类型特异功能、识别药物靶点以及构建系统级细胞模型,明确蛋白互作的组织特异性至关重要。以往研究常借助基因表达数据推测组织特异性互作,但mRNA共表达对预测蛋白互作的准确性有限。相比之下,蛋白共丰度(coabundance)被证明更能准确预测蛋白关联,因为复合物亚基往往协同表达并以特定化学计量组装,未配对亚基则倾向降解。
在本研究中,研究人员基于7,811个人体样本的蛋白质共丰度数据,构建了涵盖11种组织、约1.16亿对蛋白的组织特异性蛋白关联图谱。该图谱不仅复现了已知复合物,还揭示了功能模块间的关系,并为疾病相关基因提供了上下文关联线索。研究人员以脑组织为例,结合多种互补数据验证该图谱在组织特异性致病基因和互作优先排序中的应用价值。
研究结果
基于蛋白共丰度的全基因组蛋白关联评分
研究人员首先收集了癌症队列的蛋白质丰度数据,汇总了来自14种人体组织的50项蛋白质组学研究,包含5,726个肿瘤样本和2,085个邻近健康组织样本,并配套收集了其中2,930个肿瘤样本和722个健康样本的mRNA表达数据。利用蛋白复合物成员在转录和翻译后水平的共调控特性,研究人员计算了基于丰度数据的蛋白–蛋白关联概率。具体而言,先对数据进行对数转换和中值归一化,再在每项研究中对至少在30个样本中被共同检测的蛋白对计算皮尔逊相关系数。随后,以已知蛋白复合物亚基为正例(来自CORUM数据库),利用逻辑回归模型将相关性估计转换为蛋白关联概率。
通过与蛋白共分级和mRNA共表达等方法对比,研究人员发现蛋白共丰度在复现已知蛋白复合物上表现最佳(AUC=0.80),优于蛋白共分级(AUC=0.69)和mRNA共表达(AUC=0.70)。将mRNA与蛋白丰度联合使用并未进一步提升识别能力,且剔除基因表达后所得共丰度估计仍表现良好,提示预测能力主要源于翻译后调控而非基因表达。因此,研究人员最终仅使用蛋白共丰度来计算蛋白关联概率。
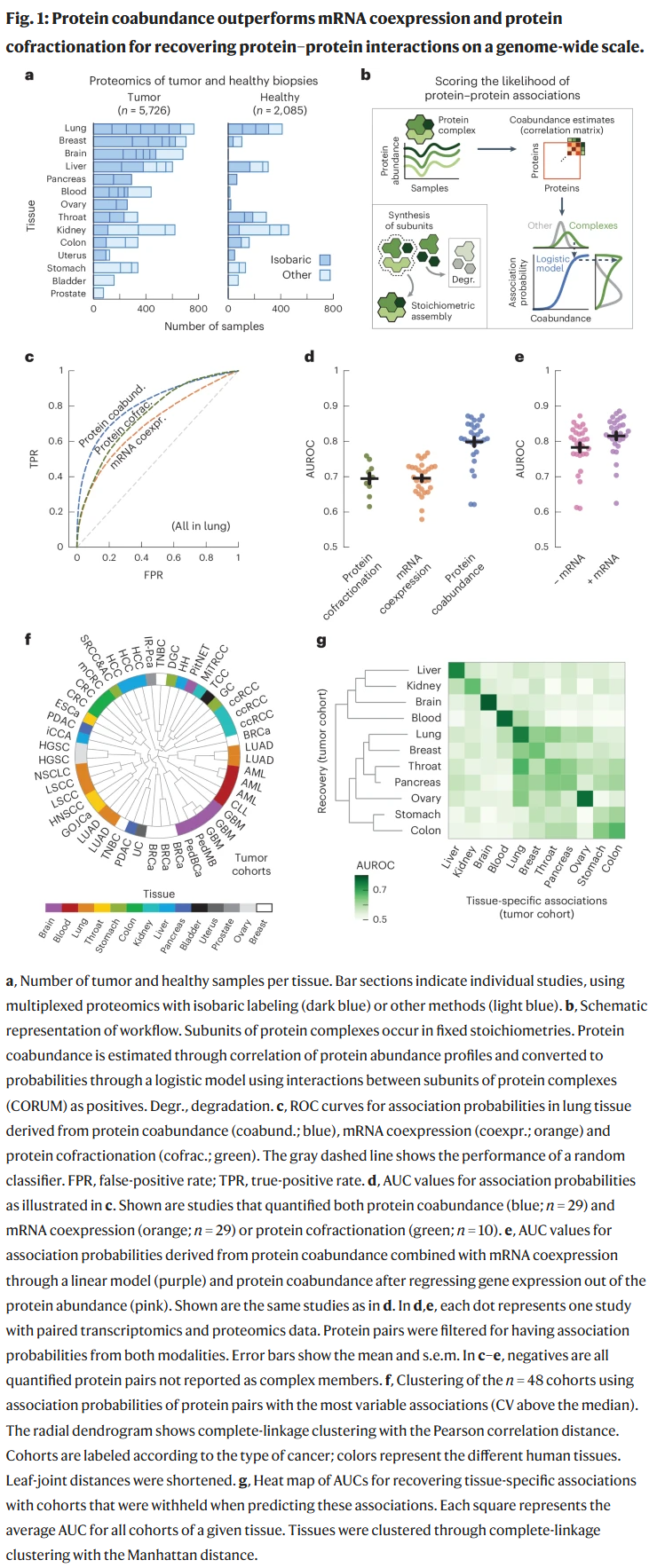
构建组织特异性蛋白关联图谱
研究人员进一步验证了同一组织的不同研究是否能反映一致的组织特征。通过聚类分析,发现相同组织的样本之间具有较高一致性。定义“组织特异性”的蛋白关联为:在某一组织中关联评分高于95百分位且在其他组织中低于0.5。使用“留一法”验证后发现,这些组织特异性关联主要由相应组织的样本所复现,说明组织来源是蛋白关联差异的关键驱动因素。
聚合相同组织的研究数据后,研究人员为11个人体组织生成了统一的蛋白关联评分。在已知蛋白互作的复现能力上,组织层级的评分优于任何单个队列,且肿瘤来源数据的表现优于健康组织。这些评分反映了肿瘤的遗传异质性及样本之间的变异。研究人员还利用健康组织得出的特异性关联,用肿瘤样本数据进行验证,结果显示一致性良好,进一步验证了该图谱的可靠性和代表性。
最终,研究人员构建了一个涵盖116,000,000对蛋白的组织特异性关联图谱,平均每个组织包含约5,600万对蛋白评分,其中约1,000万对具有较高关联性(评分>0.5),约49万对属于“高可信关联”(评分>0.8)。
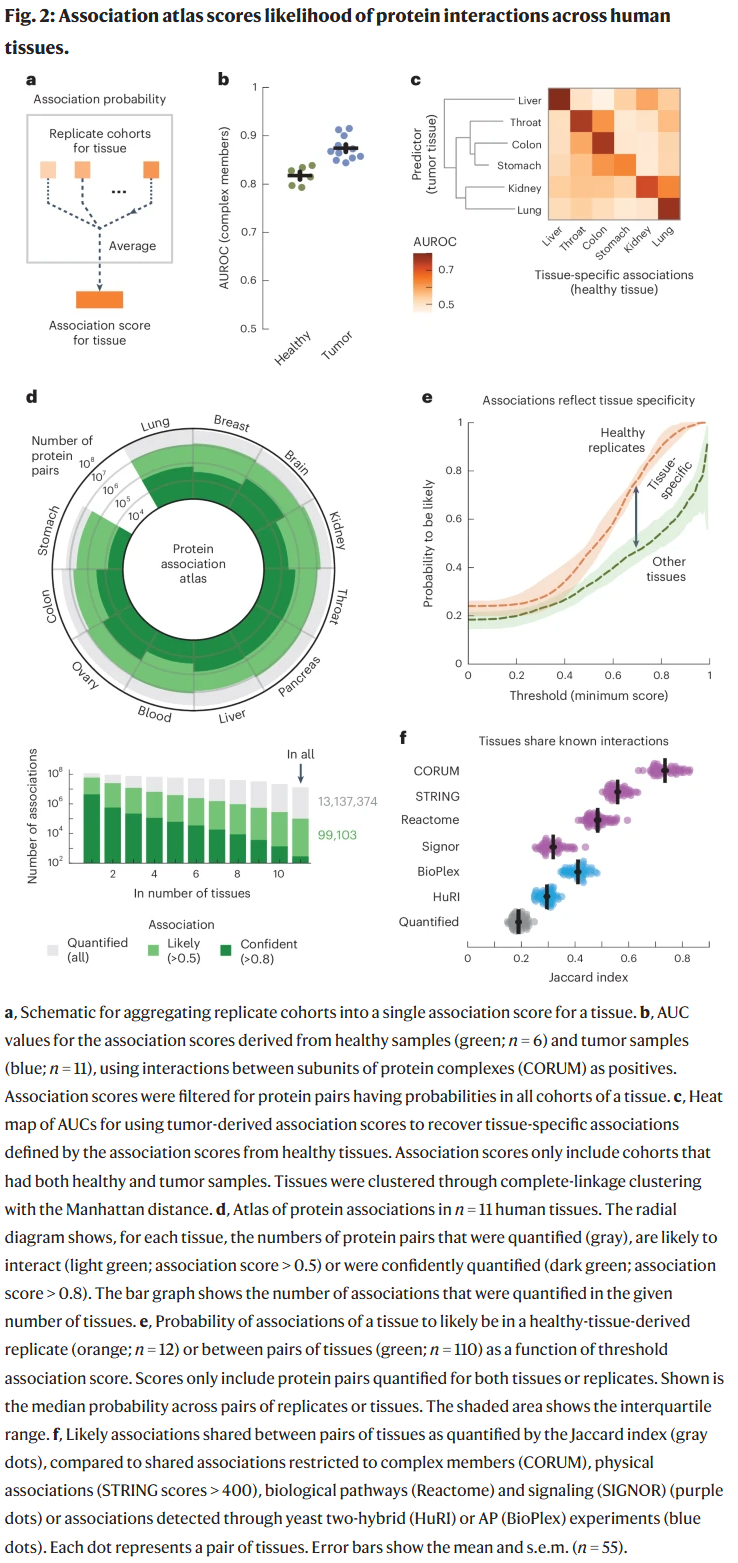
组织间差异并非由基因表达驱动
尽管蛋白互作的前提是基因在组织中被表达,但仅约7%的组织间蛋白关联差异可归因于基因表达差异。因此,蛋白共丰度反映的是翻译后层级的调控,进一步强调其独立于转录水平的预测价值。
各组织特异性关联比例
在健康组织与肿瘤组织配对比较中,46.3%的“可能关联”以及90.2%的“高可信关联”可被对方组织所复现。而在不同组织间,这些比例分别下降至32.9%和54.6%。研究人员据此估计,约有25.8%的蛋白关联具有组织特异性。
各组织复现其特异性细胞组分
通过比较组织间的Jaccard相似系数发现,已知蛋白复合物等稳定互作在多个组织间保持一致,而与信号转导或时空调控相关的动态互作则更多体现组织差异。此外,特定组织(如大脑、喉部、肺、肝)中的细胞结构成分(如突触、纤毛、过氧化物酶体)表现出显著的组织特异性,说明这些结构是组织差异的重要来源。
图谱揭示细胞类型特异性互作
以神经特异的AP2适配蛋白复合物为例,研究人员发现其在大脑中的关联更强,尤其是与突触相关蛋白的互作,而非突触蛋白则在大脑中互作较弱。研究人员还识别出如血红蛋白与贫血、乳糜微粒与克罗恩病、纤维蛋白原与肝病等组织特异的疾病相关互作。
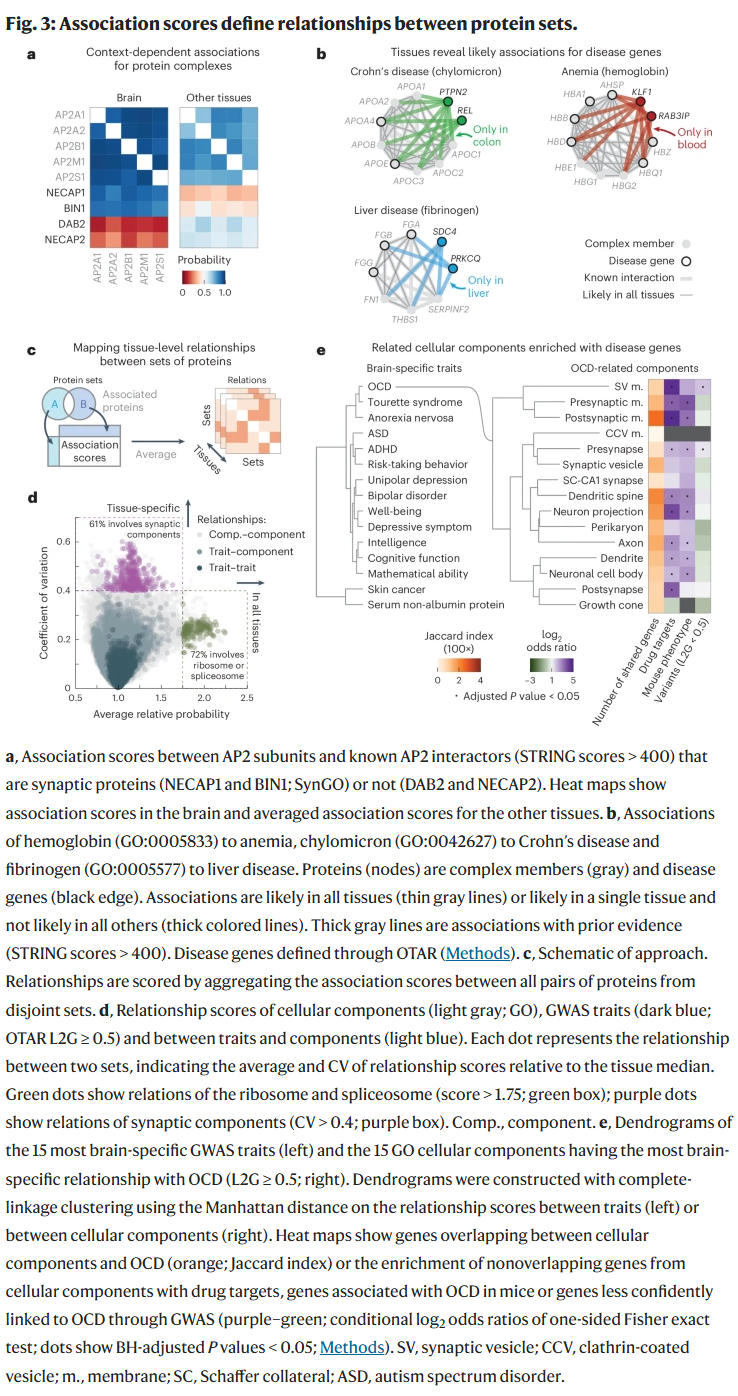
组织特异的表型与细胞结构关联
进一步,研究人员通过GO注释与OTAR数据库中GWAS基因,系统评估了表型与细胞结构之间的组织特异性关系。例如,在大脑中与强迫症(OCD)相关的15个细胞成分几乎均为神经元特异,且在剔除GWAS已关联基因后,这些成分仍富集药物靶点、小鼠模型致病基因或低置信GWAS基因,说明该方法可用于识别与疾病相关的潜在功能模块。
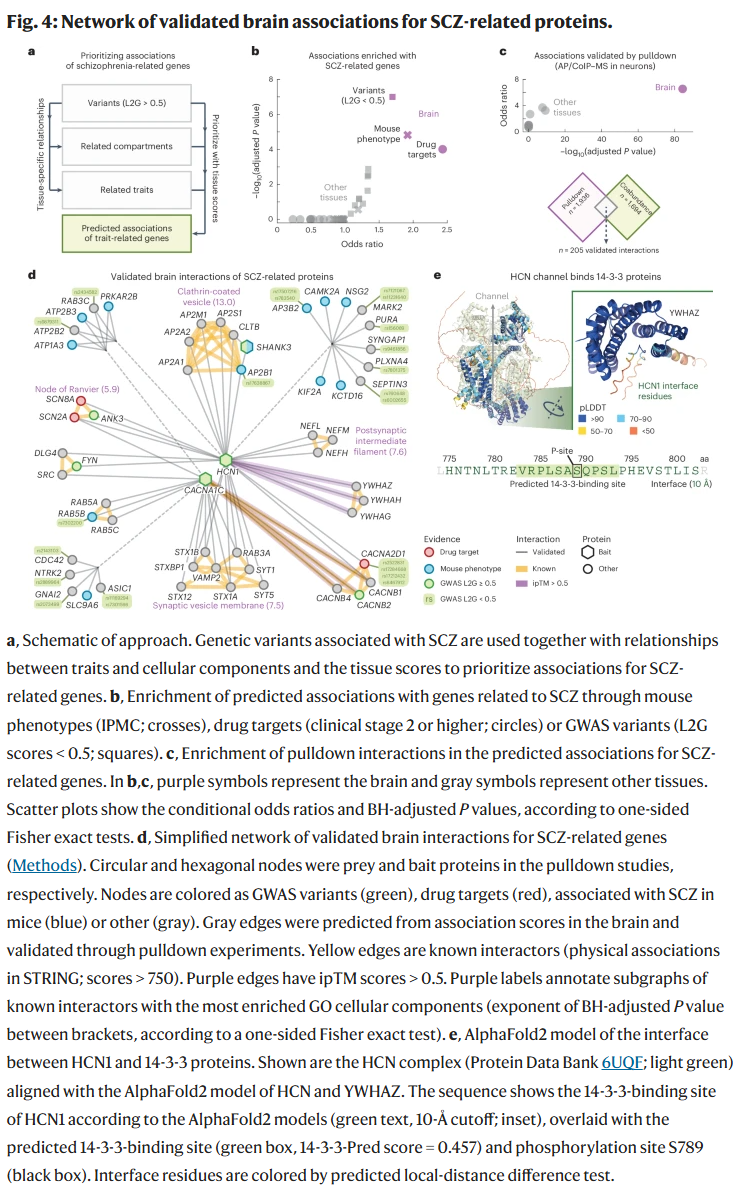
验证精神分裂症相关基因的脑组织互作
研究人员构建了一个脑组织中与精神分裂症(SCZ)相关基因的互作网络。将GWAS关联的369个SCZ起始基因与其组织特异性关联基因筛选组合,识别出高置信互作对。进一步使用脑组织中实验验证的拉下质谱互作数据对预测结果进行验证,显示SCZ相关互作在脑组织中显著富集。
研究人员从中筛选出205个已验证的脑组织SCZ互作对,并利用AlphaFold2预测其结合结构。其中三个14-3-3蛋白与HCN1之间的互作被高置信预测,且其结合界面位于HCN1的C末端无序区,中心位点S789已被实验证实具有磷酸化依赖性结合能力。
此外,该网络中包含15个GWAS位点下的弱置信SCZ候选基因,其与SCZ核心基因的功能互作表明其可能为更可能的致病候选者。
基于共分级的突触特异性互作组
作为蛋白关联图谱的最终应用,研究人员聚焦于突触的互作网络。为此,研究人员从大鼠脑组织中提取并纯化了突触体(synaptosome),作为验证脑组织蛋白互作的另一种手段。利用尺寸排阻色谱(SEC)将突触体分为75个组分,随后通过液相色谱-质谱联用(LC-MS/MS)进行检测,共鉴定出3,409种蛋白,包括CCT复合物等已知复合物,其组分在不同组分中表现出高度相关性(平均相关系数为0.96)。
研究人员对突触体的分级谱图进行了预处理,并计算了4,276,350对蛋白在组分中的共丰度值,以评估它们的共分级程度。为提高互作评分的置信度,研究人员整合了大鼠突触体、体内小鼠脑组织及人类胶质母细胞亚细胞分级的共分级数据,基于所有三套数据中均被检测的直系同源蛋白构建了逻辑回归模型(以CORUM数据库中的复合物为正例)来估算互作概率。
最终构建的突触互作组包含1,619种蛋白之间的1,309,771对互作概率,优于任何单独数据的预测能力。该互作组中,24%的蛋白为SynGO数据库标注的突触蛋白,49%在小鼠脑中已被报道为突触富集,56%曾在小鼠突触体的交联-MS中被鉴定。所有已知交联互作在本研究中均被检测,其关联评分显著高于其他蛋白对(0.59 vs 0.37)。此外,突触蛋白之间的互作显著强于非突触蛋白之间的互作,尤其在脑组织中更为明显。
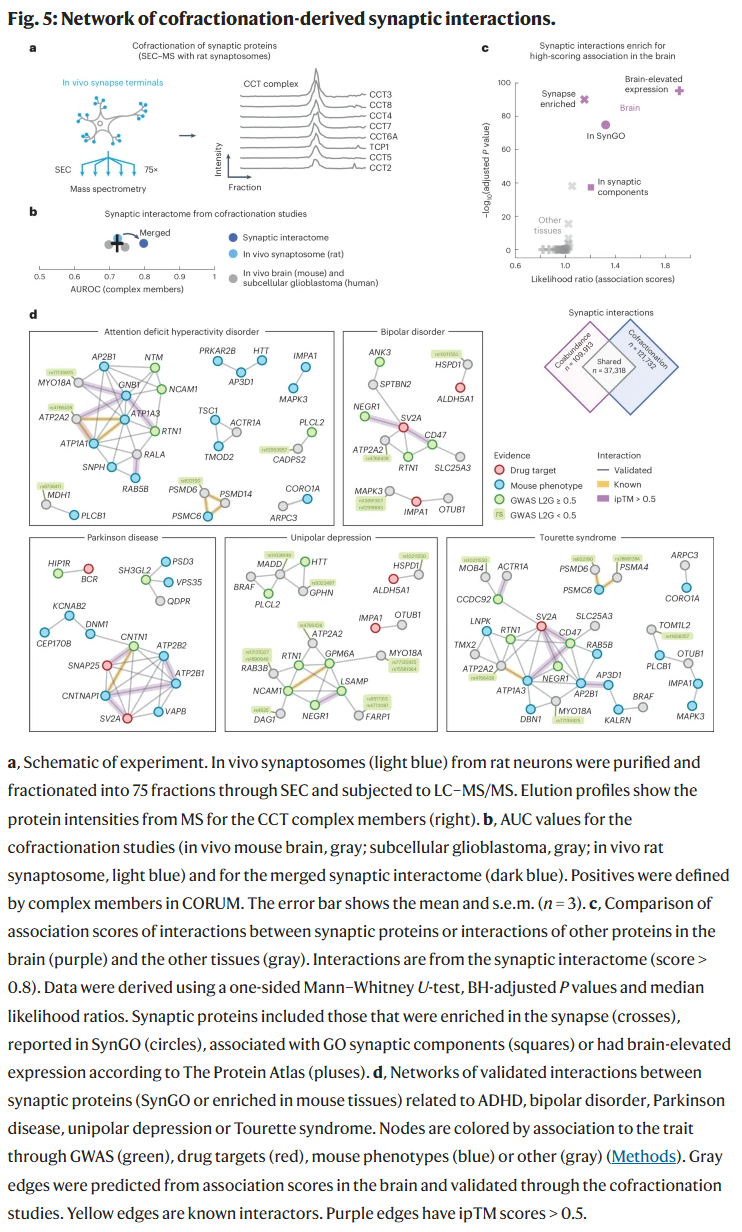
验证脑疾病相关基因的突触互作
突触富集蛋白之间的互作在脑组织中更可能发生。基于此,研究人员构建了一个仅包含突触蛋白的互作网络,这些蛋白在SynGO数据库中被定义或在小鼠脑中被认为突触富集,同时在大脑中共丰度高、在突触体中共分级概率高。最终得到的网络包含37,318个验证过的蛋白互作,且这些互作主要为脑组织特异性,仅20%在大多数其他组织中被认为是可能互作,仅有极少数被现有蛋白数据库(STRING、HuMAP、IntAct、BioPlex)收录。
研究人员进一步筛选与脑疾病相关的突触蛋白互作。这些疾病来源于GWAS研究,相关基因在小鼠表型库(IMPC)或药物靶点数据库(ChEMBL)中有支持,且其关联在脑组织中显著高于其他组织。最终筛选出727个高置信突触互作,这些互作在突触体和脑组织中的关联评分分别为0.7和0.81。
研究人员使用AlphaFold2对这727个互作对进行结构预测,发现其预测得分(pDockQ)显著高于CORUM或HuMAP中的已知互作;其中105对具有中等置信度(ipTM > 0.5),部分互作在小鼠突触体的交联-MS中有实验支持。
优先筛选脑疾病的突触致病基因
在这些互作网络中,研究人员发现部分基因虽然在遗传位点上的证据较弱,但由于其与已知疾病基因存在验证过的突触互作,可能是更有可能的致病基因。例如:
ADHD:MDH1、CADPS2、PIK3C3
精神分裂症(SCZ):TOM1L2、AP2B1、PSD3、MYO18A、ATP2B2、TMX2
阿尔茨海默病:CLPTM1、MADD
孤独症:ATP2B2、ATP2A2
单相抑郁症:MADD
双相障碍:ATP2A2
其中大多数基因在脑组织中的表达位居前列,支持其致病潜力。
此外,研究人员还发现某些突触互作对虽非L2G评分最高基因,但具有其他支持证据。例如,PAFAH1B1 与 RHOA 的互作在突触互作图谱中被确认,两者皆与抑郁症相关。虽然PAFAH1B1在其位点上的L2G评分不最高,但其在大脑中的表达更强、更特异,且编码突触蛋白,支持其作为潜在致病基因。
这一案例表明,结合亚细胞结构与组织特异性研究,可构建更具生理意义的蛋白互作网络,并助力组织相关疾病基因的筛选。
讨论
尽管已有大量蛋白互作图谱,但组织间的差异性仍未被全面理解。蛋白共丰度相比mRNA共表达在预测互作方面更为准确。本研究进一步证实其优于传统共分级分析结果。值得注意的是,即使去除由mRNA表达变化导致的丰度差异,也不会显著影响预测性能,说明蛋白互作更多受翻译后调控机制控制,如过量亚基的降解等。
研究人员的分析结果具有高度一致性:相同组织的不同队列具有类似的蛋白关联模式,肿瘤组织与健康组织的评分可互相验证,关键复合物在所有组织中均被高质量预测。约46%的可能关联与90%的高置信关联可在健康与肿瘤组织中重现,为组织特异性生物学研究提供可靠假设。
研究人员估算约25%的蛋白关联具有组织特异性,而其中因基因表达差异导致的比例仅占7%。细胞类型特异的结构(如突触)是造成组织间蛋白关联差异的重要因素,翻译后修饰也可能是潜在机制之一。
最后,该图谱也支持在组织水平上探索性状与细胞结构之间的关系,特别是在脑组织和突触功能方面的应用表现突出。研究人员展示了多个脑相关疾病中与已知致病基因或药物靶点富集的验证互作,并提出了一种基因优选策略,有助于候选药物靶点的识别。组织特异性的网络与AlphaFold2模型的结合为理解疾病机制和靶点筛选提供了整合性工具,且具备提高靶向治疗安全性的潜力。
整理 | WJM
参考资料
Laman Trip, D.S., van Oostrum, M., Memon, D. et al. A tissue-specific atlas of protein–protein associations enables prioritization of candidate disease genes. Nat Biotechnol (2025).
https://doi.org/10.1038/s41587-025-02659-z
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢