DRUGAI
RNA是一种高度多功能的分子,已被研究人员设计用于治疗、诊断以及体内信息处理系统等多种应用。然而,RNA序列、结构与功能之间复杂的关系,往往需要对大量候选序列进行广泛的实验筛选。为此,研究人员提出了一种通用且高效的神经网络架构——SANDSTORM(Sequence AND Structure-informed RNA Modeling),该模型利用RNA分子的序列与结构信息,在多种场景下实现功能预测。研究人员还将该预测模型与生成对抗式RNA设计网络(GARDN)相结合,使其能够以目标实验属性为条件,生成多样化的功能性RNA分子。该方法不仅能够设计出在功能上优于训练集中实例或传统热力学算法返回结果的新型序列候选,还可在仅使用384个示例序列的情况下部署。SANDSTORM与GARDN因此成为开发功能增强型RNA分子的强大预测与生成工具。

RNA具备同时编码遗传信息和执行多种功能的能力,使其成为一个极具潜力的分子平台,广泛应用于治疗、诊断以及合成生物系统的开发。由于RNA仅由四种标准核苷酸组成,并遵循简单的Watson-Crick碱基配对规则,相较于蛋白质系统,其分子的设计问题在计算上更易处理。因此,研究人员已开展大量工作,旨在开发稳健的工具用于RNA二级结构的预测以及基于结构的逆向设计。然而,目前尚未建立一套同样通用的计算工具来工程化RNA功能。随着RNA系统的日益复杂和广泛应用,热力学属性的确定性与实际功能之间的偏差也在不断扩大,常常需要对大量RNA序列进行实验筛选。因此,研究人员迫切需要开发数据驱动的方法,以揭示RNA序列与功能之间的关系,从而加速RNA装置的设计并提升其在生物医学应用中的性能。
为支持基于数据的方法进行RNA工程设计,研究人员在多种背景下构建了大规模并行报告系统,从而产生了多个将功能性RNA与其实验性能相对应的数据集。例如,Toehold开关、5′非翻译区(UTR)、CRISPR引导RNA以及核糖体结合位点(RBS)等RNA序列,均已通过高通量筛选进行了功能表征。在这些研究中,研究人员通常使用one-hot编码的方式表示RNA序列,并据此构建预测模型。尽管这些模型的任务都是预测RNA功能,但其架构与性能却存在较大差异,导致目前的预测方法往往针对特定功能进行了大量微调,难以推广至RNA的多种应用场景。正如热力学算法能够广泛应用于不同类别的RNA序列-结构关系预测,研究人员希望开发出同样通用的框架,用于预测RNA序列与功能之间的关系,并据此设计具备预期功能的新型RNA分子。一个稳健的工具集,若能兼具功能预测与逆向设计能力,将大大加速RNA系统在科研与工业领域的应用落地。
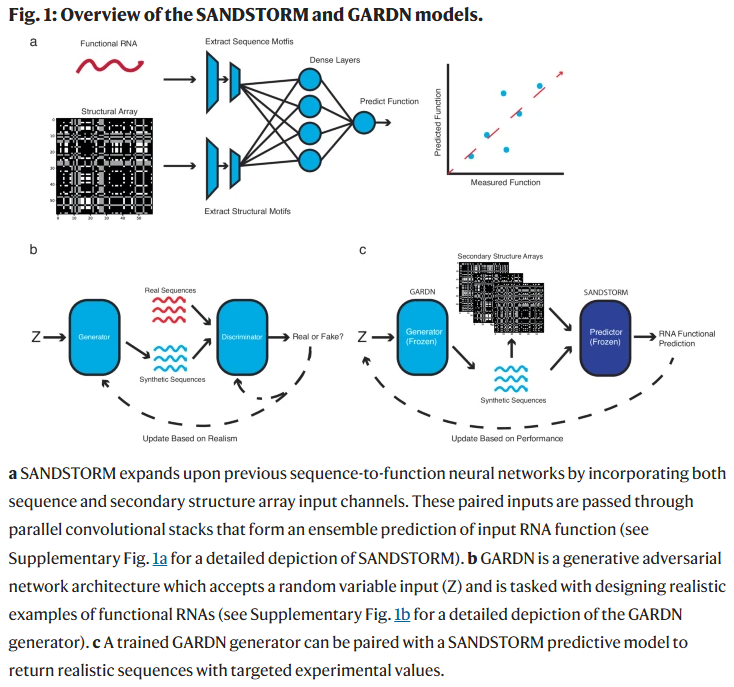
尽管RNA分子的功能受序列与结构共同影响,但目前尚无一种通用的深度学习框架能够同时整合这两类信息以提供功能预测。为此,研究人员提出了一种新的深度学习方法SANDSTORM,该方法融合RNA分子的序列与结构信息,用于预测多类RNA的功能活性。借助SANDSTORM神经网络,研究人员能够准确预测5′ UTR、RBS、CRISPR引导RNA以及Toehold开关核糖调控元件的功能表现。进一步地,研究人员将该预测模型与生成对抗RNA设计网络(GARDN)结合,成功生成了多类具有理想功能性质的新型RNA分子。该联合设计方法表明,SANDSTORM架构所学习到的二级结构抽象信息可被生成模型有效利用,从而生成符合实验热力学特征的新结构。具备在多种应用领域中对RNA序列进行精准预测与定向生成的能力,有望成为新一代计算RNA工具的重要基础。
结果
实现高效的RNA二级结构表示
为了在预测RNA功能时同时利用序列和结构信息,研究人员首先开发了一种RNA二级结构的表示方法,可与one-hot编码的序列并行输入至预测模型中。该方法灵感来源于图像分类领域,其中手工构建的特征已被能够自主学习数据抽象的深度卷积模型所取代。研究人员构建了一个基础数组,仅编码了潜在碱基配对位置,假设深度卷积神经网络(CNN)能够在训练过程中学习RNA分子的结构抽象信息。新提出的结构数组与以往用于RNA逆向设计的数组在概念上相似,但具备更高的时间效率,适合在深度学习流程中应用,并避免了传统结构预测算法所固有的假设。
通过模拟数据集学习可解释的结构信息
研究人员使用包含Toehold开关与若干干扰序列的模拟数据集,评估CNN将结构数组转化为可解释结构信息的能力。Toehold开关是一类核糖调控元件,其通过与互补RNA输入分子的结合,调控下游蛋白的翻译。由于该装置需要特定的序列和结构才能发挥作用,因而成为评估序列-结构联合输入模型性能提升的理想示例。该数据集包含标准Toehold开关以及四类干扰序列,分别为:仅包含RBS和随机序列;仅包含起始密码子;同时包含RBS和起始密码子但结构不符合Toehold开关特征;具备Toehold结构但缺乏必要序列模体的序列。
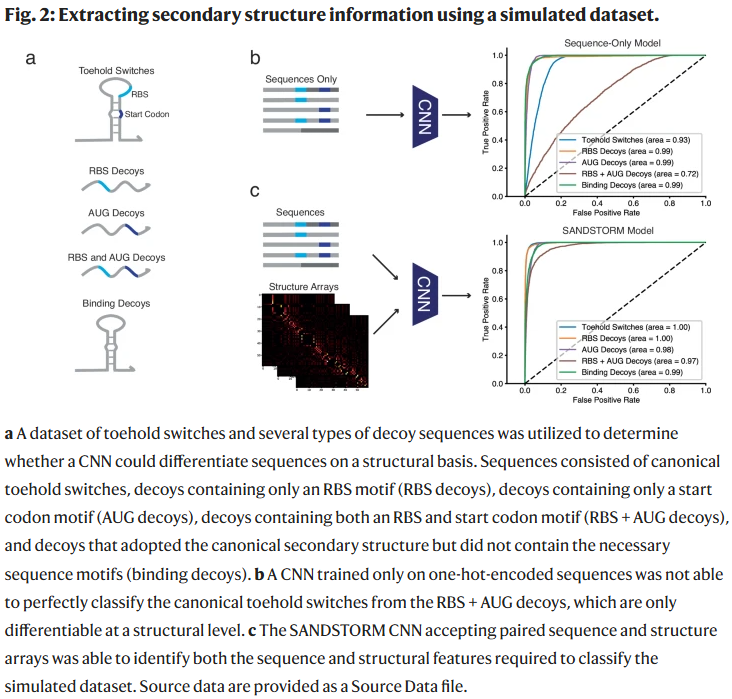
研究人员比较了仅接受one-hot编码序列的模型与SANDSTORM模型(同时输入序列和结构)的分类性能。如预期,仅输入序列的模型难以区分结构不符合要求的干扰序列,而SANDSTORM模型展现出显著优越的性能(AUC由0.72提升至0.97)。这验证了新结构表示的有效性,模型能够从中学习RNA结构抽象特征用于预测。
为了进一步验证分类模型确实识别了有意义的结构特征,研究人员采用integrated gradients技术分析了结构输入通道。对标准Toehold开关的分析显示,模型高度关注干扰茎区的碱基配对,并避免识别非特异性配对区域,该结果与最低自由能结构预测高度一致,说明模型能够正确识别关键结构模体。研究人员进一步假设,足够复杂的序列-only模型可能也能学习类似结构信息,于是通过扩大感受野和增加可训练参数的方式进行测试,发现性能虽有所提升,但始终低于SANDSTORM模型,进一步验证了结构输入的重要性。
SANDSTORM模型可预测多类RNA的功能
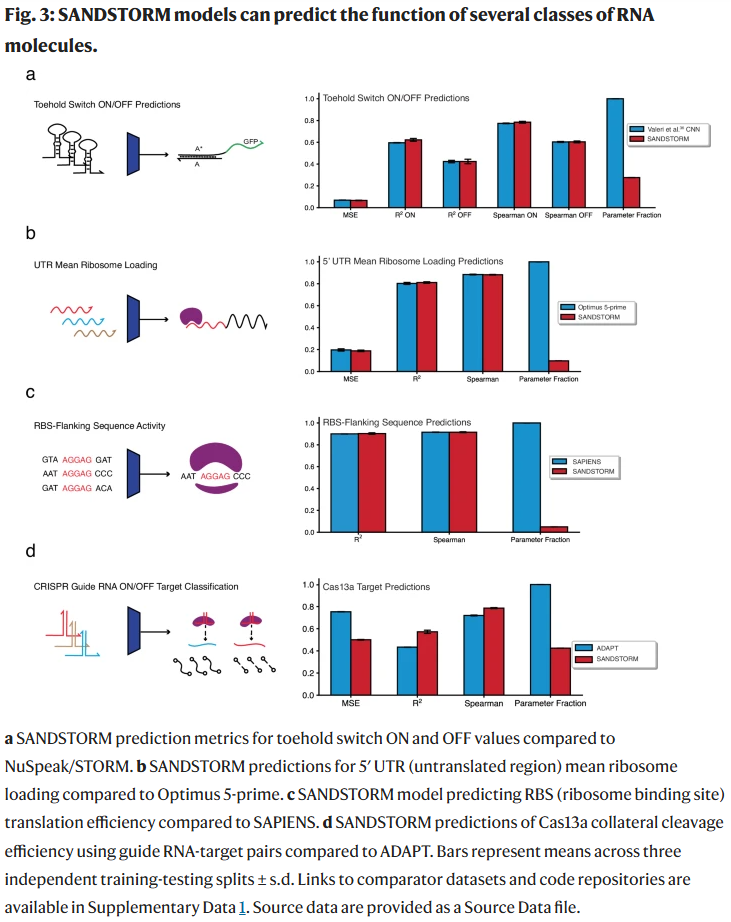
在验证SANDSTORM能有效利用结构信息后,研究人员进一步测试其在多种真实RNA功能预测任务中的通用性。为此,构建了统一的SANDSTORM架构,包括分别处理序列与结构的独立卷积通道,输出连接后输入全连接层。该模型参数量最小,未对不同任务进行特定调参。
Toehold开关预测:研究人员使用高通量荧光筛选数据进行训练,预测ON/OFF状态下的荧光强度比值。SANDSTORM模型在使用67%更少参数的情况下,可匹配以往最佳模型的预测性能。
5′ UTR预测:使用多核糖体分析数据,SANDSTORM模型在参数仅为原模型9.7%的情况下,预测精度基本一致,说明其在结构复杂、无特定模体的RNA中也具备强泛化能力。
RBS侧翼序列预测:预测影响翻译效率的RBS侧翼序列,SANDSTORM模型与原概率模型的预测性能相当,参数量减少了95%,并能捕捉调控区域对表达的显著影响。
CRISPR Cas13a预测:预测Cas13a系统中gRNA与靶RNA配对后的切割效率。SANDSTORM模型在仅使用42%参数的前提下,显著优于原始模型,展示了其在非翻译输出类任务中的推广能力。
构建RNA设计的生成式模型架构
在建立通用功能预测框架后,研究人员进一步开发了一个同样通用的生成模型——GARDN(Generative Adversarial RNA Design Network)。该模型可整合变异与保守模体、结构约束以及功能预测,适用于结构受限或非结构受限的设计任务。
5′ UTR设计:作为初步测试,研究人员训练了GARDN生成器并用SANDSTORM-UTR预测器优化输出,成功生成了结构与表达特性匹配的高表达序列。
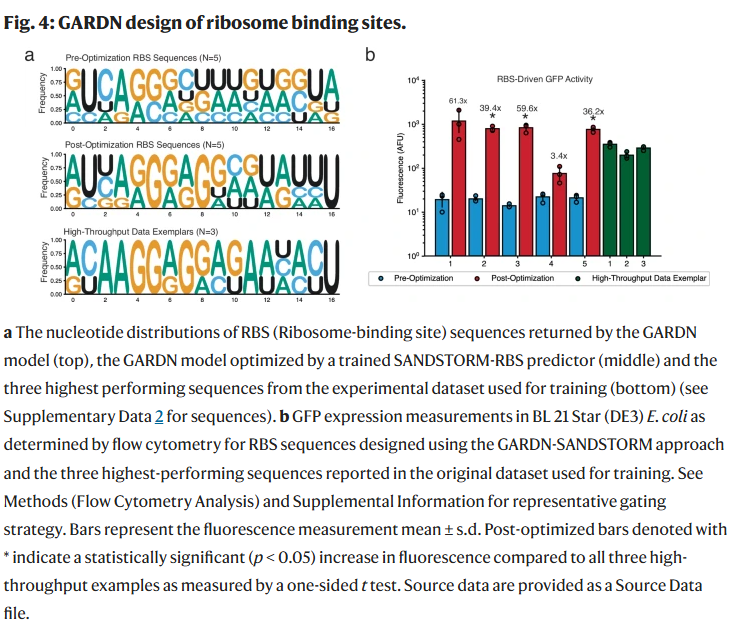
RBS设计:在加入Shine-Dalgarno模体(AGGAGG)要求的条件下,GARDN模型生成的序列在表达水平上优于未优化版本,实验验证表明优化后序列的表达强度提高了28.2倍,部分序列甚至超过训练集中最高表达的记录。
Toehold开关设计:Toehold设计最为复杂,需要序列与结构的双重满足。研究人员引入逆向互补机制,使生成模型可在拉丁空间中维持目标结构,并通过SANDSTORM优化ON状态。设计结果在结构一致性评估中表现优异,并复现了实验中高ON状态的特征序列。
与以往依赖于激活最大化的设计方法不同,GARDN通过端到端生成避免了事后修正带来的功能预测失真,保持了结构与功能的一致性。此外,GARDN生成的序列具有更好的核苷酸变异性与更快的生成速度,适用于诊断和合成生物等需要高多样性的应用场景。
通过与SANDSTORM结合优化,研究人员仅通过300次调用预测器即可将生成序列的预测ON值提升37%。不同结构复杂度的SANDSTORM变体验证了参数规模过大会造成优化过拟合,但不会影响最终序列的真实内容。这些结果表明GARDN-SANDSTORM组合在RNA结构保持、功能提升、模体保留以及适应多样化任务方面具备显著优势。
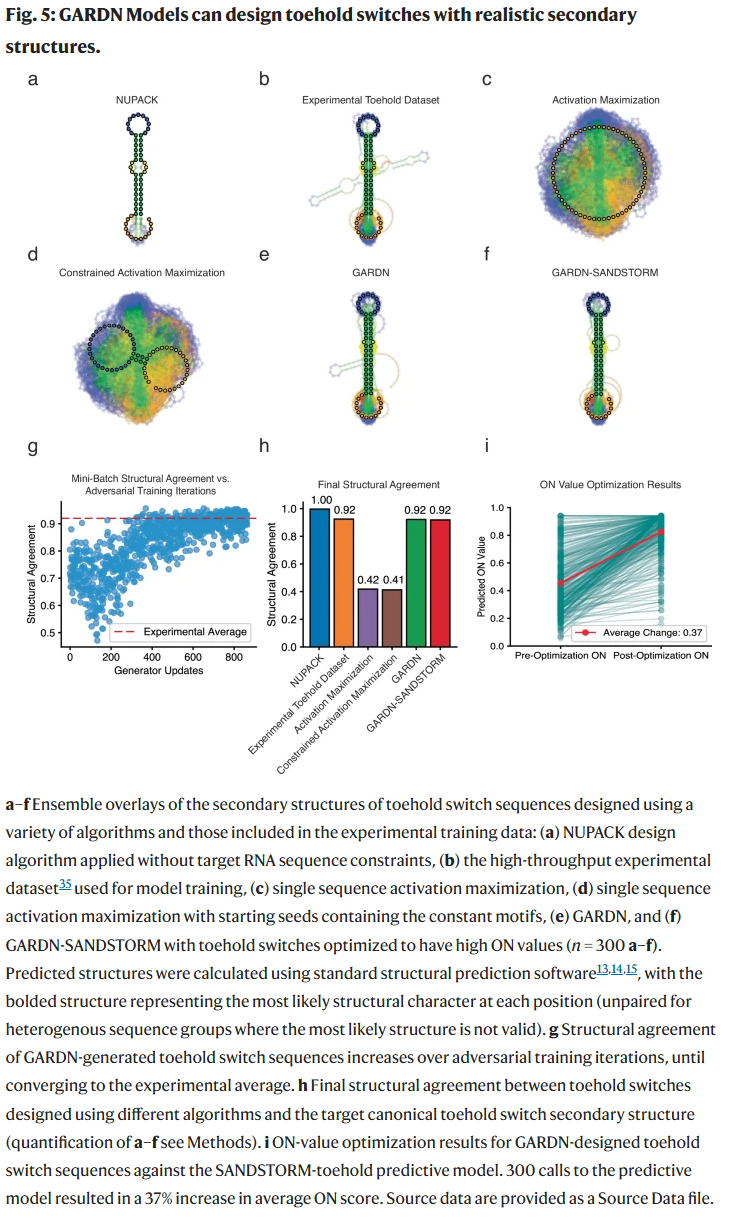
GARDN-SANDSTORM设计的Toehold开关的实验验证
为验证GARDN-SANDSTORM生成模型在RNA设计中的性能提升,研究人员对多组通过不同算法生成的Toehold开关序列进行了实验表征。这些组别包括:未优化的GARDN模型输出;使用ON状态GFP表达量优化的GARDN序列(基于仅预测ON状态的SANDSTORM模型);同时优化ON状态高表达和OFF状态低表达的GARDN序列(基于联合ON/OFF预测模型);以及使用NUPACK设计的序列。
为了进行公平比较,研究人员使用NUPACK核心算法仅设计60个核苷酸长度的开关序列,未纳入编码序列或RBS上下文信息,以便与训练数据保持一致。所有设计均在与训练数据相同的大肠杆菌体系中评估,ON状态使用带触发序列的RNA评估,OFF状态使用开关RNA自身评估。
与默认GARDN输出相比,使用ON状态SANDSTORM模型优化后的序列在与触发序列共存时的GFP荧光几何均值提高了3.8倍(仅调用预测模型300次,优化时间约为11秒)。这些ON优化序列的平均荧光强度比NUPACK设计序列高出4.8倍,说明该优化过程可显著提升RNA序列的特定功能属性。
相比之下,优化ON/OFF比值是一个更复杂的任务,因为预测器必须能传达序列在两个独立实验状态下的功能表现信息。而且,减少OFF状态泄露(如高GC含量产生稳定茎结构)所需的序列或结构特征,通常与增加ON状态表达所需的特征(如AU丰富的可展开结构)存在冲突。尽管存在此类权衡,GARDN-SANDSTORM通过联合ON/OFF模型优化的序列,其ON/OFF比值相较未优化序列提高了3.7倍,展示了该框架在多目标RNA功能优化中的强大能力。
此外,与NUPACK相比,未优化的GARDN输出和GARDN-SANDSTORM优化序列分别实现了3.8倍和11.9倍的ON/OFF比值提升,并且这一性能提升并未牺牲序列或结构的真实性。
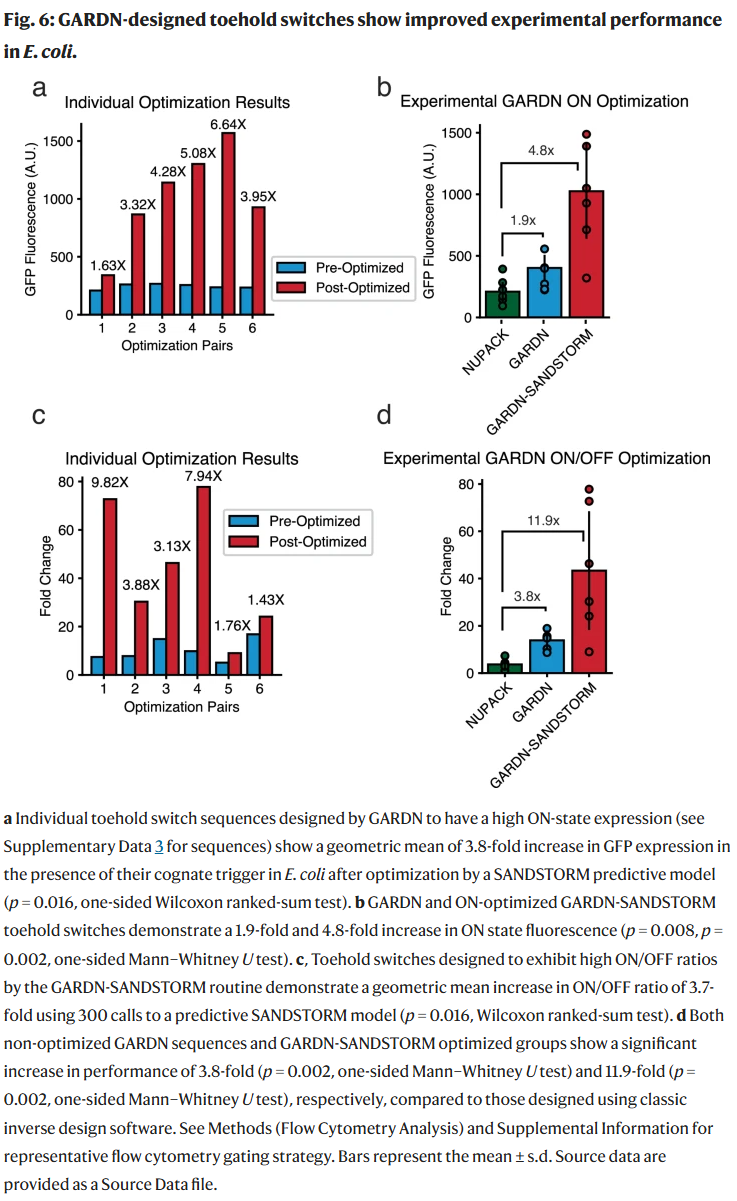
用于SANDSTORM训练的aptaswitch小型文库筛选
生成建模方法在分子设计中的应用通常受到可用实验数据量的限制,这在开发新型核糖调控元件时尤其显著,因为初期往往只有少数序列显示出预期调控效应。然而,不同RNA功能类别中往往存在共同的结构或模体设计。例如,可编程的RNA-RNA相互作用或与Toehold结构耦合的调控模体已被广泛应用于细胞内外系统中。
研究人员探讨了是否可通过在新领域中训练的预测模型来优化已有的GARDN模型,以实现新型aptaswitch的设计。为模拟无高通量数据的情形,研究人员构建了一个由384个序列组成的小型手动收集的文库,并以最近开发的aptaswitch系统为模板,该系统通过toehold触发机制将茎环结构转换为荧光适配体的构象。
与Toehold开关类似,aptaswitch的茎环结构会在与靶向RNA或DNA结合后解开,但与之不同的是,被释放区域会进一步形成新的结构以促进适配体折叠,并结合荧光配体产生信号。研究人员从Toehold开关数据集中抽取384个发夹结构,并通过替换下游结构为Broccoli适配体构建137-nt aptaswitch文库。该文库的ON/OFF比值变化达三个数量级,说明其功能表现与原始Toehold结构存在显著差异。
为在此变化背景下实施生成设计,研究人员在这些aptaswitch构象基础上训练了SANDSTORM预测模型。在数据量有限的条件下,传统深度学习模型极易过拟合,因此研究人员测试了SANDSTORM是否可在不使用迁移学习的前提下从零训练。通过100次数据划分实验发现,模型在其中37次成功收敛,具备一定的泛化能力。
为模拟实际诊断应用情景,研究人员以SARS-CoV-2 N基因的1230个30nt片段为靶标,设计了一个aptaswitch候选库,并使用训练好的SANDSTORM模型预测ON/OFF比值进行排名。结果显示,SANDSTORM排名前六的序列平均ON/OFF比值为62.46,约为排名最低六个序列的10倍,并且优于NUPACK Switch(38.59)和NUPACK Complex(56.25)两种热力学方法。这说明,即使数据极度稀缺,SANDSTORM也具备良好的序列筛选能力,且其运行速度远优于NUPACK(1秒 vs 34.7秒),更适用于快速响应如疫情暴发等紧急场景。
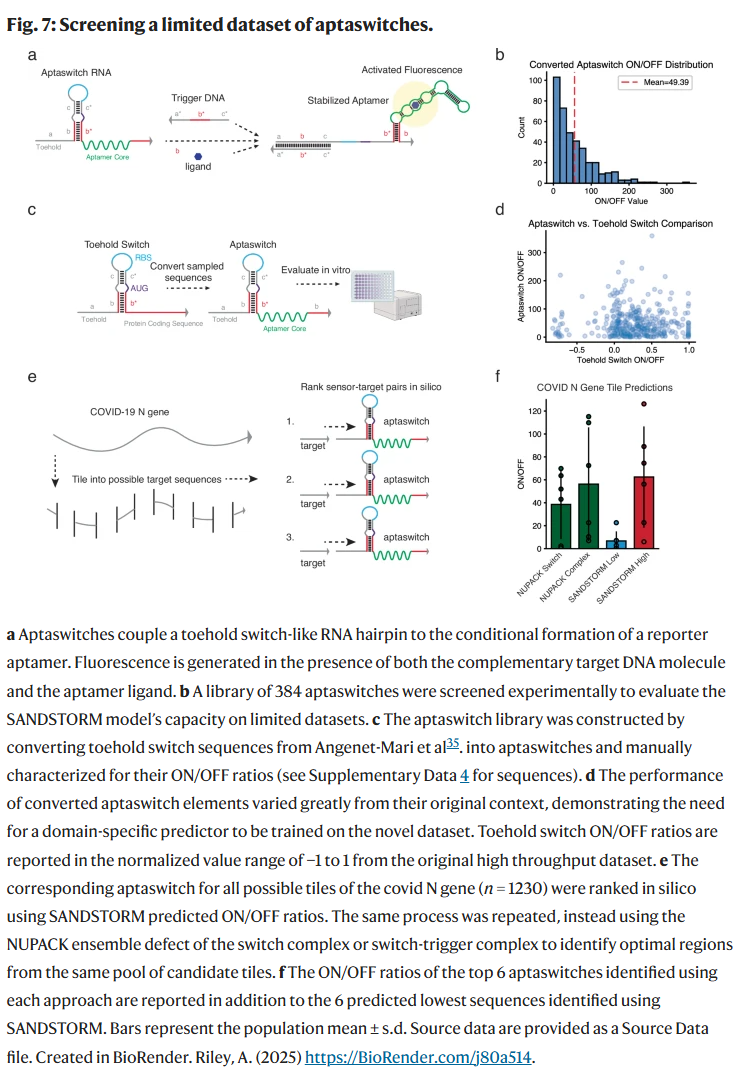
GARDN模型在aptaswitch上的设计应用
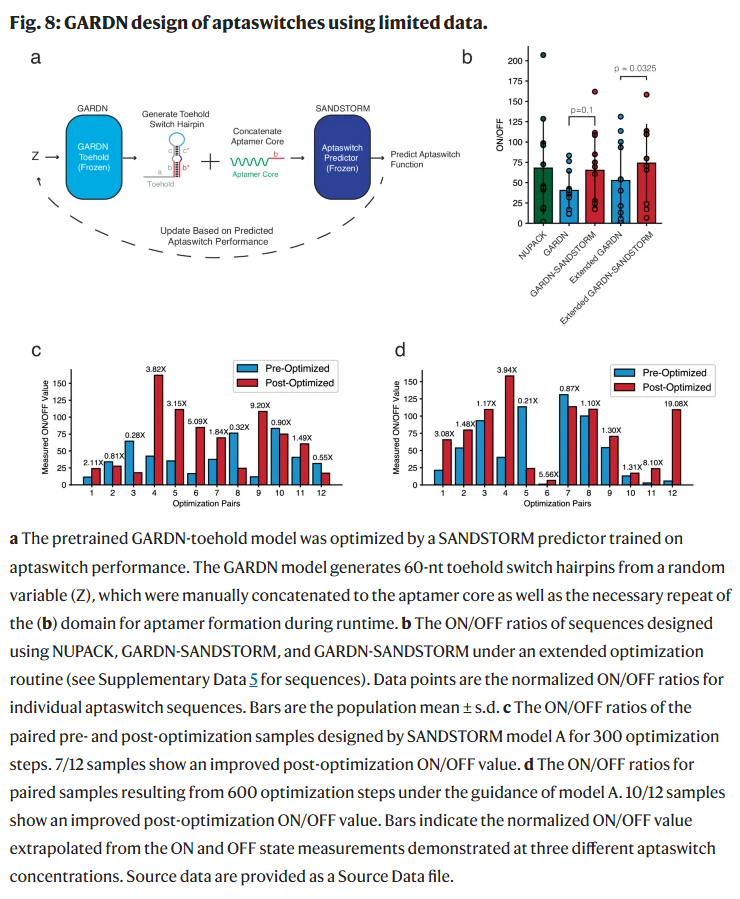
为进一步测试GARDN模型在新RNA设计任务中的适应能力,研究人员使用37个成功训练的SANDSTORM预测模型对GARDN生成的aptaswitch序列进行了优化。每个预测模型分别优化100条来自GARDN-toehold模型的序列,每条序列优化300步。最终,研究人员选定了在多模型中预测效果最优的模型A进行实验验证。
在模型A指导下,12对aptaswitch序列经优化后,平均ON/OFF比值由40.45升至65.21(提升1.49倍),尽管p值为0.1未达显著水平,但已呈明显趋势。相比之下,NUPACK设计的aptaswitch组平均比值为67.92,略高但差异不大。
进一步地,研究人员将优化步数由300增至600,结果显示平均比值升至74.02,显著高于初始组的52.57(p = 0.039),其中10/12序列显示优化效果。优化后序列的最高比值为161.9×与158.3×,远高于训练集中仅3.6%的高活性序列。这一结果表明,即使数据有限,GARDN-SANDSTORM也能生成高性能的RNA设计,具备出色的扩展能力。
讨论
研究人员展示了将RNA二级结构信息引入深度学习框架后,可以通过一个简洁而通用的卷积神经网络(CNN)架构实现RNA功能预测的最先进性能。此外,该模型不仅能够学习RNA结构的可解释抽象特征,其所构建的生成模型还可在功能优化的同时复现经过实验验证的热力学属性。这些属性直接转化为实验性能的提升,相较于现有的序列设计工具,GARDN在保持关键保守序列元素、核苷酸多样性和目标结构特征的同时,实现了更优的功能性RNA设计。
GARDN方法为设计结构真实、功能可控但无法通过传统热力学属性准确预测的RNA分子提供了强有力的解决方案,尤其适用于训练数据有限,或目标分子需以序列无关方式形成特定二级结构的设计任务。未来的研究可进一步探讨该设计范式是否因规避传统逆向设计工具的热力学假设,或因能整合高通量数据中额外信息而获得更优性能。GARDN-SANDSTORM联合优化框架在ON/OFF比值上的性能提升也超越了以往研究中激活最大化方法所达到的2.6倍提升,同时还缩短了优化时间,这可能得益于预测模型效率的提升和避免了对序列的人工后处理。
SANDSTORM和GARDN模型均可直接应用于其他RNA变体与其功能表现的映射数据中。其他领域如转录调控RNA元件、IRES样结构域、RNA-蛋白识别位点等也高度依赖RNA结构,因此非常适合利用SANDSTORM进行功能预测,并结合GARDN进行设计。尽管部署这些模型所需的最小序列数量尚无明确界限,aptaswitch实验结果表明,即使在数据稀缺条件下,这些工具仍可有效地支持新型RNA装置的设计。这一能力对于未来疫情响应等需要在数据有限条件下迅速开发诊断工具的场景尤为关键,也可作为辅助系统,用于设计含有Toehold开关模体的新型调控元件。SANDSTORM模型的高效性使其仅凭384个训练样本即可有效识别序列与功能之间的关系,为后续设计提供可行性基础。未来在数据稀缺环境下的研究可以进一步探索无监督筛选已充分训练模型用于下游设计,或尝试突破当前设计过程中对预测器调用次数(最多600次)的限制,以获得更优解。
研究人员预计,未来的工作将致力于在生物特征工程与深度学习特征抽象之间取得平衡,这种平衡正是SANDSTORM模型性能提升的关键因素。尽管通过针对不同任务微调双输入架构可进一步提升预测性能,但在生物场景中保持模型的通用性仍是设计机器学习系统的重要约束。GARDN-toehold生成器中所使用的“潜在空间反向互补”模块若能扩展至任意目标二级结构,将进一步提升该模型在多种RNA元件中实现可微分设计的能力。
本研究主要聚焦于长度不超过137 nt的短功能RNA序列,并使用了固定长度的数据集。随着RNA工具箱在序列长度与功能复杂性上的持续拓展,以及更多变长RNA文库的出现,未来还需应对随之而来的建模与生成设计挑战。
随着功能性RNA在各类生物技术应用中的广泛兴起,RNA设计的计算方法也必须同步发展。为了快速适配新任务而尽量减少实验筛选需求,可靠的功能序列生成工具将变得日益关键。SANDSTORM与GARDN模型为解决实验筛选瓶颈提供了有前景的解决方案,其具备在保持真实结构特征的前提下提升RNA分子功能表现的预测与设计能力,有望成为下一代计算RNA设计工具的核心基础。
整理 | WJM
参考资料
Riley, A.T., Robson, J.M., Ulanova, A. et al. Generative and predictive neural networks for the design of functional RNA molecules. Nat Commun 16, 4155 (2025).
https://doi.org/10.1038/s41467-025-59389-8
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢