DRUGAI
线粒体在能量生成和代谢中发挥关键作用,因此成为代谢工程和疾病治疗中具有前景的靶点。然而,尽管已知乘客蛋白会影响定位效率,但目前已被系统表征的线粒体定位标签仍然十分有限。为解决这一限制,研究人员利用变分自编码器(Variational Autoencoder, VAE)设计新型的线粒体靶向序列。计算分析显示,所生成肽段中有高达90.14%的比例具有功能性,并呈现出有助于线粒体定位的关键特征。研究人员在四种真核生物中对这些人工肽段进行了验证,并在概念验证实验中展示了其实际应用价值:通过代谢通路的亚细胞定位,3-羟基丙酸的产量得到提升,同时5-氨基乙酰丙酸合酶的递送效率分别提高了1.62倍和4.76倍。此外,研究人员还通过潜在空间插值探索了双重定位序列的进化起源。总体而言,该研究展示了生成式人工智能在基础线粒体生物学研究与实际应用中的潜力。

真核细胞是一个高度复杂的体系,包含多个细胞器,分别承担不同的细胞功能。这些细胞器由膜包裹,为众多由细胞核编码的蛋白质提供了适宜的理化环境,使其能有效发挥功能。因此,蛋白质的准确定位对于维持细胞结构、保障代谢过程的正常运行以及促进真核细胞高效运作具有重要意义。在自然系统中,蛋白质的亚细胞定位依赖于其氨基酸序列中复杂的信号。近年来,随着模式生物中定位标签的表征以及监督式机器学习模型的应用,研究人员在解析蛋白质定位机制方面取得了显著进展。这些技术进步大大提升了将蛋白质导向特定亚细胞区域的能力,推动了合成生物学、化学品生产和治疗干预等领域的发展。
线粒体在细胞代谢中的多重作用,使其成为代谢工程和疾病治疗中的重要靶点。作为真核细胞的“能量工厂”,线粒体参与多种关键细胞过程,包括能量合成、代谢调控以及细胞凋亡。此外,线粒体还是三羧酸循环的核心场所,并承担若干辅因子和代谢物的合成任务。借助线粒体这一底物富集环境,多个代谢通路已被成功定向至线粒体,以提升燃料、化学中间体和药物的产量。线粒体基质相比细胞质具有更高的pH、更低的氧浓度以及更强的还原环境,这些特性有利于外源生物催化元件的表达。此外,线粒体功能异常,尤其是源于线粒体DNA中的遗传性或获得性突变,与多种疾病密切相关,如Leber遗传性视神经病、线粒体脑病乳酸中毒伴卒中样发作综合征(MELAS)、Leigh综合征等。因此,提升线粒体靶向能力对于核酸酶和药物分子的高效递送也具有重要意义。
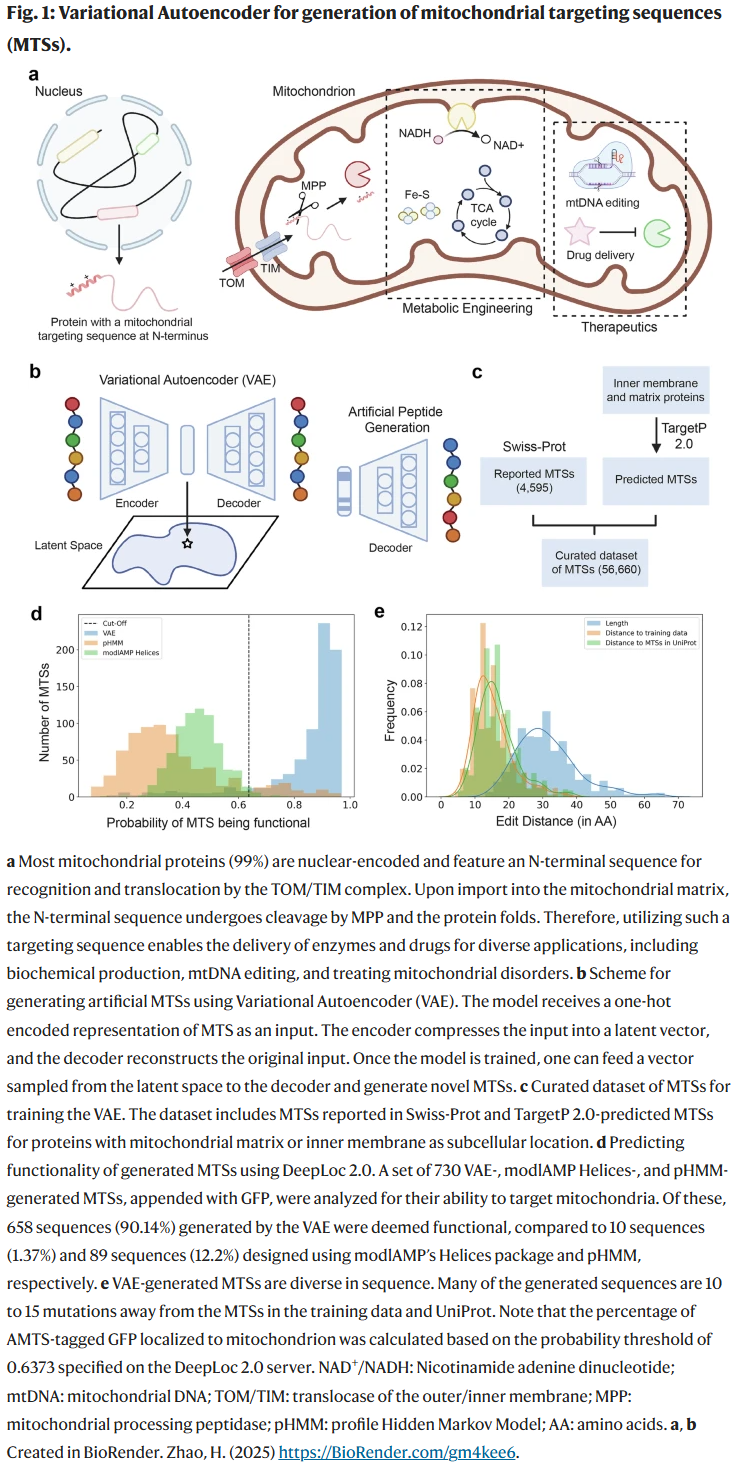
要将蛋白质定向至线粒体,需在核编码蛋白的N端添加一段靶向肽。这段带有正电荷且具两性特征的肽段可被线粒体外膜和内膜的跨膜转运复合体(TOM与TIM复合物)识别并导入线粒体基质。在进入基质后,该肽段会被线粒体加工肽酶切除,随后目标蛋白折叠成其成熟结构。然而,目前被系统表征的靶向肽数量有限,导致相关序列在工程应用中被反复使用。考虑到靶向效率与所携带的“乘客蛋白”密切相关,使用非最优的线粒体靶向序列(MTS)可能导致目标酶导入不足甚至完全失败,从而造成部分或无效的通路定向。此外,频繁使用同一内源靶向序列可能导致转运机制饱和,引发与内源蛋白的竞争,并可能对线粒体蛋白合成与完整性造成不良影响。此外,在同一菌株中重复使用高度相似的靶向序列用于多个基因表达时,可能因同源重组而导致遗传不稳定。因此,构建一个多样化且具有功能性的线粒体靶向序列工具箱具有重要价值。
结果
构建用于人工线粒体靶向序列设计的变分自编码器
线粒体靶向序列(MTSs)是一类带正电荷的肽段,具有形成两性α螺旋的倾向。这些肽段通常位于目的蛋白的N端,长度为10–120个氨基酸,典型长度约为35个氨基酸。与过氧化物酶体等细胞器不同,MTSs 并不包含特定的序列模体,其导入过程依赖于理化性质和结构特征。这一特性使得构建多样化的MTS肽段库变得具有挑战性,原因包括巨大的设计空间(对于35个氨基酸长的序列,空间为20³⁵)、缺乏明显的共识序列以及与乘客蛋白之间不可预测的关系。
深度生成模型具有学习复杂非线性映射和捕捉底层数据分布的能力,因此可用于从序列信息出发设计具备特定功能的全新肽段与蛋白质。为此,研究人员利用变分自编码器(VAE)生成自然界未见的MTS序列。VAE由两个部分组成:编码器将输入序列转换为潜在空间表示,解码器则将潜在向量重构为原始序列。该模型通过瓶颈结构捕捉关键特征,并在训练过程中优化重构损失及Kullback-Leibler(KL)散度损失,使潜在空间分布逼近多元高斯分布。训练完成后,可从连续潜在空间中采样向量并通过解码器生成新序列,从而实现类自然序列的设计。
为提升VAE的训练效果并覆盖更全面的设计空间,研究人员构建了一个MTS数据集。首先,基于Swiss-Prot数据库中以转运肽注释为线粒体定位的蛋白,收集了4984条MTS序列,其中4595条为非冗余序列。这些序列或来自实验验证,或通过序列比对及预测工具(如MitoFates、Predotar、TargetP)得出。该初始数据集较小且偏向模式生物,为增强多样性,研究人员进一步利用先进的注意力机制深度学习模型TargetP 2.0,对Swiss-Prot和TrEMBL数据库中线粒体基质及内膜定位的蛋白进行MTS预测。考虑到TargetP 2.0在植物与非植物物种上使用不同模型,研究人员通过UniProt中的分类信息将蛋白序列进行分组并分别预测,随后筛选出符合长度(11–69 aa)、氨基酸合法性及去除冗余后的有效肽段,共构建出包含56,660条MTS的训练集。
在训练过程中,为标记剪切位点,研究人员在每条序列C端添加“$”符号。考虑到肽段长度分布偏斜,数据集以9:1的比例进行分层划分,确保训练集与验证集之间具有相似的长度分布。所有肽段统一填充至70个字符,并通过One-hot编码输入编码器。起初,研究人员基于循环神经网络(RNN)构建VAE,该架构曾用于抗菌肽的生成任务。但训练后发现,模型常常从不同潜在向量生成相同序列,且含有重复氨基酸、突变较少,说明序列多样性较低。随后,研究人员改用全连接网络结构实现VAE的编码器与解码器,并未观察到此类问题,因此选用该架构用于后续分析。
计算分析显示生成的MTS序列具有功能性、多样性,且自然界中未曾存在
基于训练好的VAE模型,研究人员生成人工肽段,并对其功能性、多样性、理化性质及结构特征进行系统评估。从标准正态分布中采样1000个潜在向量,经解码器生成730条具有合法氨基酸序列的新MTS。为验证其定位能力,研究人员将其前置于不含起始甲硫氨酸的绿色荧光蛋白(GFP)前方,并使用DeepLoc 2.0预测其亚细胞定位。结果显示,90.14%的人工肽段被预测能将GFP导向线粒体。
为验证性能优势,研究人员将这些MTS与基于隐马尔可夫模型(pHMM)以及modlAMP的螺旋肽模块所设计的MTS进行对比。将其前置于GFP后,只有12.2%的pHMM生成肽段被预测为功能性,而modlAMP生成肽段中仅1.37%可定位至线粒体。此外,研究人员评估了人工序列中是否包含线粒体外膜转位酶TOM20识别模体 φχβφφ(φ为疏水残基,χ为任意残基,β为碱性残基),结果显示,730条人工序列中有514条(70.4%)含有该模体,提示其可能通过TOM20介导的导入机制。
研究人员进一步计算生成序列与训练集MTS或UniProt数据库中已知MTS之间的Levenshtein距离,评估其多样性。结果表明,人工MTS平均与任何自然MTS相差10–15个氨基酸,展示出良好的结构多样性。
模型生成序列覆盖自然MTS空间
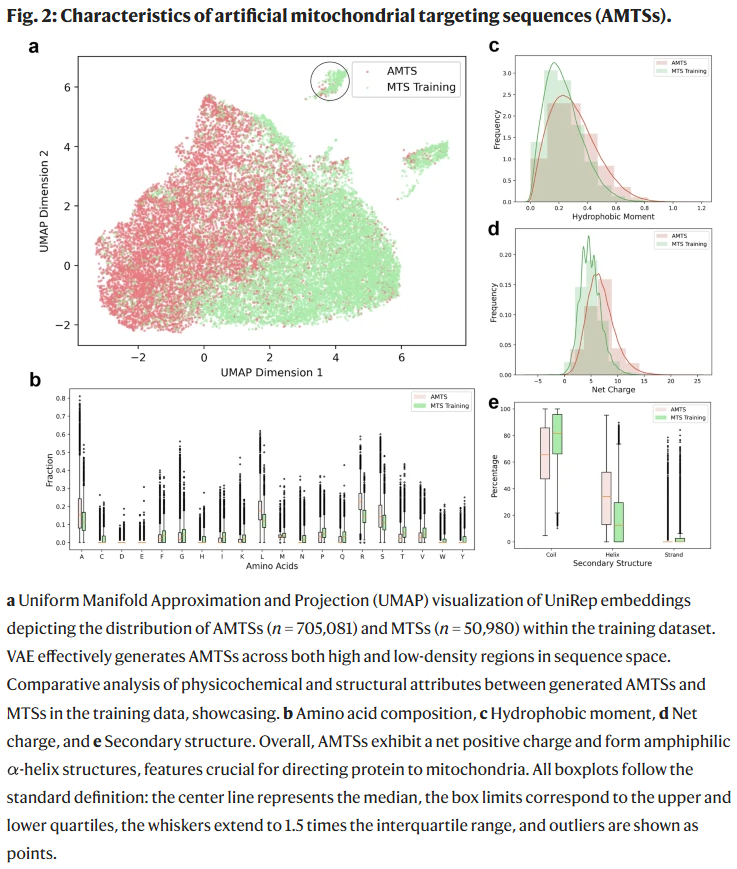
研究人员进一步评估了VAE模型对自然MTS序列空间的覆盖能力。共生成1,000,000条人工MTS,并筛选出合法氨基酸序列。随后,采用CD-HIT算法对训练集中和VAE生成的MTS进行聚类,选取序列相似度低于30%的代表性自然与人工序列。接着,利用预训练蛋白表示模型UniRep对其进行嵌入,并通过UMAP将高维表示压缩为二维进行可视化分析。可视化结果显示人工序列能够较好地覆盖自然序列空间,形成三个明显的聚类簇。其中一个聚类簇缺乏起始甲硫氨酸,这一差异源自部分UniProt训练序列本身的标注缺失。其余两个聚类簇中人工肽段在理化和结构特性上与自然MTS无明显差异。
人工与天然MTS的组成与结构特征对比
研究人员进一步分析了人工与天然MTS在氨基酸组成、理化特性及二级结构方面的异同。通过BioPython计算氨基酸频率,结果显示人工MTS中富含丙氨酸(A)、精氨酸(R)、亮氨酸(L)和丝氨酸(S);而天冬氨酸(D)和谷氨酸(E)等带负电残基则普遍偏低,从而使人工MTS总体呈正电荷状态,这一特性符合功能性MTS的要求。
此外,使用modlAMP计算的疏水矩(hydrophobic moment)略高于训练集,表明人工序列两性性稍强。Eisenberg尺度下的疏水性略偏负,而GRAVY指数显示与训练集一致。在肽段长度分布上,人工MTS亦覆盖了训练数据的范围。
在二级结构方面,研究人员借助S4PRED模型预测α螺旋、β折叠与coil(无序结构)的比例。人工MTS显示出更高比例的α螺旋倾向,伴随着coil比例下降,进一步验证了其作为MTS的功能潜力。
用于体内验证的序列筛选策略
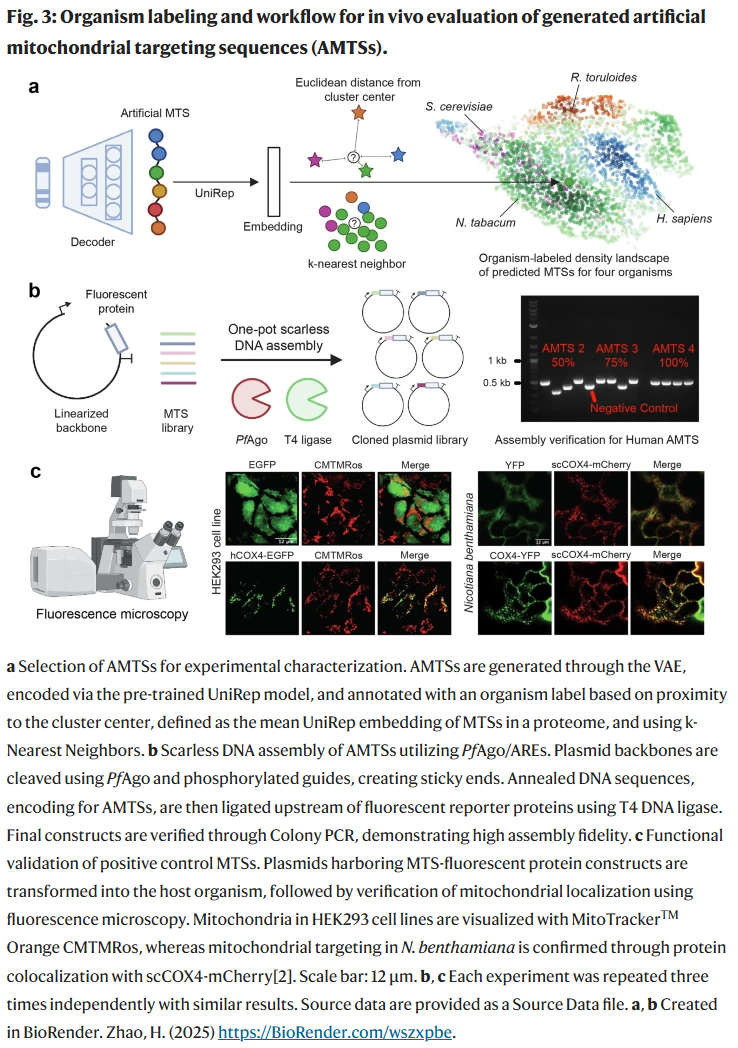
在体外预测取得高准确性后,研究人员筛选出9条TargetP 2.0预测得分高于0.9的VAE生成序列(AMTS),用于在酿酒酵母(S. cerevisiae)中体内验证。采用Gibson组装法将这些MTS连接于GFP N端,同时构建以COX4的内源MTS为阳性对照。
共聚焦显微镜显示,GFP自身无法定位至线粒体,而COX4-GFP则能与线粒体染料MitoTracker重合。在9条AMTS中,AMTS 131、205、225和335成功靶向线粒体;AMTS 6、64和96部分定位于线粒体及其他胞器;AMTS 110与245则未显示线粒体靶向能力。
潜在空间插值实现双胞器靶向肽的设计
在先前的分析中,研究人员注意到部分生成序列(27条)被预测为定位于叶绿体,另有17条同时预测定位于线粒体与叶绿体。最初怀疑这可能源于数据库标注错误或TargetP 2.0预测误差。然而,线粒体与叶绿体靶向序列在进化上具有共同起源,5%植物胞器蛋白天然具备双重定位能力,这些信号序列往往表现出MTS与CTS之间的中间特征。
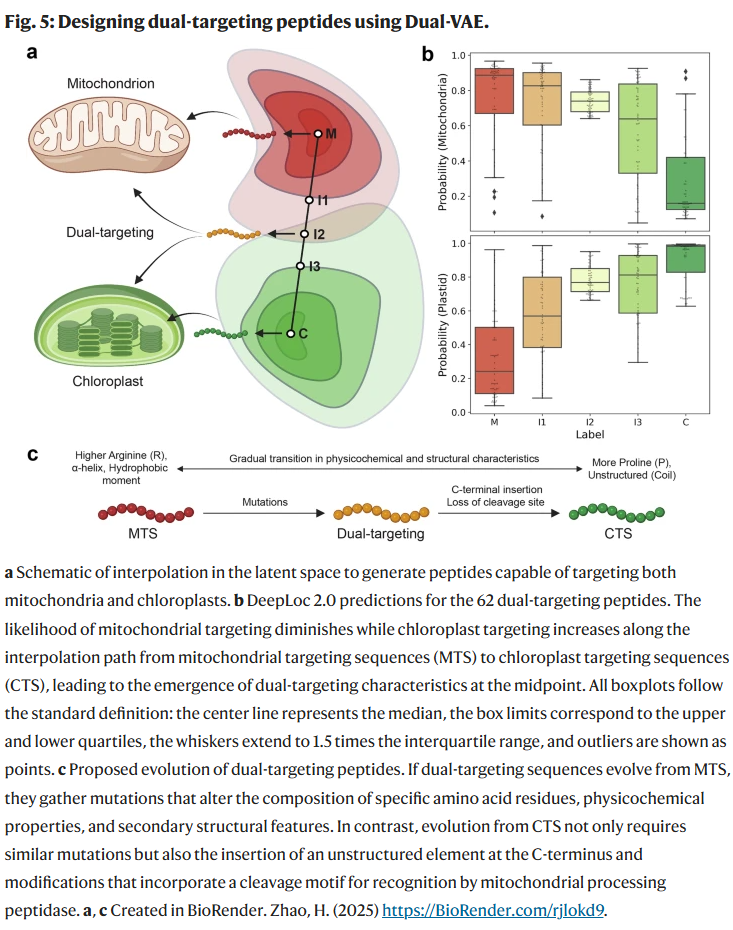
因此,研究人员尝试通过潜在空间插值探索这类双靶向肽的设计路径,并推测其进化轨迹。研究人员重新训练一个Dual-VAE模型,输入为Viridiplantae门中来源于UniProt或TargetP 2.0预测的MTS与CTS序列。结果显示模型潜在空间中形成两个不同的分布簇。
接着,研究人员使用K-Means算法提取MTS与CTS簇中心,在欧氏距离阈值0.4内选取序列对进行线性插值。共生成50组序列,每组包含三个插值点,并用DeepLoc 2.0预测其亚细胞定位功能。结果发现,插值路径上的序列在定位能力上呈现从线粒体到叶绿体的平滑过渡,其中62条序列显示出强烈的双重靶向倾向。
研究人员进一步比较这些双靶向序列的理化特性变化:在插值过程中,丝氨酸(S)与亮氨酸(L)含量上升,而精氨酸(R)与丙氨酸(A)下降,肽段长度逐渐增加;整体疏水矩下降,coil结构比例上升,α螺旋减少,表明其具有双靶向信号的中间性质。
此外,双靶向肽的C末端保留了RR、R-2或R-3剪切位点模体,提示其具备在线粒体内被正确剪切的潜力。综合分析支持双靶向序列可能是由线粒体靶向序列进化而来。
讨论
线粒体在能量代谢、大分子前体与还原当量的生物合成、以及代谢废物的高效管理中发挥着关键作用。因此,线粒体靶向在代谢工程和治疗领域中具有巨大潜力。然而,尽管线粒体在细胞代谢中至关重要,当前被系统表征的线粒体靶向序列(MTS)数量仍然有限。这种稀缺性限制了将蛋白精确导向线粒体的可选方案,可能导致靶向效率不足,进而影响载荷蛋白或代谢通路的功能。这些限制凸显了设计并系统表征多样化、功能性MTS的迫切需求。
为应对这一挑战,研究人员开发了一个深度生成式人工智能框架,用于设计人工线粒体靶向序列。研究人员首先整理了一个涵盖自然界中MTS的大型数据集,并在这些肽段序列上训练了变分自编码器(VAE)。随后,研究人员利用DeepLoc 2.0验证了所生成MTS的功能性,并通过对其理化性质和结构特征的详细分析发现,这些序列带有正电荷、具备两性结构,且倾向于形成α-螺旋,符合线粒体靶向所需的关键特征。
在此基础上,研究人员设计了一套采样策略,用于优先选择候选MTS进行实验验证。利用共聚焦显微成像,研究人员在四种真核生物中共体内验证了41条自然界未出现过的新型肽段,其靶向成功率达50–100%。此外,研究人员还在HEK293细胞系中确认了这些人工肽段可被成功剪切。
进一步分析发现,部分生成的序列具有潜在的双重靶向能力,即同时定位至线粒体和叶绿体。因此,研究人员训练了另一个模型(Dual-VAE),其训练数据来自绿色植物界(Viridiplantae)中的MTS和CTS(叶绿体靶向序列)。随后,研究人员在潜在空间中进行线性插值,生成了62条假设具有双靶向能力的肽段。通过分析插值路径中肽段的特征变化,研究人员揭示了这类双靶向序列可能源自线粒体靶向肽的进化轨迹。未来,研究人员预计这些肽段的功能验证将有助于深入理解植物中的双重定位机制,并在利用叶绿体与线粒体中乙酰辅酶A共源供体的前提下,提升诸如紫杉醇等高价值化学品的产量。
此外,研究人员还展示了这些人工MTS在代谢工程和蛋白递送中的实际应用价值。在第一个实例中,研究人员将β-丙氨酸代谢通路中的酶靶向至线粒体,实现了3-羟基丙酸产量较细胞质导向提高了1.62倍。在第二个实例中,通过构建嵌合MTS,研究人员提升了HEM1酶的线粒体靶向效率,达到了4.76倍的改进。进一步分析表明,在串联构建中MTS的顺序与数量对靶向能力提升具有重要影响。
模型结构本身仍有进一步提升空间。例如,可以采用条件变分自编码器(Conditional VAE)将物种标签与目标蛋白信息直接作为输入条件,或将该问题转化为序列到序列的翻译任务,以减少对选择性采样策略的依赖。这种方式有望更好地捕捉导入机制的偏好性以及MTS与“乘客蛋白”之间的内在关联。
除了引入元信息,还可借助高级特征工程方法——如利用理化属性、预训练大型语言模型(LLMs)、或引入结构层级信息——以更全面地建模氨基酸间长距离相互作用。此外,为特定物种构建人工MTS时,可通过提取VAE潜在空间中靠近其MTS质心的向量作为输入,从而生成定制化的人工靶向肽段。
开发高通量筛选平台将进一步促进本研究所设计的MTS的系统表征。目前,研究人员采用荧光蛋白标记结合活细胞共聚焦显微成像进行定位验证,这虽是细胞生物学中常用的重要手段,但不适合规模化地定量靶向效率。未来可引入基于自组装Split-GFP技术、流式细胞术(FACS)及高通量测序的定量方法,构建序列与靶向效率之间的高质量关联数据集,并用于快速筛选最适合特定蛋白的MTS。
此外,研究人员可利用该数据集训练监督学习模型,在体外快速筛选MTS或嵌合序列。然而,从HEM1定位实验来看,交换N端MTS会显著影响mRNA水平。因此,更合理的策略是采用“密码子语言模型”表示MTS序列,以同时捕捉其转录效率与靶向效率。
在机制层面,也需要深入研究嵌合MTS提升靶向效率的原因。近期研究表明,蛋白序列中的内部MTS-like信号可增强某些蛋白的导入效率。因此,未来值得探索嵌合结构中靠后的MTS是否通过与Tom70受体相互作用增强了前蛋白的线粒体导入能力。
总之,研究人员构建的VAE模型在设计多样化、功能性强的线粒体及双靶向序列方面表现出色,为实现特定载荷蛋白的高效导入、推进代谢工程和生物医学应用提供了强有力的技术支撑。
整理 | WJM
参考资料
Boob, A.G., Tan, SI., Zaidi, A. et al. Design of diverse, functional mitochondrial targeting sequences across eukaryotic organisms using variational autoencoder. Nat Commun 16, 4151 (2025).
https://doi.org/10.1038/s41467-025-59499-3
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢