DRUGAI
今天为大家介绍的是来自法国索邦大学Amir Pandi与Ariel B. Lindner团队的一篇论文。遗传密码具有简并性,这意味着多种不同的DNA序列可以编码同一种蛋白质。然而,为特定生物体优化密码子(codon)使用时,可能的组合数量庞大,使得这一任务极具挑战性。幸运的是,自然界中经过进化优化的序列为机器学习算法提供了宝贵的学习资源。作者提出了CodonTransformer,这是一个多物种深度学习模型,它通过分析来自164个涵盖生命所有领域生物体的超过100万对DNA-蛋白质序列进行训练。借助Transformer架构和创新的序列表示方法(结合了生物体、氨基酸和密码子编码),该模型展现出优秀的上下文理解能力。CodonTransformer能够生成针对特定宿主的DNA序列,这些序列不仅具有接近自然的密码子分布特征,还最大限度地减少了不良的顺式调控元件。研究团队还提出了共享Token表示和编码与对齐多重掩码(STREAM)策略,并开发了一个可定制的开源模型和用户友好的Google Colab界面,使研究人员能够轻松进行密码子优化,为基因设计和合成生物学提供了实用工具。

遗传密码是由64个三核苷酸密码子组成的通用系统,它指导细胞如何从基因组中产生蛋白质。这套系统具有简并性,意味着大多数20种氨基酸可以由多个不同密码子编码。有趣的是,不同生物体对这些同义密码子的使用频率各不相同,这种差异源于细胞内tRNA丰度、蛋白质折叠调控和进化压力的不同。这种同义密码子的偏好选择被称为密码子使用偏好,是区分不同物种的重要特征。在设计用于异源基因表达的DNA序列时,考虑密码子使用偏好至关重要。密码子优化是指调整DNA序列中的同义密码子,使其与宿主生物体的偏好相匹配。随着无模板DNA合成成本的下降和蛋白质从头设计技术的快速发展,密码子优化的需求日益增长。然而,探索同义密码子排列的组合空间几乎是不可能的任务。以一个含300个氨基酸的普通蛋白质为例,可能的组合数量高达10的150次方。传统优化方法往往依赖选择高频密码子(可能导致资源耗尽和蛋白质聚集)或在随机位置插入稀有密码子(可能导致蛋白质错误折叠和核糖体停滞)。这些方法不仅应提高目标蛋白质表达,还应避免扰乱宿主tRNA池而引起毒性。
深度神经网络凭借学习复杂数据模式的能力,为破译密码子使用规律提供了新思路。本研究提出了一种基于Transformer架构的密码子优化方法,利用来自164个生物体的约100万对基因-蛋白质序列作为训练数据。这不仅扩大了数据规模,还通过单一模型实现了针对特定物种的DNA设计。为解决生物体特异性的上下文识别问题,研究团队开发了结合生物体编码与氨基酸-密码子对的序列表示策略,并提出了共享Token表示和编码与对齐多重掩码(STREAM)策略。基于此,他们开发了CodonTransformer,这是一个学习多物种密码子使用模式的模型。研究人员在15个基因组中具有最高密码子相似性指数(CSI,衡量密码子使用模式相似度的指标)的基因上微调了模型。结果表明,CodonTransformer能生成具有类似自然分布的密码子序列,同时最小化有害调控元件的数量。为方便科研人员使用,团队提供了开源的基础和微调模型,以及一个完整的Python包,简化从数据处理到序列评估的整个工作流程。他们还开发了用户友好的Google Colab界面,让研究人员能轻松设计密码子优化的DNA序列,为合成生物学和基因工程提供了实用工具。
模型架构
密码子优化本质上是一个“翻译”问题——将蛋白质序列“翻译”回DNA序列。传统机器翻译通常采用两种方法:一种是先将内容编码后再解码到目标语言,另一种是直接用提示词引导模型完成翻译。这两种方法都是一次生成一个单元,从头到尾依次进行。然而,在密码子优化中,这种逐步生成的方法存在问题:序列前部分的密码子选择会影响后部分,而一旦选择了某个密码子就无法更改。因此,研究团队采用了双向优化策略,使用“仅编码器”架构和“掩码语言建模”(MLM)方法,允许序列的所有部分同时被优化。
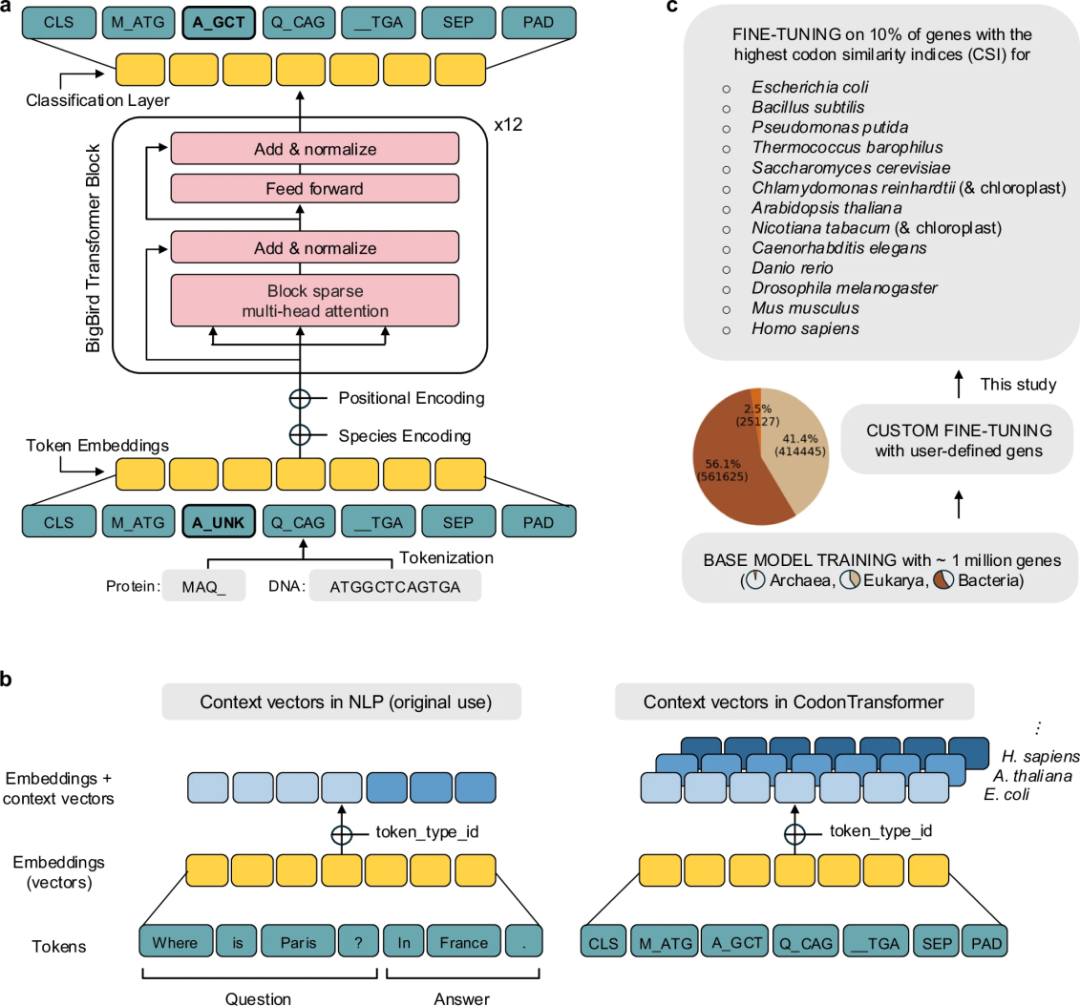
图 1
如图1所示,研究人员设计了一种特殊的表示系统,其中每个氨基酸可以与特定密码子关联或保持“未知”状态。例如,“A_GCC”表示由密码子GCC编码的丙氨酸,而“A_UNK”表示丙氨酸但不指定具体密码子。训练时,部分信息被隐藏;推理时,模型会为所有“未知”密码子提供最优选择。模型基于BigBird Transformer架构,这是一种能处理长序列的深度学习结构。为了让模型能够适应不同生物体的密码子偏好,研究人员巧妙地利用了Transformer的上下文标记功能,为每个物种分配独特的标识符。这使模型能学习各物种特有的密码子使用模式,同时允许用户为任何序列指定目标生物体。
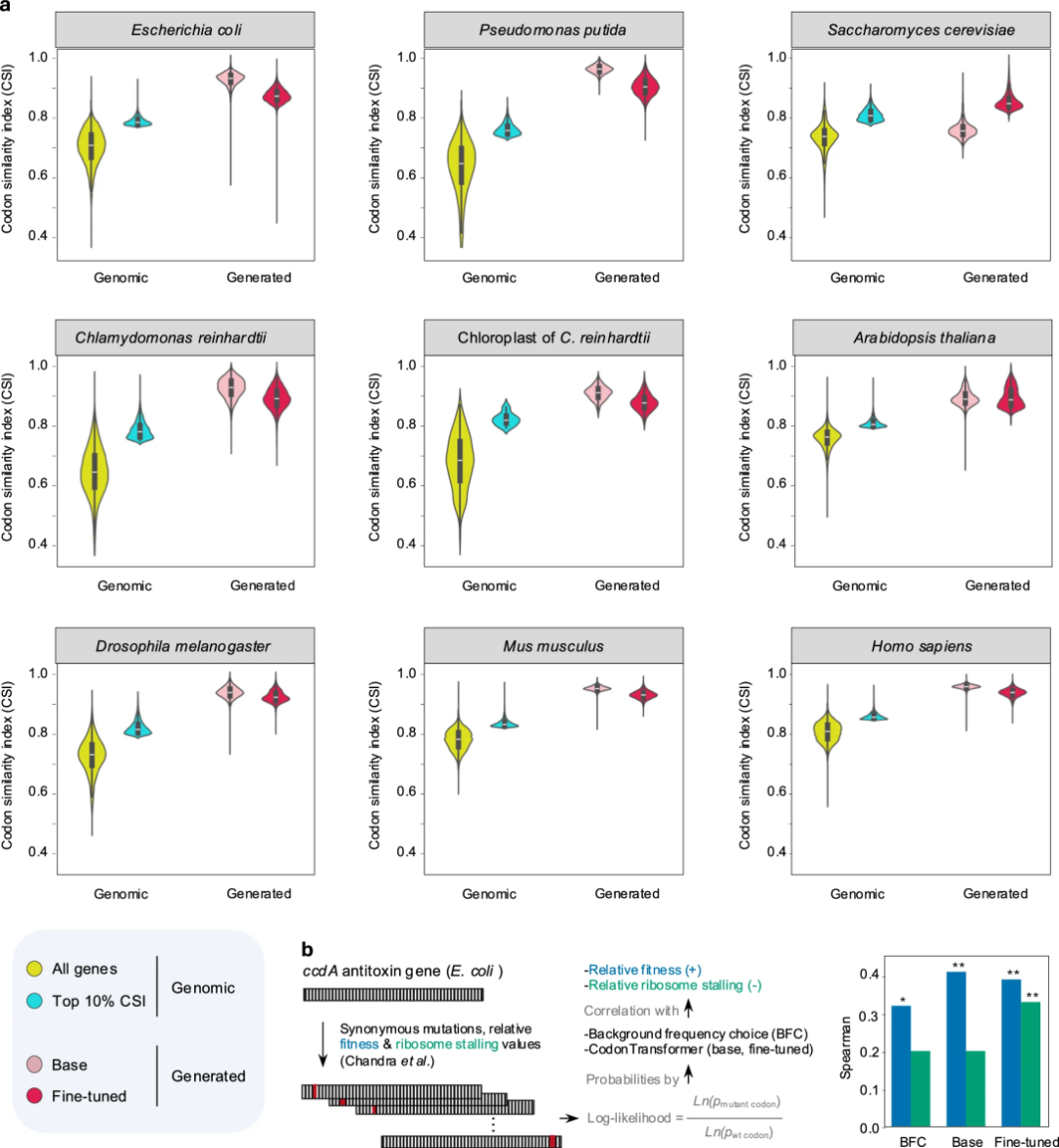
图 2
如图2所示,基础模型CodonTransformer使用来自164个生物体的约100万个基因进行训练,涵盖了细菌(56.1%)、古菌(2.5%)和真核生物(41.4%)。这个模型可以直接用于跨物种密码子优化,也可以在特定基因集上进行精细调整,以适应更具体的需求。研究团队在15个不同生物体(包括大肠杆菌、酵母菌、拟南芥和人类等)中表达效率最高的10%基因上进行了模型微调。他们使用密码子相似性指数(CSI)作为衡量标准,这是一种比传统指标更适合多物种比较的度量方式。
实验结果
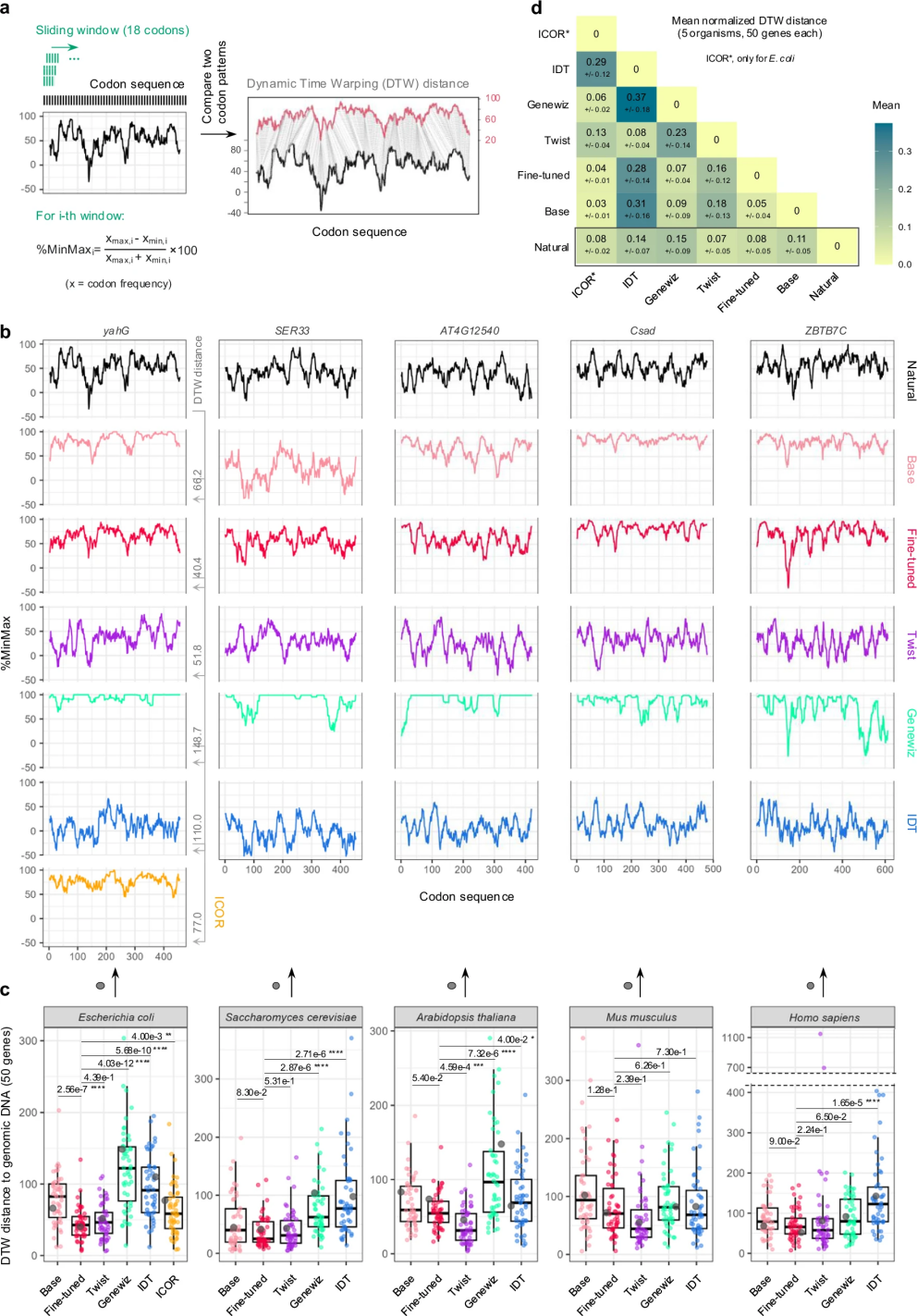
图 3
CodonTransformer模型成功生成了具有自然密码子分布特征的DNA序列。研究团队通过%MinMax指标(一种衡量密码子分布模式的方法)评估了模型表现,图3展示了不同工具生成序列的比较结果。通过分析五个模式生物的50个高表达基因,研究发现微调后的CodonTransformer和Twist生成的序列最接近自然DNA的密码子分布模式,明显优于其他商业工具。此外,CodonTransformer生成的序列在RNA折叠能量和GC含量等关键特性上也与自然序列高度一致,证明该模型能全面模拟自然DNA序列的多种特征,为基因设计和合成生物学提供了有力工具。
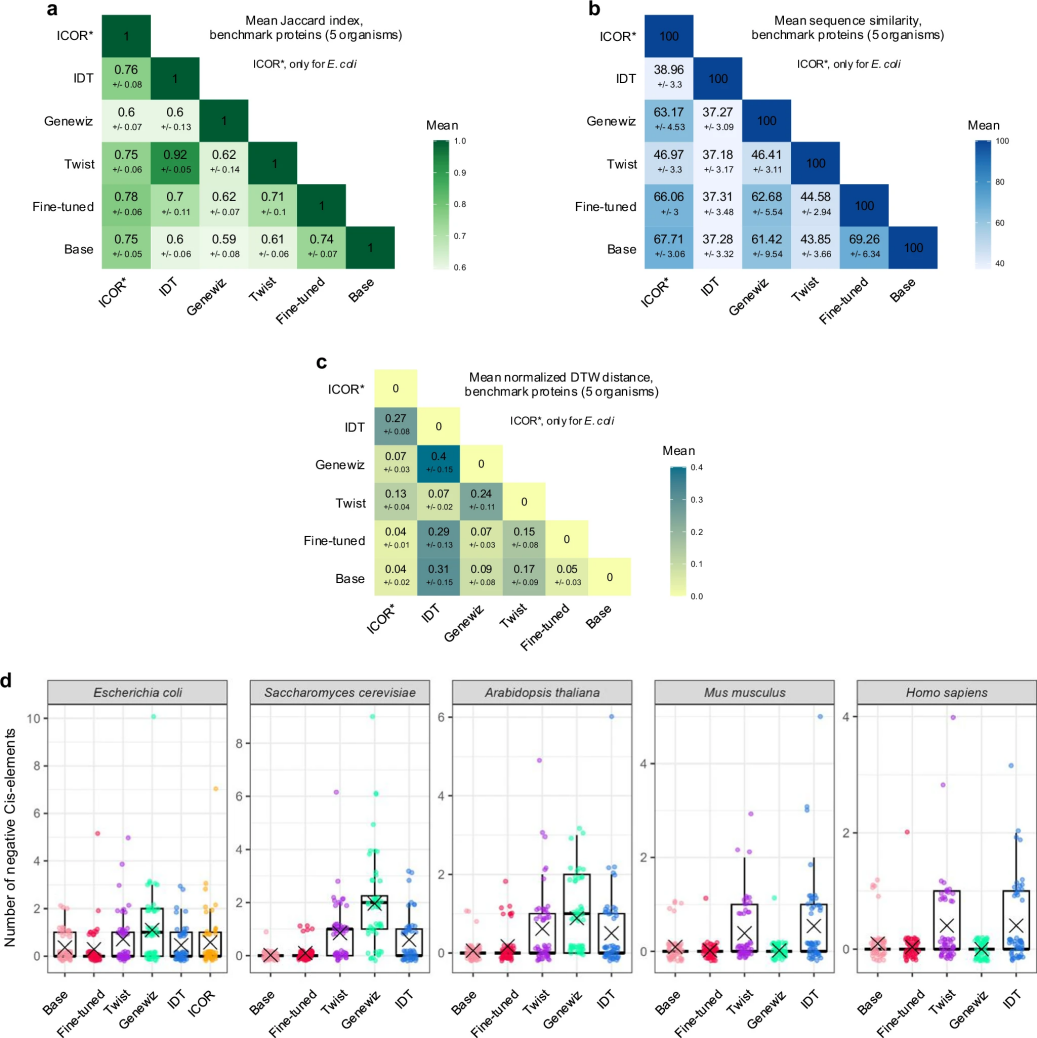
图 4
研究团队对CodonTransformer在异源蛋白表达中的表现进行了全面测试,选取了52个具有实际应用价值的重组蛋白进行分析。如图4所示,他们从多个维度比较了CodonTransformer与市场主流工具(Twist、IDT、Genewiz等)的性能差异。测试结果表明,CodonTransformer生成的DNA序列在密码子分布模式上更接近自然序列,且在所有测试生物体(大肠杆菌、酵母菌、拟南芥、小鼠和人类)中都能最大限度减少负向顺式调控元件(可能干扰基因表达的DNA片段)。特别值得注意的是,尽管CodonTransformer并未专门针对减少这些有害元件进行训练,但它在这方面的表现优于大多数商业工具,这表明该模型成功捕捉到了高效表达基因的内在规律。这一特性使CodonTransformer成为异源蛋白表达和基因设计的理想工具。
讨论
CodonTransformer是一款创新的多物种密码子优化工具,通过深度学习技术解决了基因设计中的关键挑战。这一模型利用来自164个生物体的基因组数据进行训练,能够为广泛的宿主生物体生成最佳DNA序列。如图2-4所示,CodonTransformer具有四大优势:能够学习并应用多物种密码子使用规律;可通过微调适应特定基因集的需求;能生成具有自然密码子分布的序列,避免传统方法中高低频密码子聚集的问题;能最小化有害调控元件的出现,减少对基因表达的干扰。作为开源工具,CodonTransformer不仅提供了用户友好的界面,还可被研究人员进一步定制,特别适用于合成生物学、蛋白质设计和生物制药领域,为解决异源蛋白表达这一生物技术中的关键瓶颈提供了有力支持。
编译|于洲
审稿|王梓旭
参考资料
Fallahpour A, Gureghian V, Filion G J, et al. CodonTransformer: a multispecies codon optimizer using context-aware neural networks[J]. Nature Communications, 2025, 16(1): 3205.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢