近日,Cell Press出版集团数据科学期刊《Patterns》在线发表了中国农业科学院深圳农业基因组研究所(岭南现代农业科学与技术广东省实验室深圳分中心)纪宏超团队的研究论文,题为“Graph-Sequence Enhanced Transformer for Template-Free Prediction of Natural Product Biosynthesis”。研究提出了一种基于图增强Transformer模型用于天然产物生物合成路径规划的方法。

研究背景
天然产物是由生物体合成的有机分子,通常从自然界中的动物、植物或微生物中提取,广泛应用于化学和生物医学等多个领域,尤其在药物发现与开发中发挥着至关重要的作用。然而,天然产物的提取与合成过程仍面临诸多技术挑战,尤其是超过90%的天然产物生物合成途径尚未被充分解析,这极大地限制了其深入研究与实际应用。因此,开展天然产物的逆合成预测具有重要意义。逆合成预测中的机器学习研究通常分为两个方向:单步逆合成预测和多步逆合成路径规划。单步逆合成预测旨在将目标产物分解为一组反应物,而多步逆合成路径规划则涉及使用搜索算法找到目标分子通往从可购买化合物分子的路径。
研究内容
由于产物SMILES序列到反应物SMILES序列的预测过程可以类比为两个字符串之间的翻译任务,目前主流的无模板单步逆反应预测方法普遍将该任务建模为序列到序列的机器翻译问题。然而,此类模型在处理SMILES序列时,往往难以有效利用和挖掘分子的拓扑结构信息,从而限制了对分子反应机制的深层次建模。尽管已有部分研究尝试融合SMILES序列与分子图信息,但多数方法仍将两者割裂对待,难以实现真正意义上的协同建模。
针对上述问题,该研究基于SMILES序列与分子结构之间的一一映射关系,构建了SMILES序列对应的图结构,并在Transformer编码器中引入图神经网络,提出了单步逆合成预测模型GSETransformer。该模型以SMILES序列及其图结构为联合输入:其中,图神经网络处理图结构信息,融合拓扑结构信息改善局部交互;Transformer的多头自注意力机制则用于建模序列中的全局依赖关系。两者结合,使得模型在处理具有拓扑结构的复杂序列数据时,能够提供更强大的特征提取能力。此外,为进一步增强模型性能,该研究引入了根对齐的数据增强策略,进一步提升了模型的鲁棒性与泛化能力。实验结果表明,GSETransformer在生物反应数据集Biochem-Plus和有机反应数据集USPTO-50K上均取得了领先性能,验证了所提方法在逆合成预测任务中的优越性与泛化性。
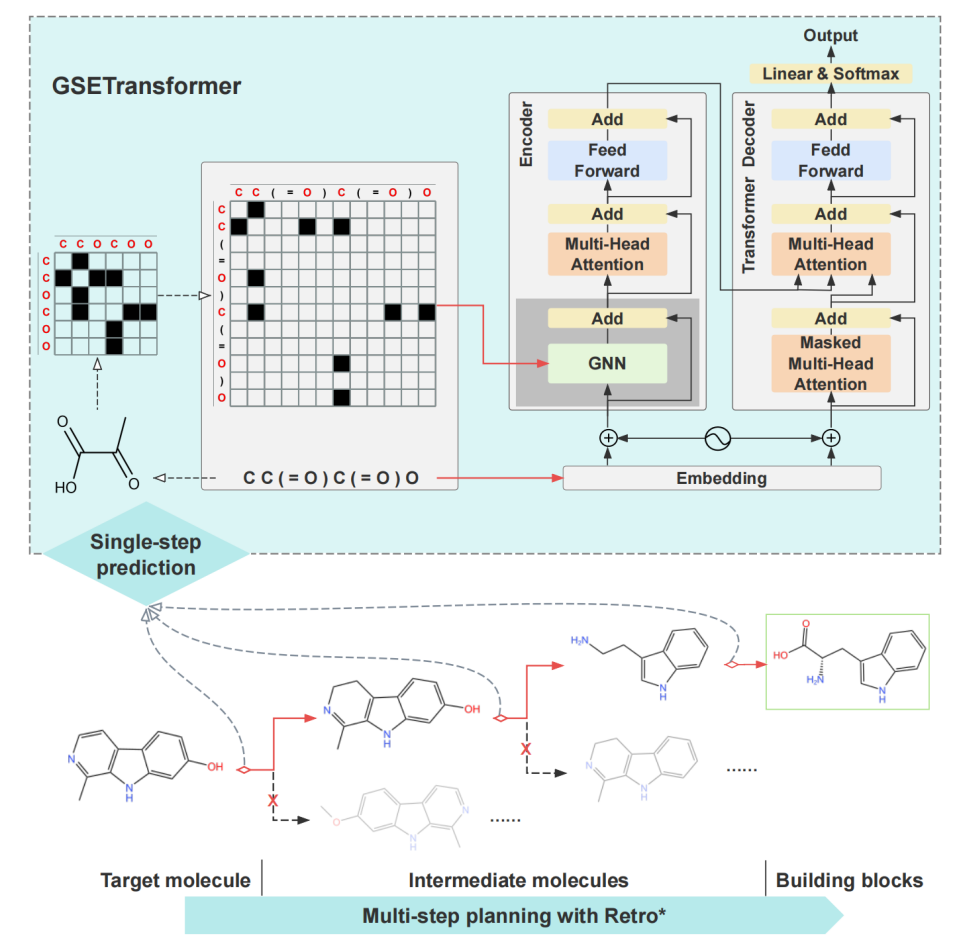
图1:GSETransformer用于天然产物逆合成预测工作流程及架构
为进一步实现天然产物的多步逆合成规划并评估GSETransformer在该任务上的性能,该研究采用Retro*搜索算法,进行了多步逆合成规划的实现与评估。最后,为方便研究人员一站式开展天然产物的逆合成分析及评估,该研究基于QT框架开发了一款集成化、跨平台且用户友好的图形用户界面软件。该软件集成了上述的逆合成预测算法,以及先进的酶预测算法和ADMET预测算法,支持用户在统一的平台上高效完成生物合成路径的探索与药物特性的全面评估,大幅提升天然产物研发的高效性与实用性。
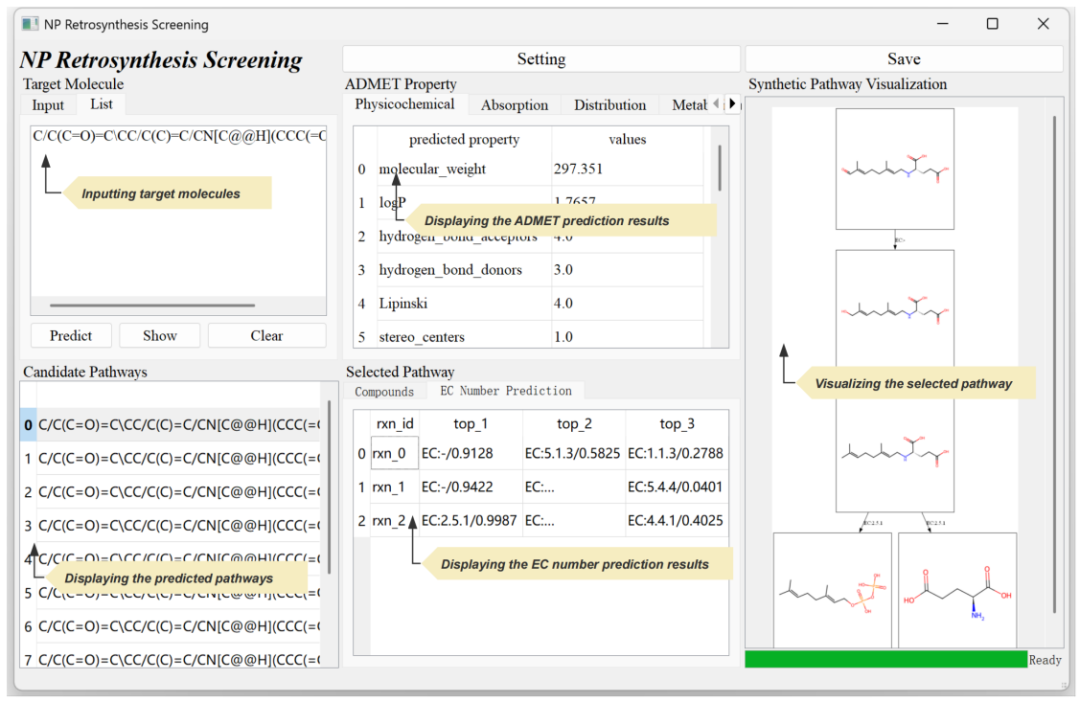
图2:合成路线规划软件GUI界面
本工作由中国农业科学院深圳农业基因组研究所(大鹏湾实验室)与哈尔滨工程大学合作完成,哈尔滨工程大学丛山副教授、哈尔滨工程大学与大鹏湾实验室联合培养硕士研究生张萌为本文共同第一作者,大鹏湾实验室纪宏超研究员为本文章通讯作者。
参考资料:
Cong, Shan, Meng Zhang, Yu Song, Sihao Chang, Jing Tian, Hongji Zeng, and Hongchao Ji. "Graph-sequence enhanced transformer for template-free prediction of natural product biosynthesis." Patterns (2025).
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢