
今天为大家介绍一篇由中南大学董界副教授和曾文彬教授近期在生物材料领域权威期刊《Bioactive Materials》(IF=18.0).上发表题为“Harnessing advanced computational approaches to design novel antimicrobial peptides against intracellular bacterial infections”的研究论文,第一作者为中南大学博士研究生房岩鹏。



背景
胞内菌是指寄生于宿主细胞中生长、繁殖的病原菌。根据对宿主细胞的依赖程度常被分为专性胞内致病菌和兼性胞内致病菌两类(专性胞内菌:衣原体和立克次体等;兼性胞内菌:李斯特菌、布鲁氏菌和致病奈瑟菌等)。胞内菌可通过寄生于宿主细胞实现持续性感染,或利用“特洛伊木马”方式劫持巨噬细胞广泛传播实现多部位感染。目前,临床上对于胞内菌感染,主要依靠长期大剂量使用抗生素。然而,常规抗生素药物难以突破宿主细胞的生物膜屏障,不仅治疗效果大打折扣,还会带来严重的药物不良反应。因此,开发兼具高穿膜效率、强抗菌活性和良好生物相容性的新型抗胞内菌感染药物具有重要意义。
人工智能等先进计算方法为抗菌药物的开发带来变革性发展。例如,基于深度学习模型从ZINC15数据库中筛选出的新型抗生素Halicin,Fuente-Nunez的团队在人工智能的辅助下从微生物的宏基因组数据库中挖掘出具有独特抗菌机制的新型抗菌肽。然而,这些研究主要针对于细胞外细菌,对于具有更加复杂生理环境的胞内菌并不适用。在这项工作中,作者围绕胞内菌药物开发面临的多维挑战:宿主细胞膜的高效渗透;维持宿主细胞内活性和稳定性;致病菌和宿主细胞靶标之间的特异性识别。基于人工智能模型在药物活性预测方面的显著优势以及分子动力学模拟提供的药物作用机制微观视野。将人工智能、分子动力学模拟和实验验证相结合用于抗胞内菌药物的高效开发。
为了确保候选药物的穿膜效率,研究者选取Gautam团队开发的CPPsite 2.0数据库作为研究对象,该数据库包含1850条经实验验证的细胞穿膜肽,这为抗胞内菌药物设计提供了重要基础。随后基于机器学习算法构建了活性预测模型AMP_model和Hemolysis_model,前者确保靶肽的抗菌活性,而后者则侧重于生物相容性,将二者联用以从目标数据库中快速筛选出兼具穿膜效率、抗菌活性和安全性的候选肽。在此基础上,基于跨尺度分子动力学模拟研究构建肽-膜相互作用综合评价系统。从全原子到粗粒化,在不同维度对候选肽与磷脂膜(细菌细胞膜POPE/POPG;哺乳动物细胞膜POPC)之间的相互作用进行分析,从而提高筛选结果的精确性和成功率。最后,通过体外和体内实验对靶肽Crot-1的生物活性进行全面验证(图1)。结果表明Crot-1有效地消除了细胞内MRSA,并对宿主细胞没有明显的细胞毒性,突出了其临床应用的潜力。这项工作为开发新型抗胞内菌药物提供了一种新的思路。

结果

基于人工智能模型高效筛选候选肽


针对抗胞内菌药物的三个关键活性:穿膜、抗菌和生物相容性。研究者首选选取CPPsite 2.0数据库作为筛选文库,确保穿膜特性。随后,从抗菌肽数据库APD3、DBAASP和DRAMP中收集活性数据,并且结合7种机器学习算法和5种分子表征方法构建了一系列模型,经过全面的性能评价选取用于抗菌活性预测和溶血效应预测的分类模型AMP_model和Hemolysis_model。前者采用XGBoost算法和PEP描述符(通过PyBioMed计算的肽描述符)组合构建,在测试集上的SE为0.935,ACC为0.953,AUC为0.990。Hemolysis_model模型由Catboost算法和PEP描述符(组合构建,在测试集上的SE为0.672,ACC为0.721,AUC为0.763。将两个模型依次对CPPsite 2.0数据库中的序列进行筛选。通过以下规则确定候选肽序列:a)AMP_model预测为抗菌肽概率大于0.9;b)Hemolysis_model预测结果为非溶血肽;c)肽序列长度小于10个氨基酸。经过模型预测及理化性质限制,研究者快速锁定6个兼具穿膜特性、抗菌活性和生物相容性的候选肽(图2)。
跨尺度分子动力学模拟解析肽-膜相互作用

通过人工智能模型初步得到6条候选肽序列之后,研究者借助分子动力学模拟对候选肽和磷脂膜之间的相互作用进行系统性研究。通过比较肽在细菌细胞膜体系(POPE/POPG)和哺乳动物细胞膜体系(POPC)中的行为差异分析其靶向性和作用机制。具体工作包含三个部分:
a) 牵引分子动力学模拟(500 ns):通过计算候选肽跨越POPC膜的PMF来评估其对宿主细胞的渗透效率。
b) 全原子分子动力学模拟(100 ns):通过分析单个肽分子在两种膜系统中的构象稳定性、能量和氢键相互作用来评估肽与膜的亲和性。
c) 粗粒化分子动力学模拟(2 μs):在更长的时间尺度上探索Crot-1对磷脂膜的扰动作用。
为了更加精确量化6条候选肽的穿膜效率,研究者借助伞形采样方法对候选肽穿过宿主细胞膜过程的自由能分布进行计算,结果表明Crot-1具有最低的能垒24.3 kcal/mol。靶向性是确保药物高效清除胞内菌而无细胞毒性的关键因素。全原子分子动力学模拟被用于从原子层面探索肽-膜互作,在氢键、能量、溶剂可及化表面积以及构象稳定性方面分析结果表明,相较于哺乳动物细胞膜,所有候选肽与细菌细胞膜之间具有更稳定的相互作用。其中Crot-1的表现最为出色。随后,研究者开展粗粒化分子动力学模拟来探索不同浓度的Crot-1对于膜的扰动,这种方法降低了系统的自由度,从而扩展了模拟的时空尺度。与全原子分子动力学模拟结果一致,Crot-1对于POPC膜无明显扰动,而在POPE/POPG膜体系中,Crot-1分子稳定嵌在磷脂膜的内部,并聚集形成孔道,导致溶剂和离子内流。研究者从Crot-1的质量密度分布、磷脂膜有序度参数以及膜曲率方面对肽-膜相互作用进行量化分析(图3)。这些跨尺度的模拟结果表明6条候选肽都倾向于与细菌细胞膜结合而对哺乳动物细胞膜无明显相互作用,在抗胞内菌感染方面具有巨大的应用潜力。
Crot-1生物活性验证


接着,研究者合成制备Crot-1,并开展体内外实验对其活性进行验证。依据团队前期的研究基础对Crot-1进行荧光标记对其穿膜过程进行可视化。随后,将RAW 264.7细胞和MRSA进行共孵育以构建胞内菌模型,平板涂布和死活染色实验结果表明,32 μM浓度的Crot-1可以有效清除寄生于巨噬细胞内的MRSA,而阳性对照组则无明显的抑菌效果。为了验证Crot-1的生物相容性,研究者开展溶血实验和MTT实验。MTT分析显示,经过128 μm处理后RAW 264.7细胞存活率仍高于86%,溶血率则为16.23%。这些结果与人工智能模型预测和分子动力学模拟分析的结果高度一致。
最后,研究者基于MRSA诱导的皮下感染模型和急性腹腔炎模型对Crot-1的抗胞内菌效果进行在体评估。接受Cro-1治疗的小鼠感染部位成功愈合,感染部位无明显细胞损伤和炎症浸润。平板涂布结果表明,Crot-1治疗组小鼠感染部位的细菌载荷为PBS组的7.18%。在急性腹膜炎模型中。与万古霉素和PBS组相比,Crot-1的治疗显着降低了腹膜巨噬细胞内的细胞内MRSA负载量。主要器官病理学分析和体重统计结果表明Crot-1具有良好的生物相容性(图4)。这些体内和体外实验结果表明,Crot-1可以有效地消除细胞内细菌,同时对宿主细胞无明显毒性。

结论
本研究首次将计算机辅助药物设计应用于抗胞内菌药物开发,提出整合人工智能预测、分子动力学模拟与实验验证的三位一体开发策略。针对胞内菌感染复杂生理环境,采用多级人工智能预测系统平衡候选药物的活性和毒性。基于跨尺度分子动力学模拟构建肽-膜相互作用综合评价体系,从原子层面解析候选分子的靶向性与作用机制。随后,制定系统的实验方案对于目标分子的生物活性进行全面验证。该研究不仅从CPPsite 2.0数据库成功发掘出候选药物Crot-1,更构建了一套可扩展的计算驱动框架。其核心价值在于: 宏观性质预测(AI)与微观机制解析(分子模拟)互补,突破传统试错模式;可适配不同靶点与疾病场景,为新型抗菌药物开发提供新的范式。

图文赏析

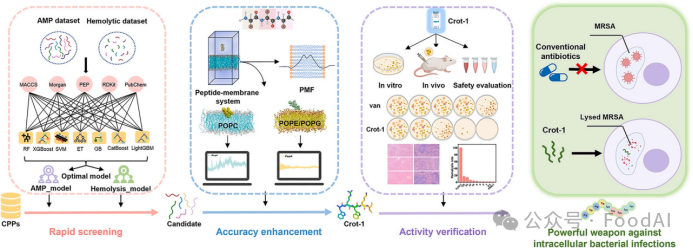
图1. 新型抗胞内菌药物设计思路
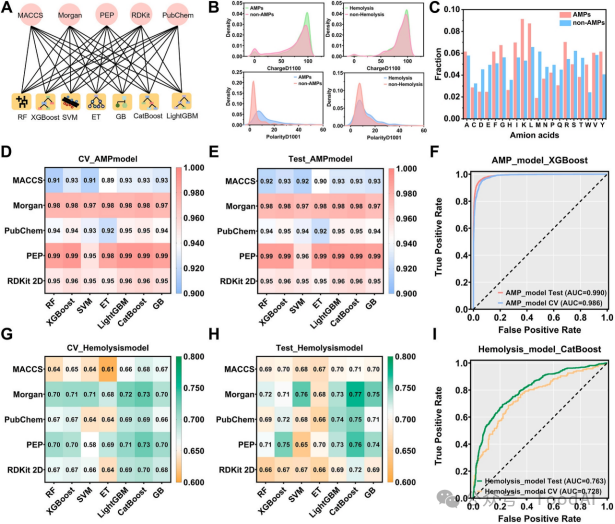
图2. 最优筛选模型的构建。(A)模型构建的分子表征方法和算法。(B)训练数据集的性质分布。(C)AMP_model筛选结果的氨基酸组成分布。(D-F)AMP_model的性能评价(G-I)Hemolysis_model模型的性能评价。
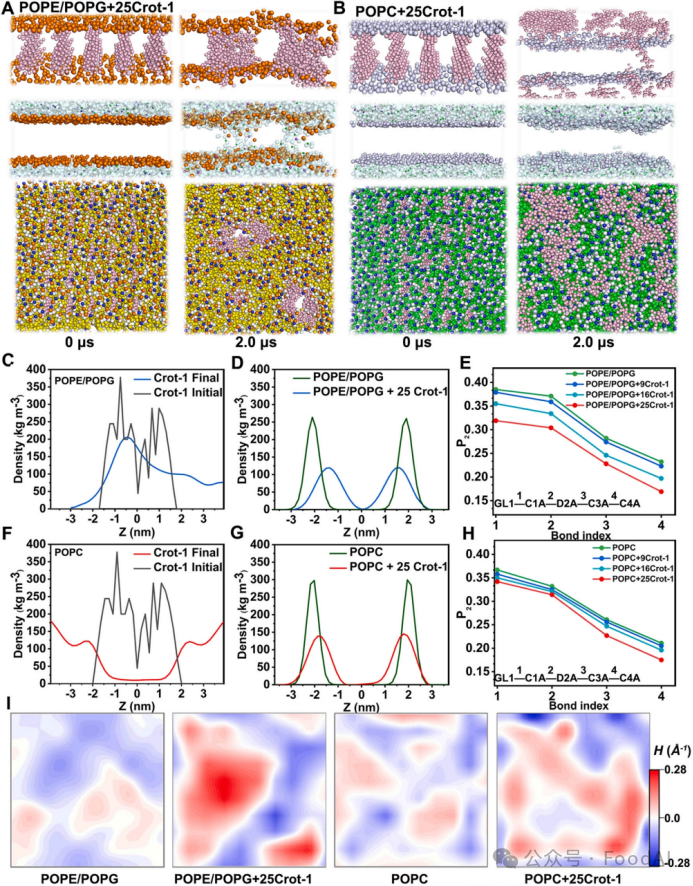
图3. Crot-1与磷脂膜的粗粒化分子动力学模拟。(A)25个Crot-1分子与POPE/POPG分子模拟闪照。(B)25个Crot-1分子与POPC分子模拟闪照。(C,F)粗粒化模拟起始Crot-1的质量密度分布。(D,G)粗粒化模拟起始磷原子的质量密度分布。(E,H)不同模拟系统中磷脂分子的油酰酰基链有序参数。(I)不同模拟系统中磷脂膜的平均曲率。
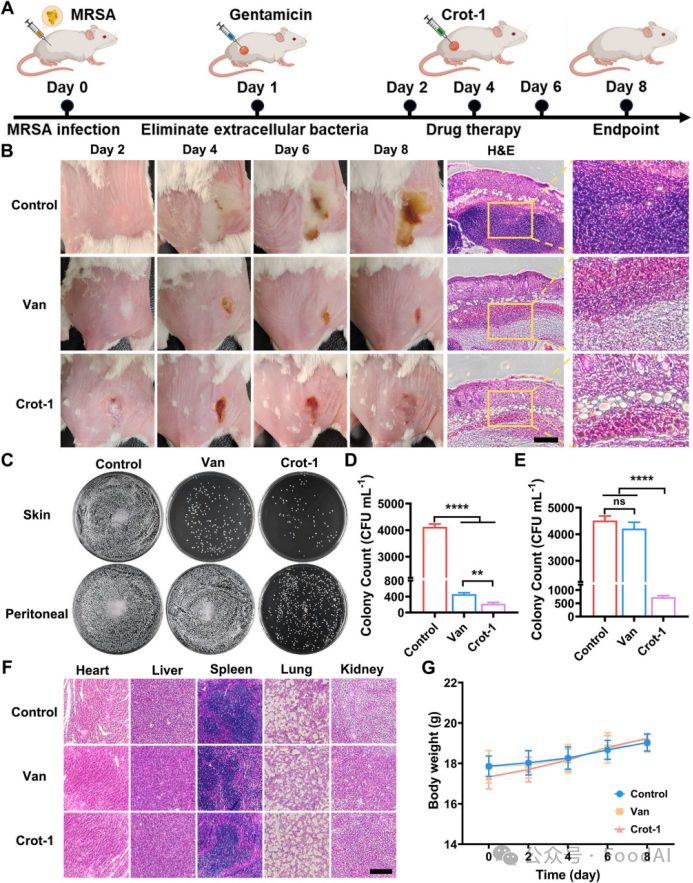
图4. Crot-1抗菌活性和生物相容性的体内评估。(A)Crot-1清除小鼠皮下感染模型中细胞内MRSA的示意图。(B)感染部位记录。(C)感染部位分离菌落平板涂布结果。(D,E)感染部位菌落负载分析。(F)小鼠主要器官的H&E染色分析。(G)小鼠体重变化统计结果。

参考资料

Fang, Y., Fan, D., Feng, B. et al. Harnessing advanced computational approaches to design novel antimicrobial peptides against intracellular bacterial infections. Bioact. Mater, 50, 510-524 (2025).
https://doi.org/10.1016/j.bioactmat.2025.04.016
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢