DRUGAI
粗粒化建模已成为实验测量的重要补充工具,使研究人员能够探索超越全原子方法的时空尺度。GōMartini 模型结合了结构驱动与物理驱动的粗粒化策略,在保持计算效率的同时,准确刻画蛋白质动力学,并支持在多种生物环境中的研究。本文提出了增强版 GōMartini 模型,融合了虚拟位点实现的 Gō 模型与 Martini 3。该实现自新版 Martini 重新参数化以来已被广泛验证。研究人员通过多个案例展示了模型的适用性,包括蛋白-膜结合、蛋白-配体相互作用以及原子力显微(AFM)力谱分析。此外,该模型还能修正 Martini 蛋白模型中近期发现的问题。最后,作者探讨了 Martini 3 与 Gō 模型结合的优势、局限与未来发展方向。

理解蛋白质如何选择性、高效地折叠并发挥功能,需依赖其结构与动态行为的深入认知。尽管实验技术取得了巨大进展,研究人员仍借助分子建模作为重要的补充手段。全原子(AA)分子动力学模拟已广泛应用于蛋白质动力学与简单系统的折叠研究,但受限于高计算成本,通常仅能捕捉微秒级别的局部过程,难以涵盖蛋白质结构域的大尺度运动、诱导契合机制,或膜蛋白系统中的慢速动力学。此外,对于需要大量采样的非平衡拉伸、无序蛋白的构象集建模等任务,AA方法也面临挑战。
粗粒化(CG)模型作为替代方案,通过简化自由度和能量景观,大幅提升了模拟效率,能够覆盖更广泛的时空尺度。结构驱动的Gō模型适用于研究折叠、展开与机械响应等构象变化过程,但常忽略环境影响;而物理驱动的Martini力场则能模拟蛋白质与膜、配体及其他生物分子的相互作用,但在保持蛋白质稳定折叠方面存在局限,常依赖弹性网络(EN)维持结构,可能引入蛋白“黏性”问题。
为兼顾动力学精度与环境交互,研究人员提出将结构驱动与物理驱动模型结合,即GōMartini策略。GōMartini可再现蛋白柔性特征,并在蛋白力学研究中展现出潜力,但仍难以处理远程构象变化,且部分继承了Martini 2的局限性。
本文提出增强版GōMartini模型的虚拟位点实现,并结合最新的Martini 3力场。研究人员展示了该模型在多个场景中的应用,包括蛋白-膜结合、蛋白-配体相互作用、变构通路解析及单分子力谱模拟等。同时,模型引入环境偏倚修正机制,解决Martini在无序蛋白维度预测不足及某些小肽疏水性低估的问题。文章还探讨了GōMartini 3的优势、局限与未来发展方向,展示其在蛋白质建模中的广阔前景。
研究结果
Martini 3 蛋白模型
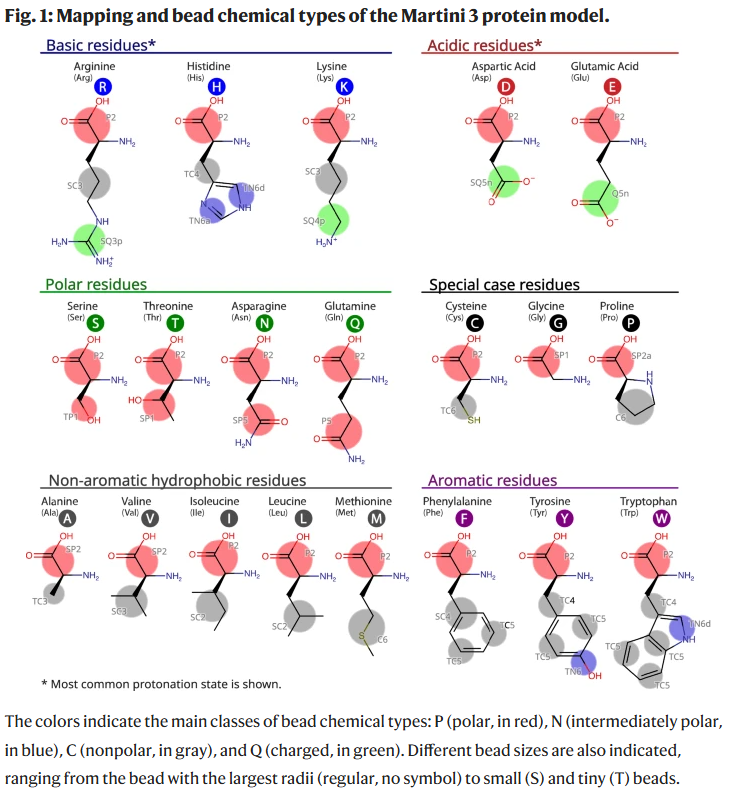
Martini 3 蛋白模型是 Martini 2 的自然演进,尽管其尚未完全采用新版参数规则,因此仍属于原型模型。模型以一个主链粒子(BB)表示每个氨基酸主链,位置对应原子质量中心,并根据残基类型附加 1–5 个侧链粒子(SC)。主链之间通过键长、键角、二面角等定义构象限制,这些参数来自于约 2000 个 PDB 蛋白的统计分析。Martini 3 模型中所有主链默认为 P2 极性粒子,少数残基(如 GLY、PRO)采用不同粒子类型以更准确表达结构特性。
侧链部分完全重参数化,基于 backbone-less 模拟与实验数据(如油水分配系数)进行调优。此外,采用了 dihedral 校正项(scFix)以避免侧链过度柔性。由于仅依赖弹性网络(EN)维持三级结构,Martini 3 蛋白仍需要与 Gō 模型等结构偏倚方法联合使用。当前 EN 采用 0.85 nm 截断与 700 kJ/mol·nm² 力常数,但过强可能导致蛋白“黏性”问题,影响聚集行为。
Martini 3 已在多种体系中验证,包括可溶蛋白、跨膜肽、蛋白-小分子与脂质相互作用等,显示出较强的通用性。
GōMartini 模型的增强实现
研究人员引入了基于虚拟位点的 GōMartini 实现,用以更灵活地构建结构偏倚接触图。虚拟位点不参与体系其他相互作用,仅用于 Gō 力场的 Lennard-Jones 接触定义。其优势在于允许采用非键合截断、增强并行化能力,并改善蛋白质构象柔性控制。增强版还排除了接触对之间 Martini LJ 力,以避免接触位置偏移与结构扭曲。
接触图构建引入了距离筛选(0.3–1.1 nm)和图距离限制(k ≥ 4),以避免过短或冗余接触。整体而言,增强版 GōMartini 模型在保持蛋白折叠结构的同时,显著提升了模拟效率与动态表现。
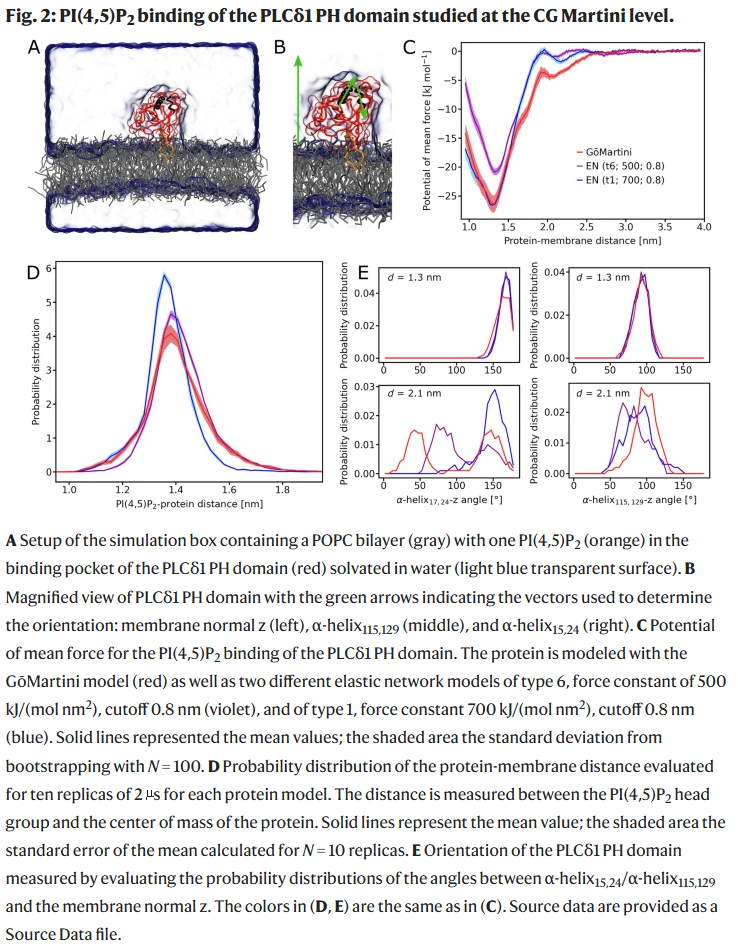
案例研究一:PH 结构域与膜结合
研究人员通过 GōMartini 与两种 EN 模型对 PLCδ1 PH 结构域与 PI(4,5)P₂ 脂质结合进行了比较。三种模型均能再现高亲和力结合,GōMartini 模型在松散结合状态下展现出更高柔性与多构象取向,揭示其更能反映实际膜结合动态。
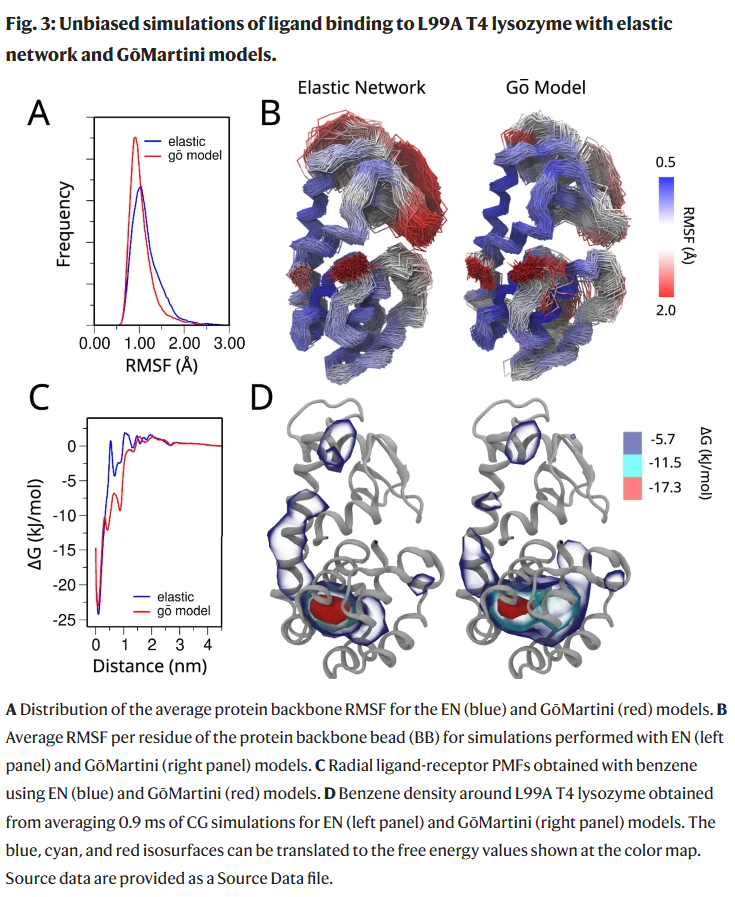
案例研究二:T4 溶菌酶配体结合
在 T4 溶菌酶 L99A 突变体系统中,GōMartini 模型成功捕捉到苯分子结合路径中的中间稳态,与 EN 模型相比展现出更丰富的自由能谷。这表明 GōMartini 更能揭示诱导契合机制下的蛋白微结构变化。
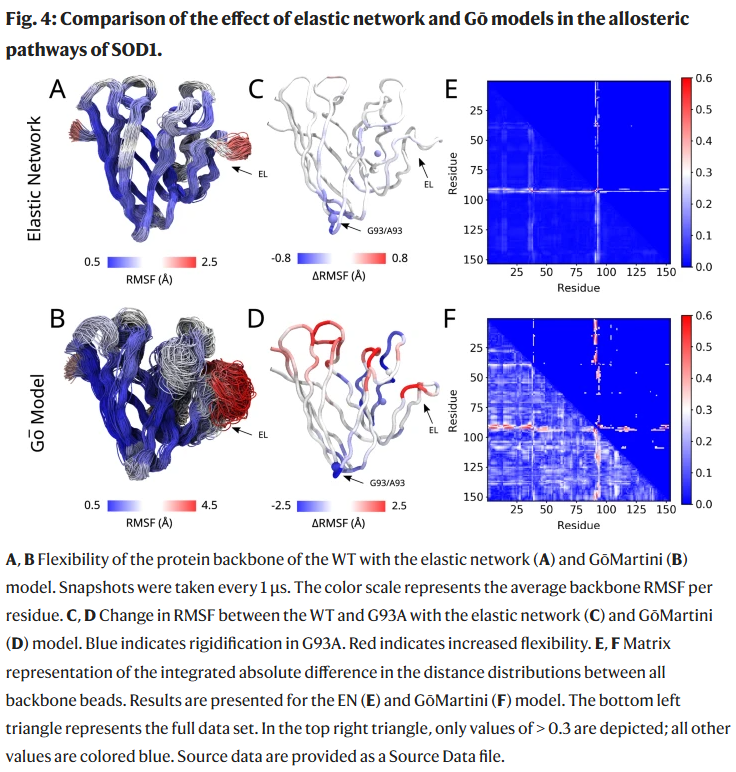
案例研究三:SOD1 的变构通路
在研究 G93A 突变影响 Zn²⁺ 解离的过程中,GōMartini 模型识别出突变位点与电荷环之间的长程耦合动态通路,而 EN 模型未能揭示此差异,显示出 GōMartini 更强的变构通路捕捉能力。
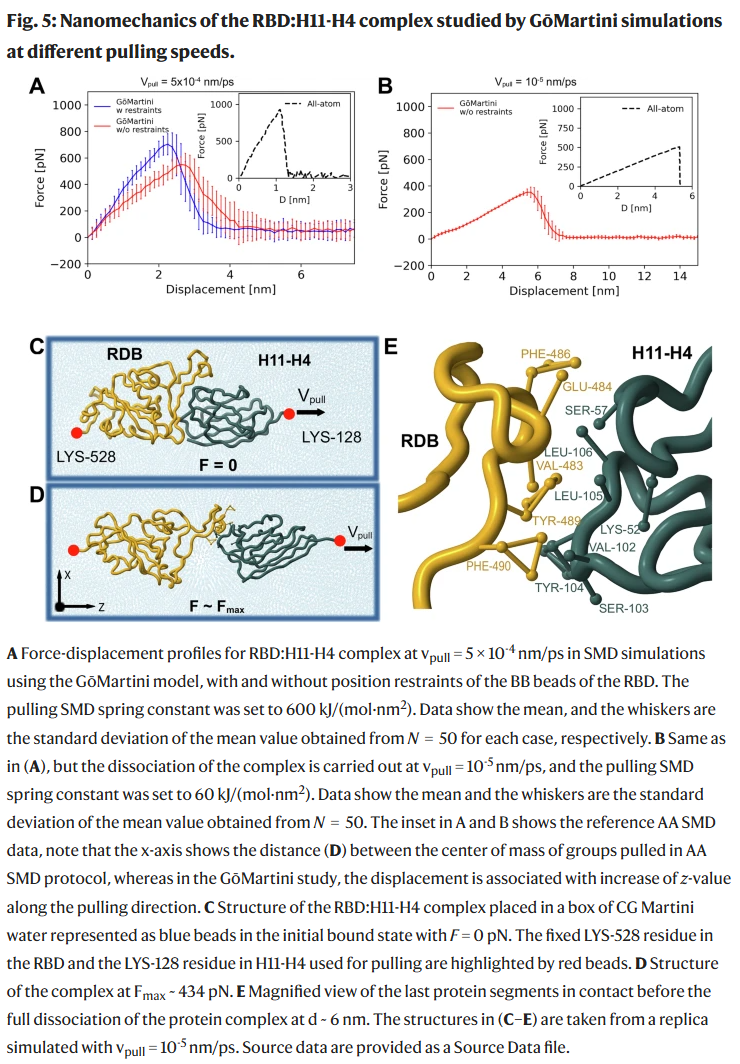
案例研究四:抗原-纳米抗体复合物的力学稳定性
在无位置限制条件下进行单分子力谱(SMFS)模拟,GōMartini 3 成功再现实验测得的断裂力(~925 pN)并避免了 EN 模型中常见的刚性过强问题。此外,GōMartini 模拟揭示了纳米抗体结合界面关键残基的机械响应路径,体现出该模型在蛋白纳米力学研究中的应用前景。
整理 | WJM
参考资料
Souza, P.C.T., Borges-Araújo, L., Brasnett, C. et al. GōMartini 3: From large conformational changes in proteins to environmental bias corrections. Nat Commun 16, 4051 (2025).
https://doi.org/10.1038/s41467-025-58719-0
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢