DRUGAI
单细胞测序以单细胞分辨率实现转录组水平分析,前所未有地揭示了细胞异质性。然而,现有分析方法仍受噪声、批次效应和数据稀疏性的限制,亟需统一模型来表征细胞状态。为解决这一问题,研究人员开始基于大规模数据训练单细胞基础模型,但现有模型受限于训练数据量和参数规模。在本研究中,研究人员整合了1亿个人类细胞的多样性数据,构建了包含8亿参数的单细胞基础模型CellFM。该模型在MindSpore上采用改进的RetNet架构进行训练,兼顾效率与性能。多项实验证明,CellFM在细胞注释、扰动预测、基因功能预测和基因关系捕捉等任务中均优于现有模型。

单细胞RNA测序(scRNA-seq)技术革新了分子生物学,使研究人员能够以前所未有的规模和精度获取细胞水平的转录组信息。随着技术发展,海量单细胞数据迅速积累,但这些数据常伴随噪声、稀疏性和批次效应,给分析带来挑战。尽管已有多种针对单细胞的分析工具,研究人员发现这些方法在面对新数据或大规模数据时往往性能有限,且难以充分利用大型图谱数据中的丰富信息。
为应对这一挑战,研究人员开始开发单细胞基础模型,借鉴大语言模型在自然语言处理中的成功经验。部分工作尝试通过少量单细胞数据对预训练语言模型进行微调,如将细胞表达数据转化为基因排序序列输入GPT模型,或基于基因注释构建嵌入向量。这类方法虽取得一定进展,但仍未充分利用大规模单细胞表达数据,亟需更系统的策略。
近年来,从零开始构建单细胞基础模型的研究不断推进,主要包括三类策略:基因排序、表达值分类、表达值投影。排序方法如iSEEEK和tGPT基于基因表达水平对基因排序,训练模型学习排序模式;scBert和scGPT等分类方法将表达值离散化为类别,利用分类机制进行建模;表达值投影方法则直接预测原始表达值,如scFoundation和GeneCompass,保留了数据的连续性和完整性。另有模型尝试整合蛋白语言模型、元数据描述等信息,以提升泛化能力。
尽管已有诸多尝试,但尚无模型专门在1亿个人类细胞数据上进行全面训练。由于数据收集和格式不统一等问题,以往模型训练数据多在5000万细胞以下,参数规模也普遍低于1亿,限制了模型能力的提升。
为此,研究人员从多个公共数据库整合并清洗了不同平台生成的单细胞数据,最终构建了一个包含约1亿人类细胞的大规模数据集,数据量为现有最大人类模型的两倍。基于此,研究人员提出了CellFM,一个具有8亿参数的单细胞基础模型,参数规模是现有模型的8倍。CellFM采用改进的线性Transformer架构ERetNet,兼顾训练效率与性能。作为一种基于表达值投影的模型,CellFM通过掩码重建方式学习基因表达模式。
CellFM在华为MindSpore平台上开发,并部署于配备Ascend910芯片的Altas800服务器上进行训练。大量实验证明,CellFM在细胞注释、扰动预测、基因功能推断等多项任务中表现优异,显著优于现有单细胞基础模型。
研究结果
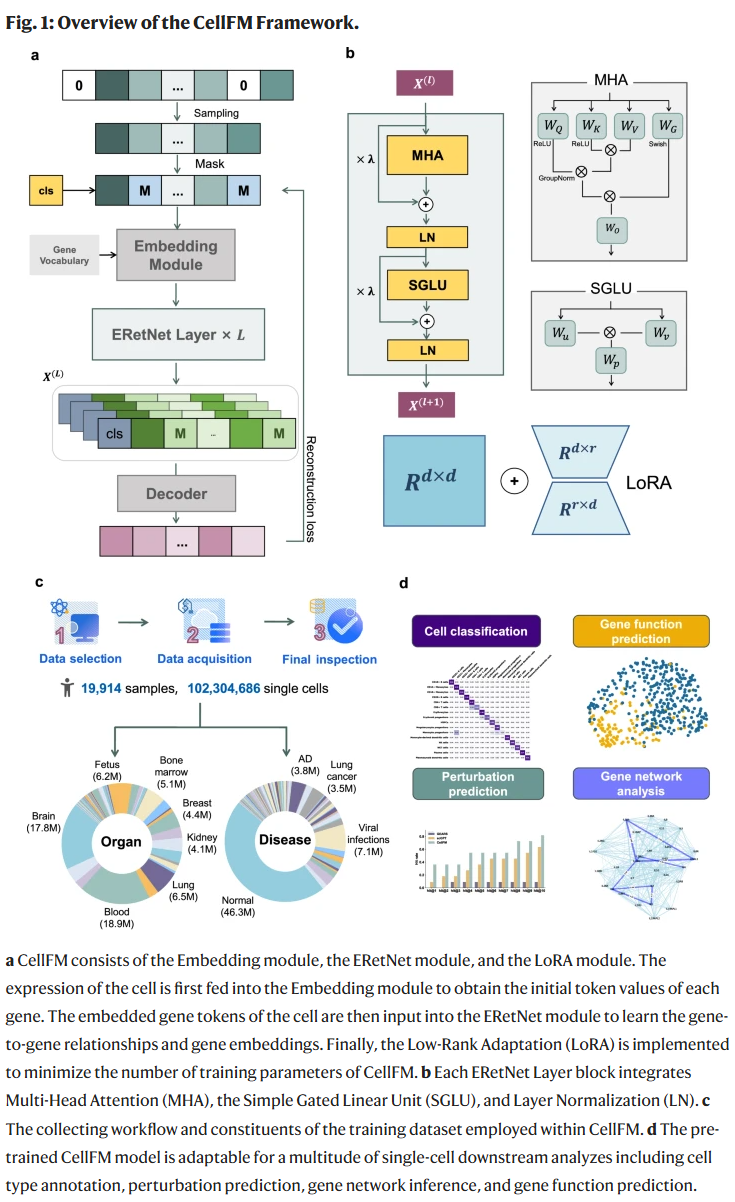
CellFM概述
单细胞测序技术能够以细胞分辨率揭示细胞多样性和功能。研究人员从多个公共数据库中收集并标准化处理了大规模人类单细胞转录组数据,涵盖近2万个样本,共计超1亿个细胞。这些数据覆盖多个器官和测序平台,其中超过7000万个细胞带有注释信息。基于该数据集,研究人员构建了CellFM,一个具有8亿参数的高效基础模型。CellFM采用改进的RetNet结构ERetNet,并结合LoRA模块提升微调效率。该模型可用于基因功能预测、细胞类型注释、扰动响应预测和基因网络分析等任务。
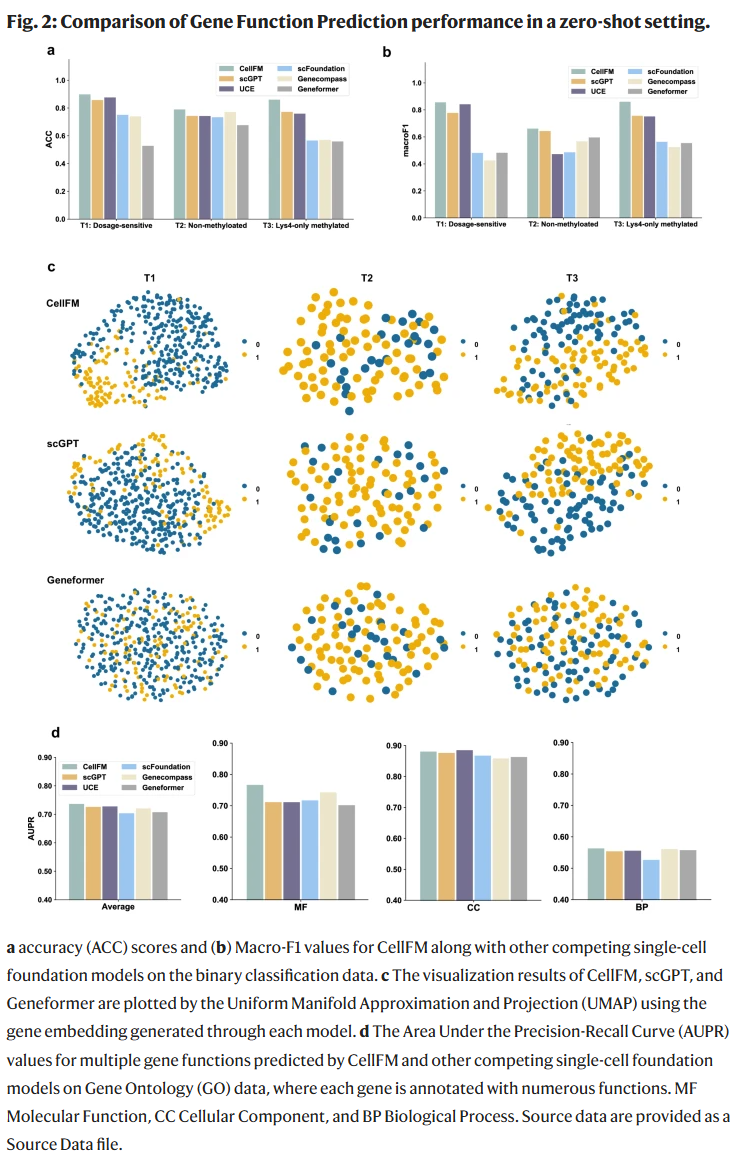
提升基因功能预测精度
研究人员评估了CellFM在三类基因功能的零样本预测任务中的表现。与现有模型相比,CellFM在准确率和Macro-F1指标上均取得最优结果。在基于GO的多分类预测中,CellFM同样在三大类功能(BP、CC、MF)中展现出更高的平均AUPR值,优于GeneCompass和UCE等模型。
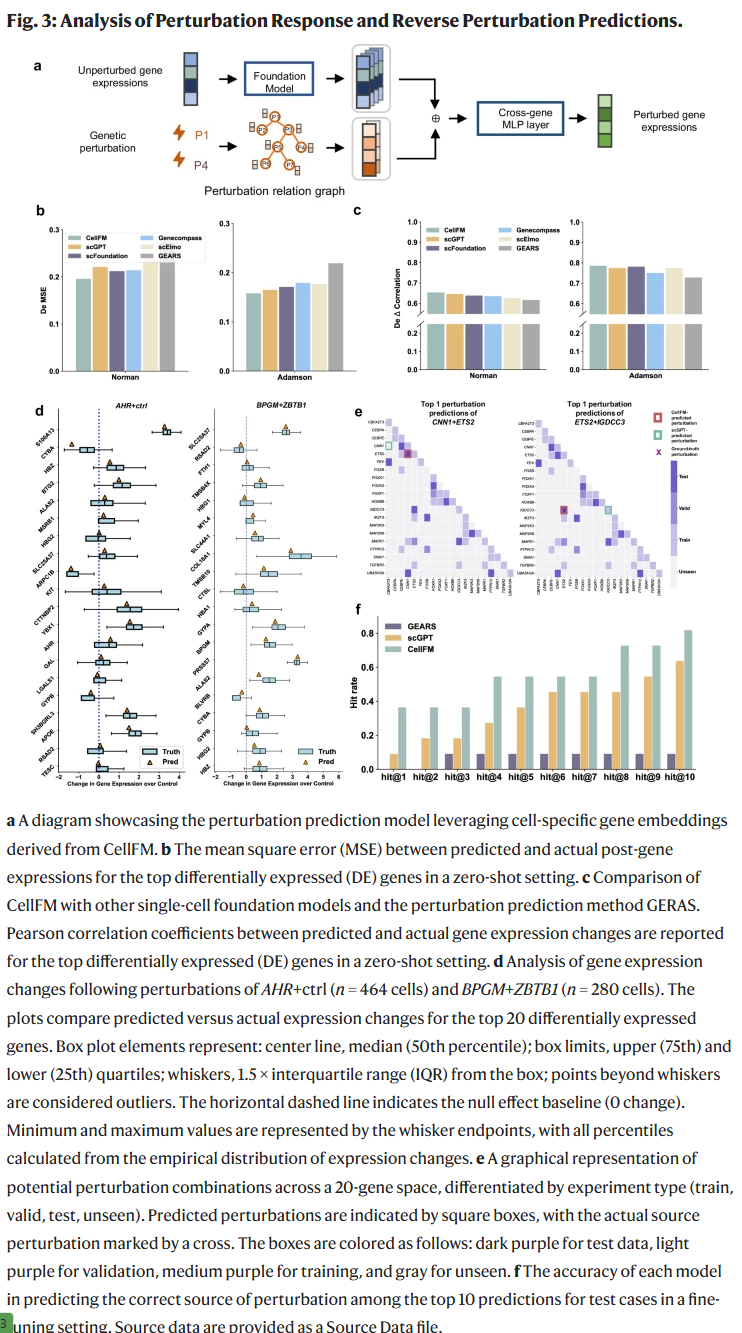
实现扰动响应预测
CellFM在两个Perturb-seq数据集上预测基因扰动效果表现优越。结合经典模型GEARS,CellFM的预测精度在PCC和MSE指标上分别提升1%和1.45%,且优于GEARS本身。在模拟未知扰动组合中,CellFM成功区分不同扰动的细胞反应,并准确识别主导基因。此外,CellFM在与CellOT结合用于药物扰动预测时,同样在多个药物条件下取得更优结果。
反向扰动预测能力强
在模拟逆向扰动预测中,CellFM准确预测了可逆转病态的关键基因组合。在多次随机分割实验中,CellFM在Top-K命中率上显著优于scGPT和GEARS,表现出更强的稳定性和泛化能力。
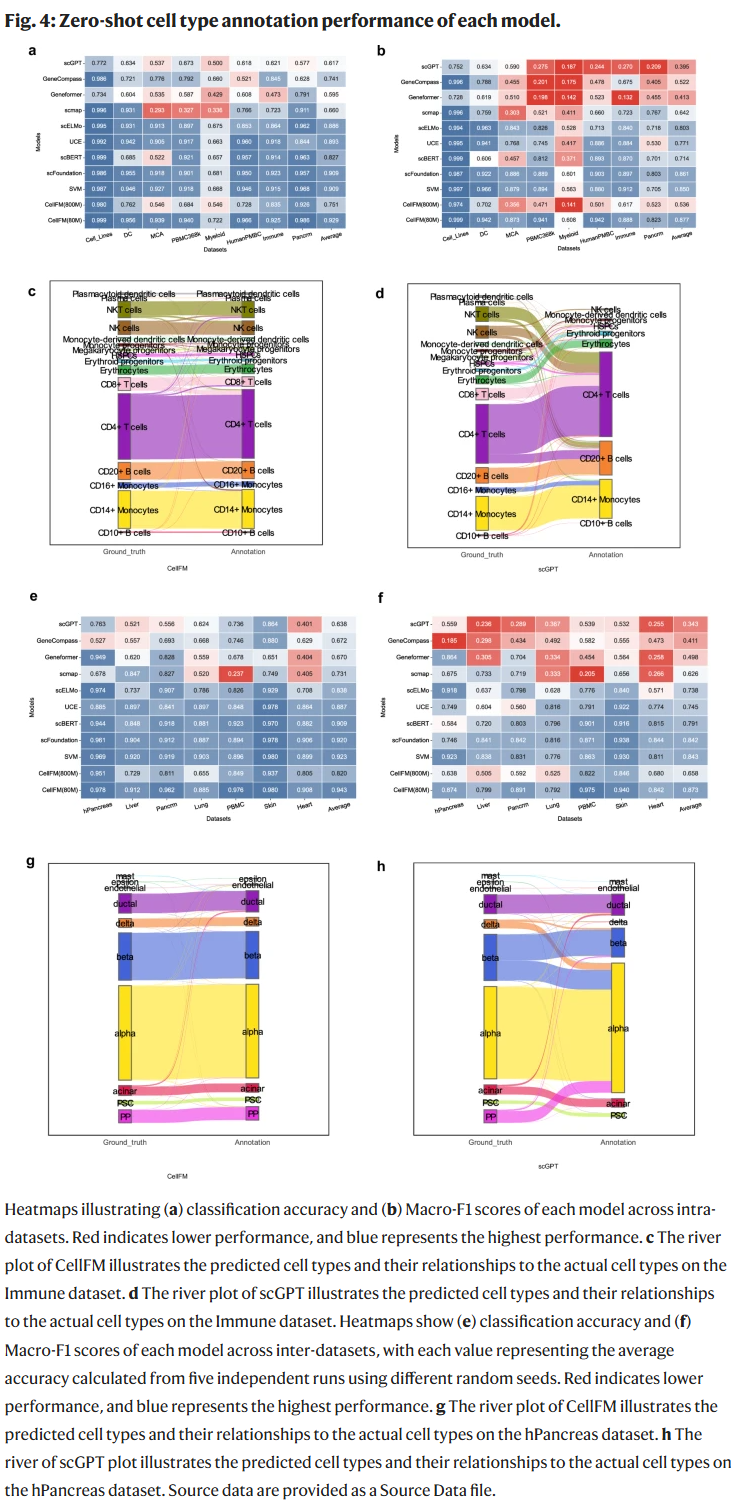
细胞类型注释表现优异
在多个数据集上的细胞注释任务中,CellFM表现出色,无论是零样本预测还是微调后,均超过现有模型。在跨批次和亚型识别(如耗竭/激活CD8+ T细胞)中,CellFM同样优于UCE等模型,平均准确率高出2.3%。在使用LoRA模块微调时,CellFM在不影响性能的同时大幅减少训练时间。
揭示lncRNA功能和模型结构效率
CellFM能够通过注意力机制识别与细胞类型相关的lncRNA,如HOTAIRM1在CD14+单核细胞中的关键作用。同时,在不同正则化策略(如scTransform)和网络结构的消融实验中,CellFM保持了稳定的高性能,并在计算效率上优于经典Transformer结构。
整合数据集与批次效应修正
研究人员在多个跨平台数据集中评估了CellFM对批次效应的处理能力。结果显示,CellFM在AvgBio得分上优于scGPT和UCE等模型,展现出出色的数据整合能力。
解析基因调控关系
通过注意力图谱和嵌入向量,CellFM成功捕捉了多个关键免疫相关基因(如IL-2, IL-3, IL-4)之间的调控关系,并能区分细胞特异性网络。相比共表达网络分析,CellFM识别出更多富集通路,尤其在免疫通路中表现更好。
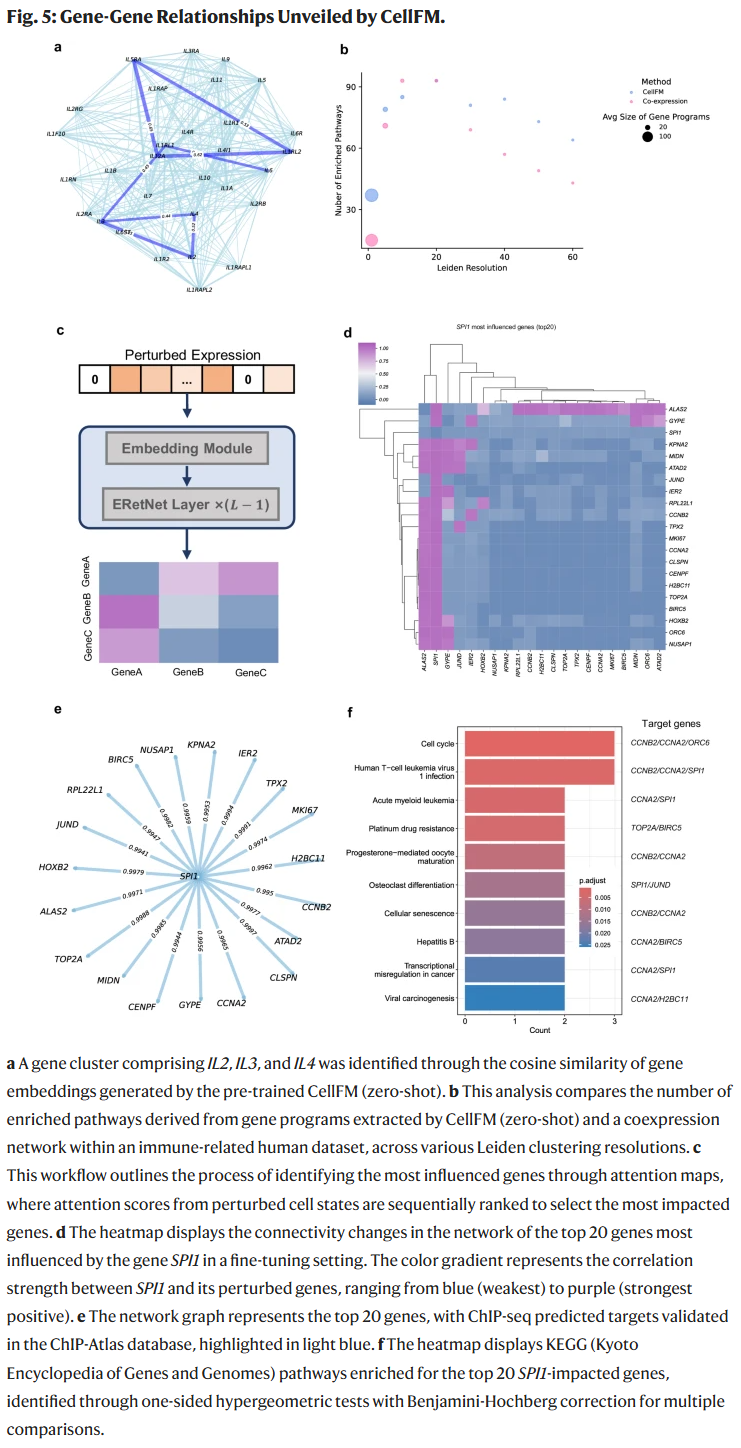
识别扰动影响基因与通路
利用注意力机制,CellFM可识别在扰动实验中受影响最显著的基因,其结果与ChIP-Atlas等数据库高度一致。例如在SPI1和JUN扰动中,CellFM捕捉到多个关键转录因子及通路(如HTLV-1感染、急性髓系白血病、焦点黏附等),提供了精准的机制解析能力。
结论
为高效解析单细胞数据并充分利用大型图谱数据中蕴含的生物学信息,研究人员提出了CellFM这一先进的基础模型。该模型基于精心整理的约1亿个真人类单细胞转录组数据进行预训练,参数规模达8亿,是当前同类单物种模型参数量的8倍。为了提升训练效率,CellFM采用了改进的RetNet架构ERetNet,具备更强的并行处理能力与高效推理能力。同时,模型中引入了低秩自适应模块(LoRA),在微调阶段显著减少可训练参数数量,在保持泛化能力的同时提高任务适应性。
在多个下游任务中,CellFM表现出色,包括细胞类型注释、扰动响应预测、基因网络解析和基因功能预测等。模型基于华为MindSpore框架开发,并在四台搭载Ascend910芯片的Atlas800服务器上完成训练。相比其他单细胞基础模型,CellFM不仅具备更大的训练数据量(约1亿细胞),其参数规模也远超GeneCompass(约8倍)和UCE(约1.23倍),并在多项评估任务中持续优于这些模型。
在模型架构方面,CellFM未采用Transformer,而是构建于RetNet变体之上,通过整合SGLU模块提升训练效率,并引入LoRA机制优化相似数据集上的微调性能。这种结构结合大规模人类数据,构建了目前参数规模最大的单细胞基础模型。为促进科研共享,研究人员计划公开CellFM的源代码和预训练模型,为后续研究者提供统一的基础框架,便于在不同任务中快速部署和应用。
尽管CellFM取得了显著进展,仍存在一些局限性需进一步探索。首先,模型中的注意力机制在识别全局或静态生物学知识相关的基因关系方面仍有限,未来将引入新的可解释性技术加以改进。其次,当前模型仅基于人类数据,缺乏多物种信息,限制了其在跨物种分析中的适用性。此外,模型构建尚未整合生物学先验知识,这可能影响其在深入解析生物机制时的解释深度与准确性。
整理 | WJM
参考资料
Zeng, Y., Xie, J., Shangguan, N. et al. CellFM: a large-scale foundation model pre-trained on transcriptomics of 100 million human cells. Nat Commun 16, 4679 (2025).
https://doi.org/10.1038/s41467-025-59926-5
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢