DRUGAI
小环肽作为治疗手段日益受到关注,然而受限于训练数据不足,深度学习方法在该领域的发展较为缓慢。研究人员在此提出 AfCycDesign,一种用于环肽结构预测、序列重设计与全新构建的深度学习方法。该方法成功设计出超过一万个结构多样的环肽,其预测结构与设计高度一致。八个新设计肽的X射线晶体结构验证了原子级精度(RMSD < 1.0 Å)。此外,研究人员还基于这些构建肽设计出对 MDM2 和 Keap1 具有纳摩尔级 IC50 的结合肽。该方法和骨架为靶向多种蛋白的定制化肽类设计提供了基础。

深度学习方法如 AlphaFold2 和 RoseTTAFold 在根据氨基酸序列预测蛋白质三维结构方面取得了显著成果,并已成功应用于蛋白质结构和相互作用网络的预测。这些结构预测网络也推动了多种功能蛋白的设计方法的发展。然而,相关研究主要集中在由标准氨基酸组成的大分子蛋白上。尽管已有研究显示这些方法在小肽及其复合物结构预测中具有一定适用性,但环肽的设计仍需专门方法。
环状大分子因具备结构稳定性、耐酶性和透膜性等优势,广泛存在于天然产物和治疗性肽类分子中。环肽缺乏自由末端,使其对外切酶更具抵抗力,虽然缺少常规二级结构,但环化约束可稳定其折叠结构。此外,环肽具备干扰胞内蛋白相互作用的潜力,能作用于传统小分子和抗体难以靶向的目标。研究人员此前已使用 KIC 算法结合 Rosetta 设计序列,开展了环肽的计算设计,但该方法计算成本高,需大量采样。
为此,研究人员开发了基于深度学习的环肽结构预测与新分子设计方法。由于高分辨率环肽结构数据稀缺,直接训练专属模型具有挑战。尽管可用合成数据训练模型,但其表现仍受限于数据生成方法的准确性。近期的原子级打分生成模型在小肽构象生成方面取得一定成功,但仍无法实现从零设计。相比之下,预训练网络如 AlphaFold2 可通过引入环化信息,调整输入编码方式来识别环肽,并用于结构预测和设计。
本研究提出一种方法,将N-C末端环化编码为 AlphaFold2 的输入,并评估其在预测已有环肽结构中的准确性。研究人员进一步使用该模型进行序列重设计,以增强环肽折叠倾向。随后,研究人员提出从零生成环肽序列与结构的方法,展示了7–13个残基环肽的丰富结构多样性。最后,研究人员将生成的环肽用作初始骨架,设计出针对特定蛋白靶点的结合肽。
研究结果
环肽结构预测
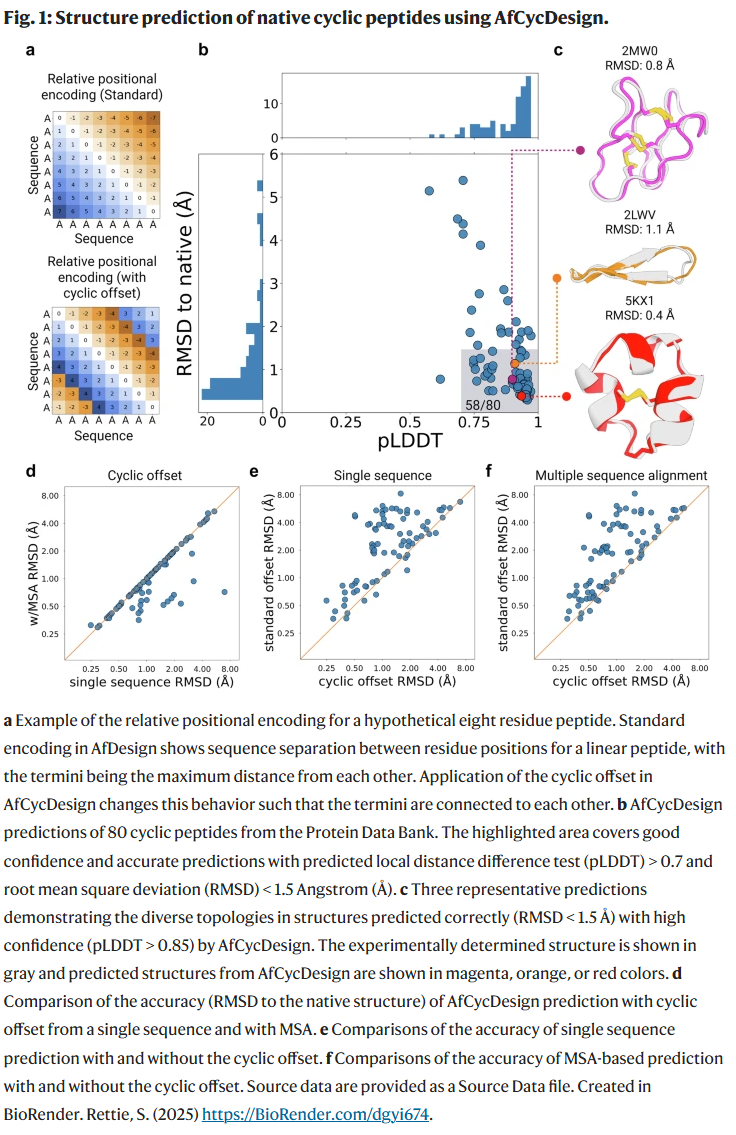
研究人员通过修改 AlphaFold2 的相对位置编码输入,使其适用于环肽结构预测,并在 ColabDesign 框架中实现该方法,命名为 AfCycDesign。该方法在预测环肽终端连接、构象保持和结构稳定性方面表现良好,且对序列循环排列具有稳定性。研究人员评估了 80 个来源于 PDB 的环肽结构,AfCycDesign 在多数情况下能够准确预测其结构,pLDDT 中位数为 0.92,RMSD 中位数为 0.8 Å。在 58 个案例中,预测结构的 RMSD < 1.5 Å,pLDDT > 0.7。更高置信度(pLDDT > 0.85)时准确率更高。此外,即使未指定二硫键连接,AfCycDesign 仍能正确预测其构象。与传统方法如 Rosetta 相比,该方法大幅减少计算成本,仅需数分钟即可完成预测。
环肽序列重设计
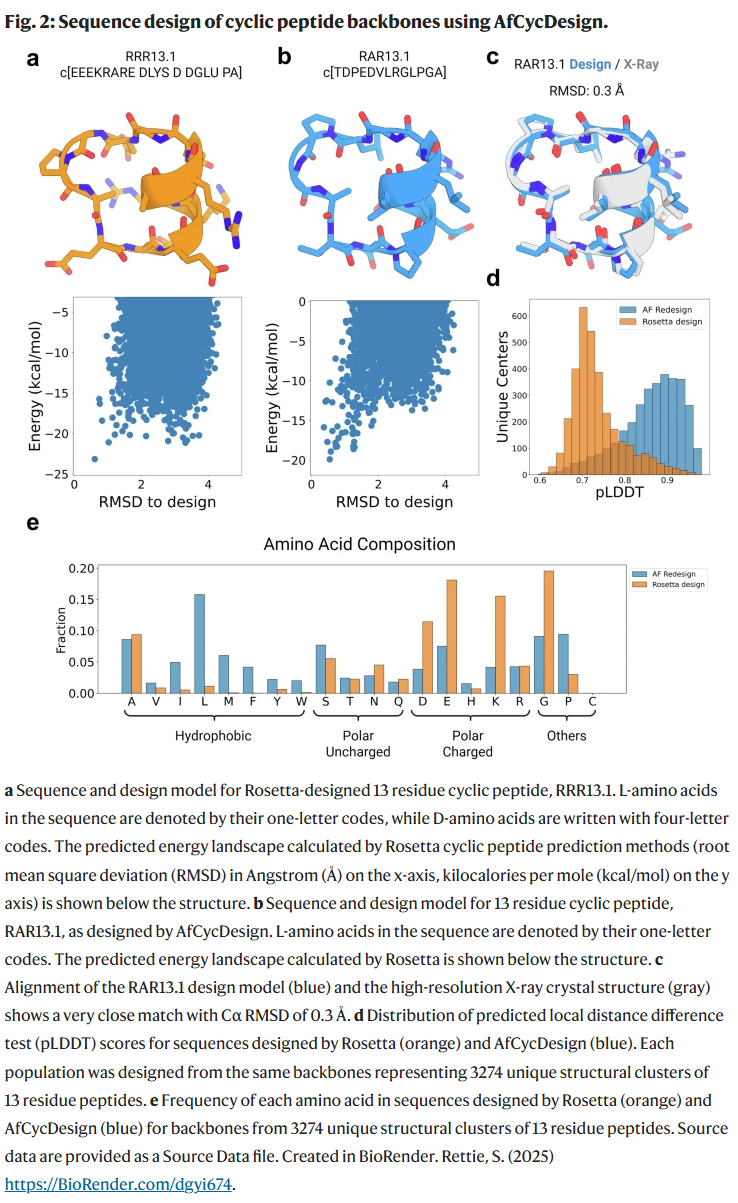
研究人员进一步将 AfCycDesign 应用于环肽骨架的序列优化,以增强其折叠倾向。在构建的大规模 13mer 骨架数据集中,AfCycDesign 所设计的序列相比 Rosetta 结果表现出更高的置信度(pLDDT > 0.9 的序列显著更多)。实验验证显示,所设计的序列 RAR13.1 的晶体结构与预测结构高度一致(Cα RMSD = 0.3 Å),说明 AfCycDesign 能生成高保真结构。
全新环肽设计
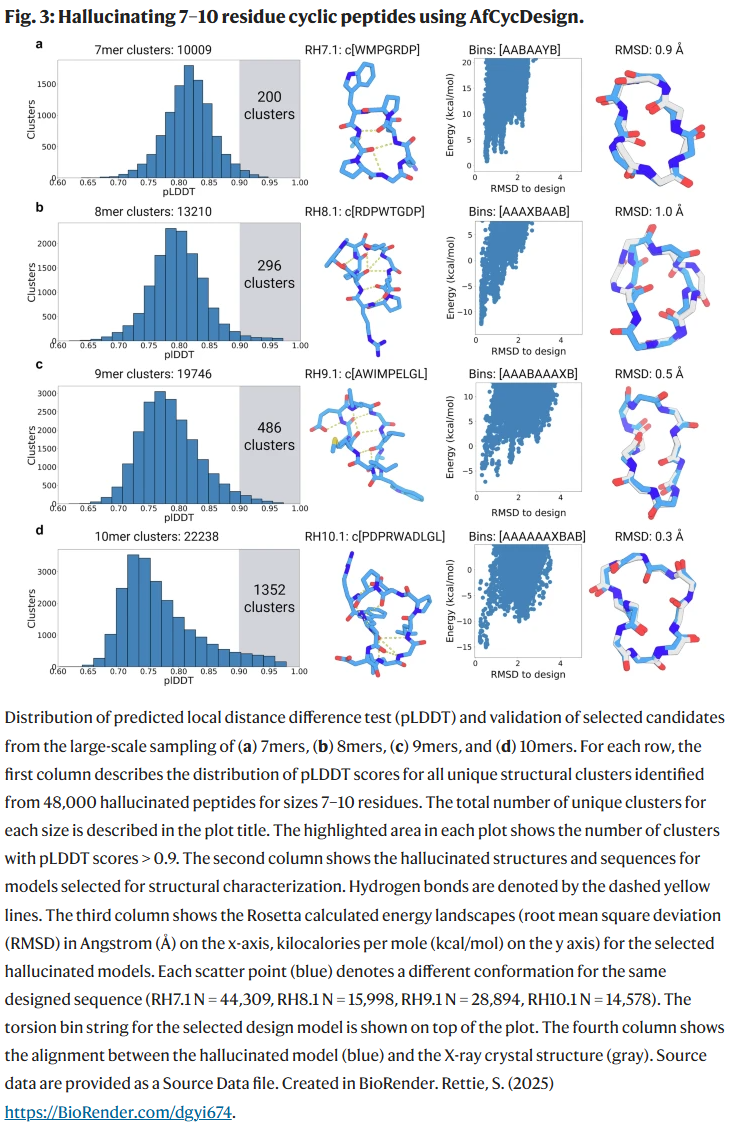
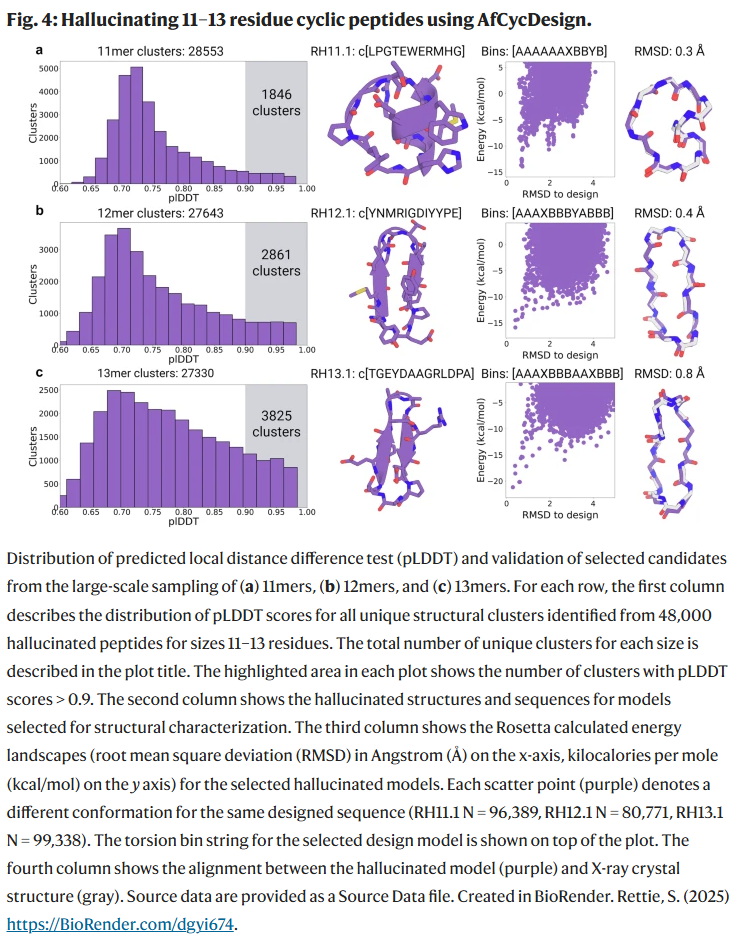
研究人员利用 AfCycDesign 实现了同时生成序列和结构的环肽设计,覆盖 7–13 个残基的多种构象,构建出大量新颖结构簇。高置信度模型(pLDDT > 0.9)经过 Rosetta 能量景观分析进一步验证其折叠倾向。选取的代表性模型(如 RH7.1, RH10.1 等)均通过 X 射线晶体学验证,结构与设计模型高度一致(RMSD 最低为 0.3 Å)。这些肽不依赖规则的二级结构,但通过内部氢键和回转角精确约束形成稳定折叠。
靶向结合肽设计
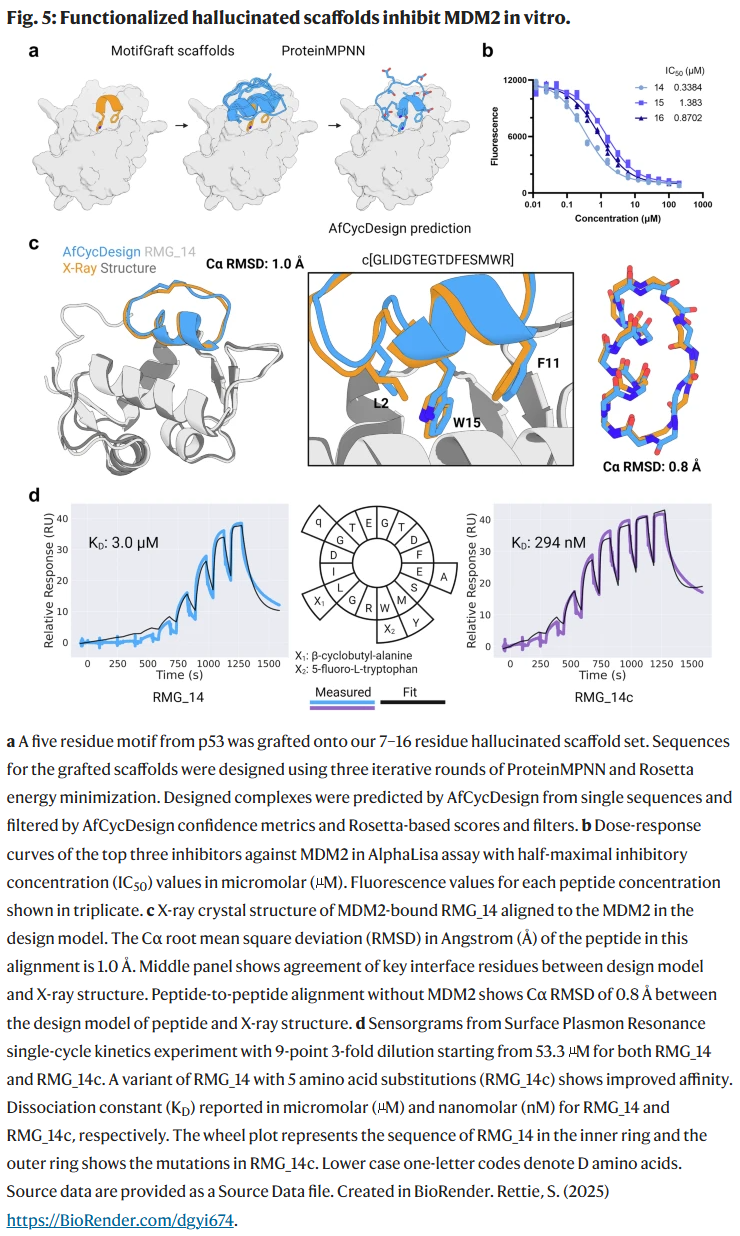
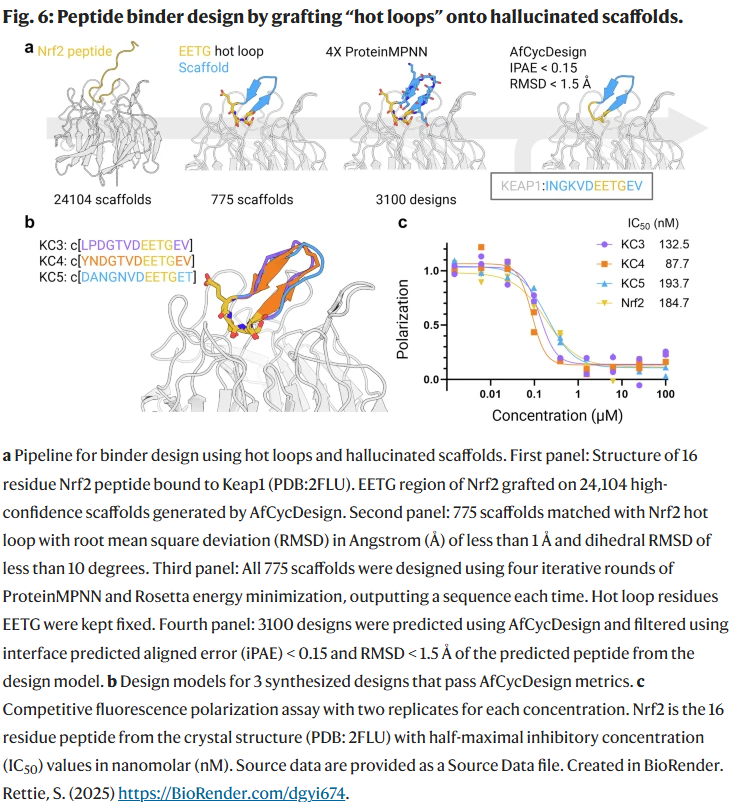
研究人员尝试将高置信度 scaffold 应用于蛋白靶标结合肽设计。以 MDM2 为示例,研究人员将 p53 的结合基序移植至环肽 scaffold 上,设计并合成多个候选肽,其中 RMG_14 展现出纳摩尔级结合活性(IC50 = 338.4 nM),其晶体结构与设计模型一致。进一步测试表明,该 scaffold 可容纳多种结合基序(如 Keap1 的 EETG),并通过设计获得高亲和力结合肽(如 KC4),其效力优于 Nrf2 线性肽。
综上,AfCycDesign 在环肽结构预测、序列设计、de novo 生成以及靶点结合方面展现出强大性能,为多功能肽类药物开发提供了新工具。
讨论
研究人员提出了一种将环状相对位置编码引入蛋白结构预测网络的方法,并将其应用于多个关键任务,包括环肽结构预测、天然或设计骨架的序列重设计、全新环肽结构生成(hallucination),以及靶向蛋白的环肽结合肽设计。在 PDB 环肽数据集中的测试显示,80 个案例中有 58 个预测结构与实验结构高度一致(RMSD ≤ 1.5 Å,pLDDT ≥ 0.7),其中高置信度预测(pLDDT ≥ 0.85)中 80% 的结构 RMSD 低于 1.5 Å,展示了该方法在结构预测方面的出色表现。
在序列重设计任务中,AfCycDesign 设计的序列相较于 Rosetta 方法具有更高的结构置信度和更强的折叠倾向。大规模的 13mer 环肽重设计结果表明,AfCycDesign 更偏好使用疏水性和构象受限的氨基酸。此外,研究人员还拓展该方法用于同时生成序列和结构,构建出涵盖 7–13 个残基、置信度高(pLDDT > 0.9)的 10,681 个环肽结构簇。X 射线晶体学验证显示,所有测试的设计模型(1 个重设计,7 个 hallucination)均与预测结构高度一致(RMSD < 1.0 Å),虽然未能观察到可能存在的其他非晶体构象。
这些高置信度的 hallucinated 环肽及其镜像形式为进一步功能化(如结合蛋白、跨膜转运)提供了坚实基础。研究人员在 MDM2 靶点上验证了该方法,通过移植 p53 的短螺旋结合基序,设计出的结合肽 RMG_14 与晶体结构高度一致。进一步分析显示,已知的 1014 个蛋白-蛋白互作“热点环”中有 798 个可移植至 hallucinated 骨架上,并成功设计出 3 个具有亚微摩尔活性的 Keap1 结合肽。
值得注意的是,这些 hallucinated 环肽仅由 L-型氨基酸组成,仍能形成稳定结构,这突破了此前强调 L/D 氨基酸混合设计的经验。尽管如此,D-型和非天然氨基酸仍可用于优化结合亲和力与稳定性。在 MDM2 结合肽中,研究人员通过引入 5 个位点突变(包括 D-氨基酸和非天然残基),成功提升了结合亲和力约 10 倍。尽管本研究未系统评估所设计肽的酶解稳定性,但未来的大规模实验数据可进一步指导 AfCycDesign 的稳定性导向优化。尽管当前模型不支持直接引入非天然氨基酸,但可与 Rosetta 等物理建模方法结合实现更丰富的化学多样性。
未来研究将聚焦于进一步提升 de novo 环肽结合肽的计算设计能力。深度学习在蛋白设计领域的迅速发展也将推动结构化环肽的定制化设计向更高水平迈进,为治疗性分子的开发提供有力工具。
整理 | WJM
参考资料
Rettie, S.A., Campbell, K.V., Bera, A.K. et al. Cyclic peptide structure prediction and design using AlphaFold2. Nat Commun 16, 4730 (2025).
https://doi.org/10.1038/s41467-025-59940-7
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢