在人工智能驱动的科学研究时代,高质量、大规模的数据集对于机器学习模型的成功至关重要。在化学领域,PubChem和ChEMBL等综合数据库提供了大量有关化合物结构、性质及其生物活性的信息,美国专利商标局(USPTO)数据库则包含丰富的化学反应数据。这些资源使得研究人员能够应用机器学习方法探索新的化学见解、优化反应条件并加速药物发现。然而,尽管关于分子结构和反应的数据极为丰富,但公开可用的化学光谱数据仍存在严重不足,NMR数据的收集仍主要依赖手工,耗时且效率低下。
近日,中国科学院上海药物研究所郑明月课题组,基于微调的大型语言模型开发了NMRExtractor,能够从大量开放获取的文献中提取实验核磁共振(NMR)数据。研究团队通过处理5,734,869篇开源科学出版物,创建了NMRBank,一个包含225,809条记录的数据库,系统收录了化合物IUPAC名称、NMR条件、1H NMR化学位移和13C NMR化学位移、数据置信度以及参考信息。分析表明,NMRBank的化学空间显著超过现有的公开NMR数据库。该提取过程具备高度可扩展性,支持自动处理新的科研论文并持续更新NMRBank。此方法不仅扩展了可用的开放NMR数据空间,还为基于AI的NMR预测及相关化学研究提供了基础。该研究成果于2025年5月28日在Chemical Science期刊上在线发表,题为“NMRExtractor : leveraging large language models to construct an experimental NMR database from open-source scientific publications”。

1. 背景
核磁共振(NMR)光谱是化学研究中强大且应用广泛的技术之一,NMR提供了关于分子环境的详细信息,这些信息对结构和原子间相互作用非常敏感。在过去二十年中,研究人员开发了多个数据库用于存储分子的1H和13C NMR光谱。例如HMDB、NMRShiftDB2和NP-MRD,然而这些数据库的规模仍有限,最大的开放NMR数据库NMRShiftDB2仅包含53,954个实验测得的光谱,涵盖约44,909个分子[1]。
为了解决数据稀缺问题,Jia等人开发了SRCV,一种基于机器学习的NMR光谱识别系统,但该系统依赖标准化的NMR图像,无法提取诸如化合物结构和测量条件等相关信息[2]。近年来,大型语言模型(LLMs)如ChatGPT展现了强大的文本理解与处理能力,使其成为挖掘科学文献的有前景工具。该团队之前的研究表明,微调后的LLMs在多项化学文本提取任务上表现优异,本地微调开源LLM如Mistral-7b-instruct-v-0.2是可行的替代方案,不仅在性能上可与主流模型媲美,而且计算成本更低,更适合私有部署[3]。在本研究中,提出的NMRExtractor,是一个高精度、易扩展的NMR数据提取工具。团队成员使用该工具构建了NMRBank,一个包含225,809条实验NMR数据记录的数据集。NMRExtractor能够自动处理新的研究论文,确保NMRBank可以不断更新和扩展,从而满足化学研究中对全面和最新NMR数据的持续需求。
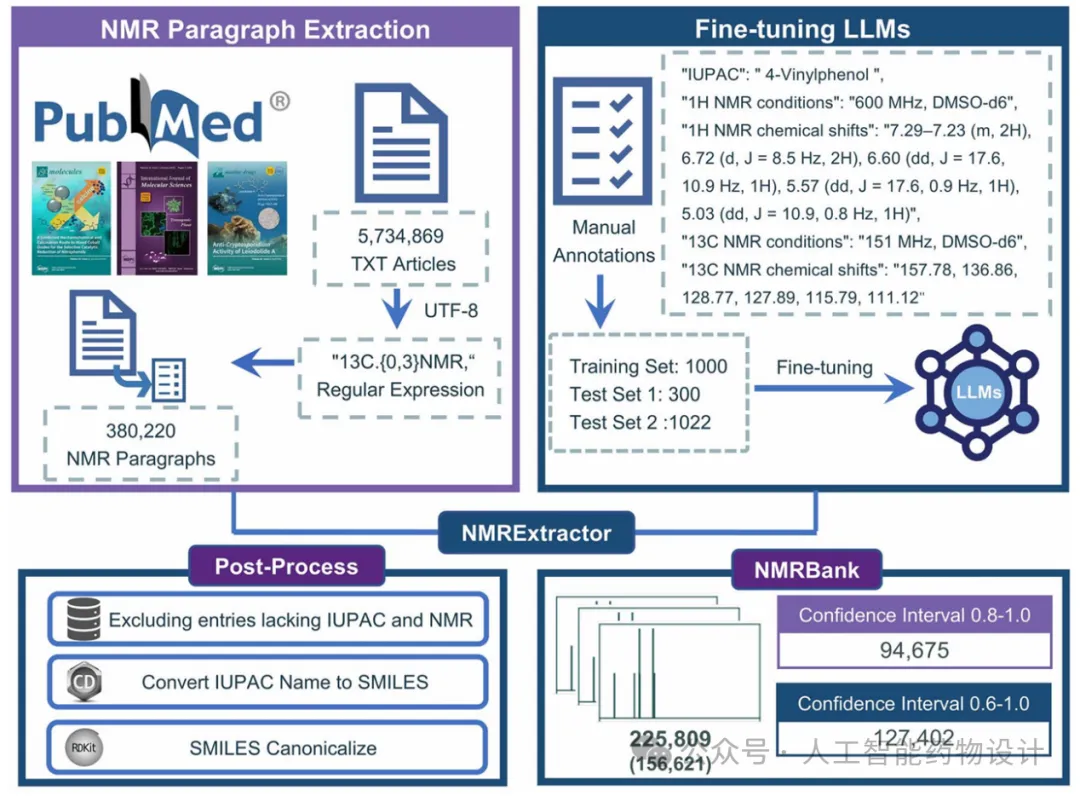
图1. NMRExtractor提取流程和NMRBank数据集构建的示意图。
2. 结果与讨论
2.1 使用NMRExtractor提取NMR数据的工作流程
使用NMRExtractor提取NMR数据并构建NMRBank的过程如图2所示。首先访问PubMed中所有开放获取的TXT文档,并将其统一转换为UTF-8编码格式,然后使用正则表达式提取所有提及NMR数据的文本段落。随后,从这些段落中使用大型语言模型提取NMR数据,仅保留IUPAC名称非空的数据,并进一步筛除1H和13C NMR化学位移为空的数据。最后,将化合物的IUPAC名称转换为SMILES,并对SMILES进行标准化。
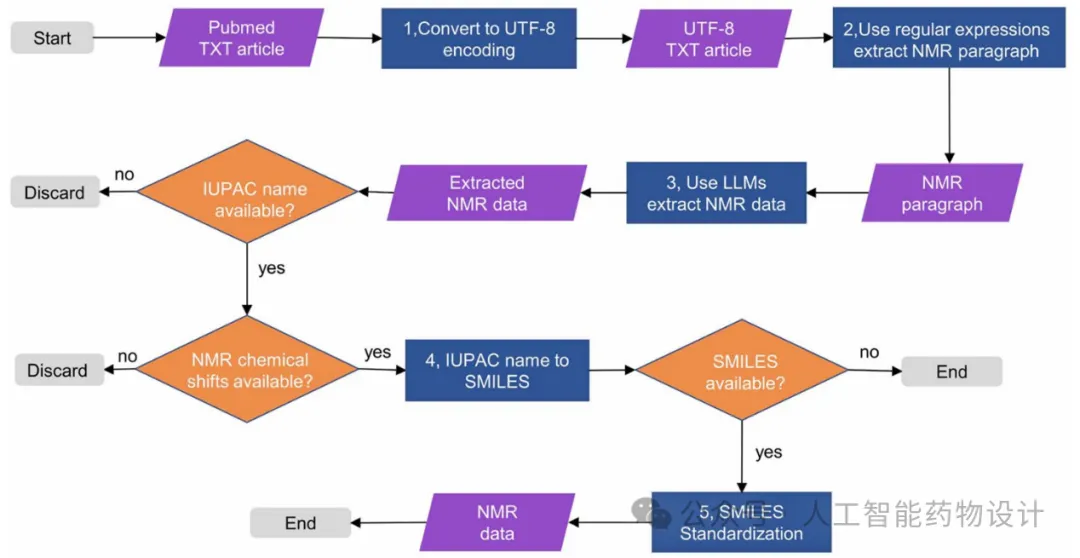
图2. 使用NMRExtractor提取NMR数据的工作流程。
2.2 数据准备
研究团队获取了PubMed数据库的5,734,869篇开放获取文章,并通过基于规则的方法从其中58,795篇文章中识别出380,220个提及NMR的段落(图3a)。其中,大约260,000个段落包含NMR数据,来自35,270篇文章(图3b)。为了增强模型评估的全面性,在之前的300条测试数据(测试集1)的基础上,构建了一个更具多样性的测试集,包含1022条2023年发表的NMR数据段落(测试集2)。这些段落来自115种不同类型的期刊,由化学专家人工标注,并根据文献中NMR数据的表述格式将这1022个段落分类为标准化描述和非标准化描述(图3c)。此外,研究团队还在之前300条训练数据的基础上,另外随机选取并标注了700条2023年前发表的NMR段落,使训练集扩大至1000条。
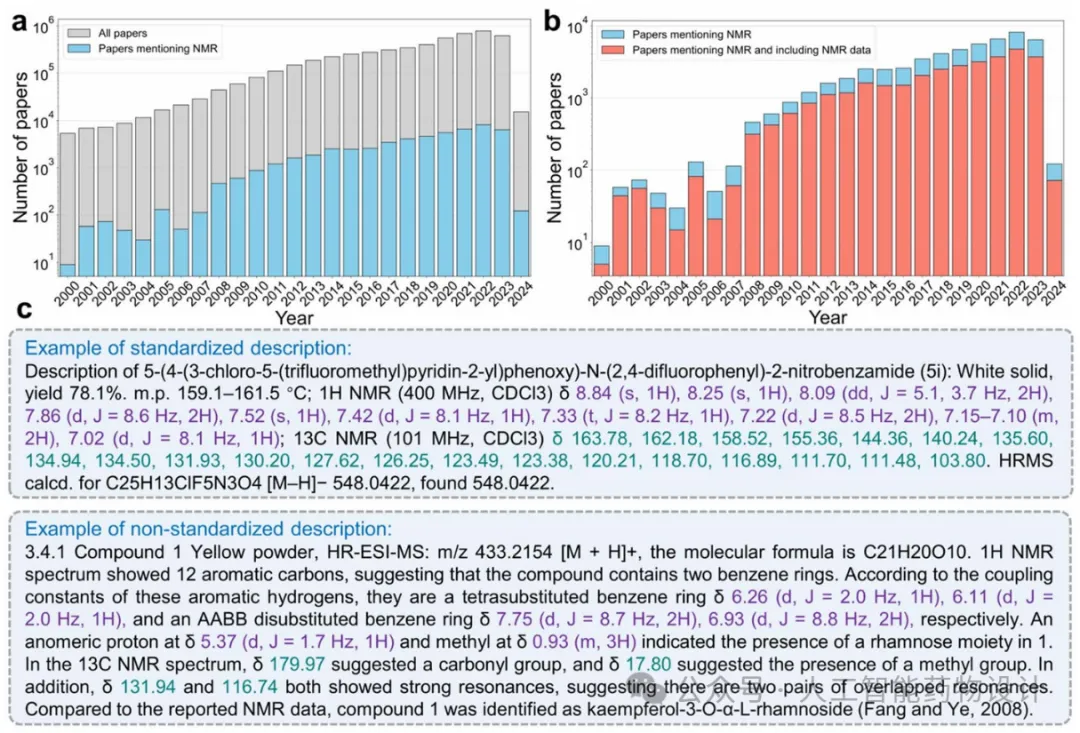
图3. 论文数量和NMR数据格式描述。(a)PubMed中的开放获取论文总数(灰色)与提及NMR(蓝色)的论文总数。(b)在PubMed 中提及NMR(蓝色)和包含NMR 数据的论文(红色)。(c)论文中标准化和非标准化NMR数据描述的示例。注意:该数据基于PubMed 2024年6月更新,因收录尚不完整,2023–2024年的数据代表性不足。
2.3 模型性能
如图4a所示,随着训练集规模的扩大,微调后的Mistral-7b-instruct-v-0.2模型在不同测试集上的表现持续提升并逐渐收敛。在800条训练样本时,模型在多个测试集中均达到最优表现,优于Llama3-8b-instruct与Llama2-13b-chat模型。在测试集1(300条)中,各元素抽取的准确率均超过0.96,在测试集2(1022条)中也超过0.85。进一步分析测试集2,根据文本描述方式划分后发现,在标准化描述部分(test set 2_standard, 788条)中,各元素抽取的准确率超过0.9;而非标准化描述部分(test set 2_non-standard, 244条)则因语言结构复杂、信息缺失等问题对提取造成一定困难,导致性能略有下降。为进一步验证模型提取结果的完整性与准确性,研究团队统计了“同时正确提取IUPAC名称和NMR化学位移”的样本比例。只有在两者均准确时,该预测才被计为正确。随着训练样本扩展至800条,在测试集2中,IUPAC名称与1H NMR化学位移的联合准确率达到0.78,IUPAC名称与13C NMR化学位移的联合准确率达到0.804(图4b)。
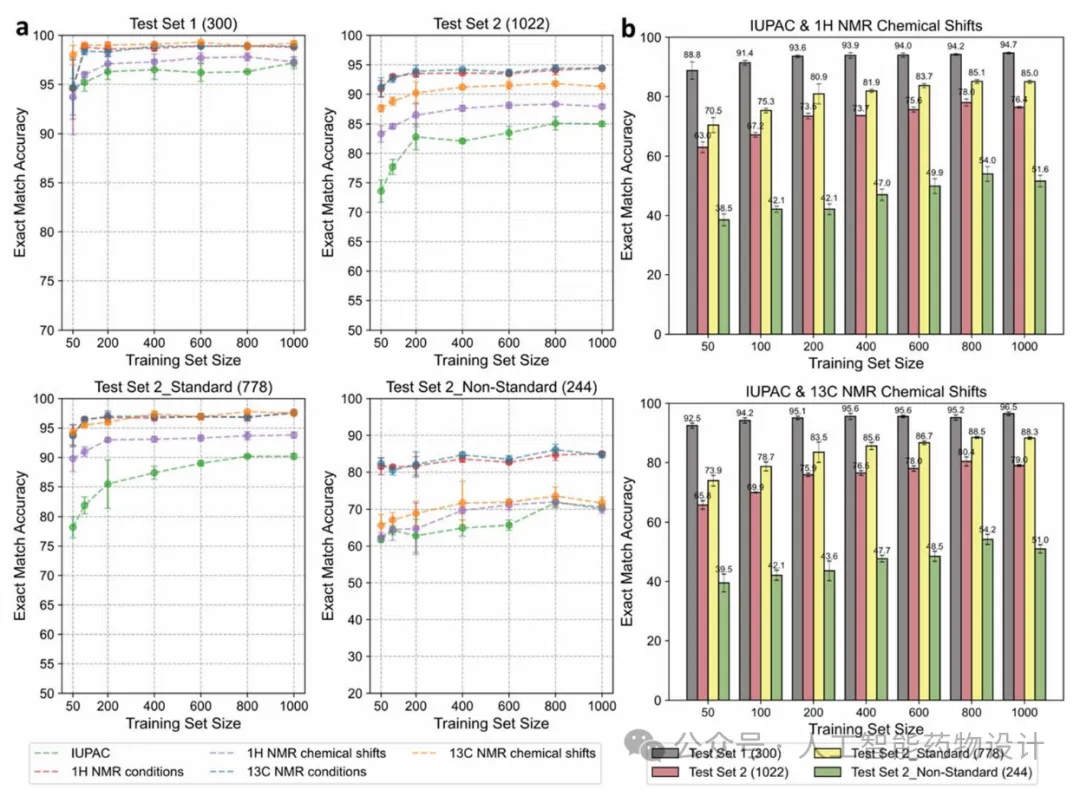
图4. NMRExtractor模型性能。(a)微调后的Mistral-7b-instruct-v0.2在不同测试集上的表现随着训练集数量的变化。(b)IUPAC名称和1H/13C NMR化学位移均正确的提取准确度随着训练集数量的变化。误差线表示在三次独立采样训练数据的运行中所得标准差。
2.4 数据置信度和准确性
为客观评估NMRExtractor的性能,研究团队不仅考察了整体准确率,还分析了模型对每条数据的置信度评分。置信度范围为0到1,基于预测Token的累计对数概率进行计算。在测试集1(300条)与测试集2(1022条)中,当置信度超过0.6时,所有数据的抽取准确率超过86%;当置信度超过0.8时,准确率超过97%(图5a、b)。在训练集上进行三折交叉验证的结果进一步表明,随着置信度升高,模型性能稳步提升。据以往研究,人工标注的生物活性数据库(如ChEMBL和WOMBAT)中的错误率约为5%,因此研究团队认为,当置信度超过0.8时,模型预测可达到人工标注的水平,支持其在大规模应用中的可靠性[4]。在测试集1中,有261条实体的置信度高于0.6,其中209条置信度超过0.8;在测试集2中,有594条实体置信度高于0.6,386条置信度超过0.8(图5a、b)。标准化描述测试集(test set 2_standard)具有较高置信度值与较好表现;而非标准化描述测试集(test set 2_non-standard)整体性能较低,但当置信度高于0.8时,其元素提取准确率仍超过88%(图5d)。这表明在批量抽取数据后可依据置信度对结果进行筛选,从而提高整体数据质量。
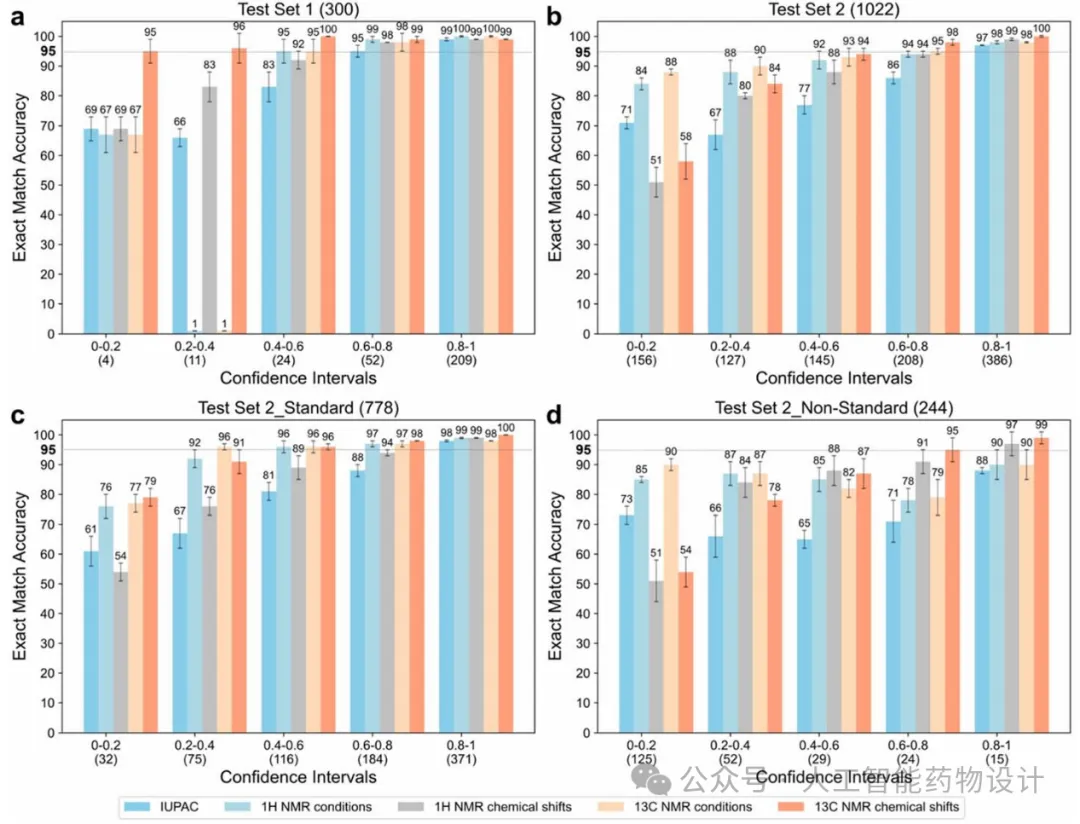
图5. NMRExtractor在不同测试集和置信区间下的性能表现。每个置信区间中的数据数量显示在其下方。(a)测试集1(300条数据)在不同置信区间下的性能表现。(b)测试集2(1022条数据)在不同置信区间下的性能表现。(c)测试集2_standard(778条数据)在不同置信区间下的性能表现。(d)测试集2_nonstandard(244条数据)在不同置信区间下的性能表现。误差线表示在三次独立采样训练数据的运行中所得的标准差。
2.5 数据提取方法比较
在使用微调后的大型语言模型进行NMR数据提取之前,研究团队也探索了传统的基于规则的方法。初步尝试中,研究团队依据文献中NMR数据的标准描述方式书写正则表达式,识别包含NMR信息的段落,并在此基础上进一步解析出NMR数据。对于IUPAC名称,研究团队整理了618个常见化合物基团词列表,通过文本扫描提取相关信息。然而,在实际应用中,该方法存在显著局限性:由于文本多样性强,规则需不断调整,且难以覆盖所有情况;同时,文中存在的反应物与溶剂信息也常导致提取错误或遗漏。与NMRExtractor相比,基于规则的方法在测试集1和测试集2中的表现均明显较差(图6a、b)。此外,在批量处理任务中,基于LLM的方法还能依据置信度对数据进行质量评估,而基于规则的方法则无法提供准确性评估。
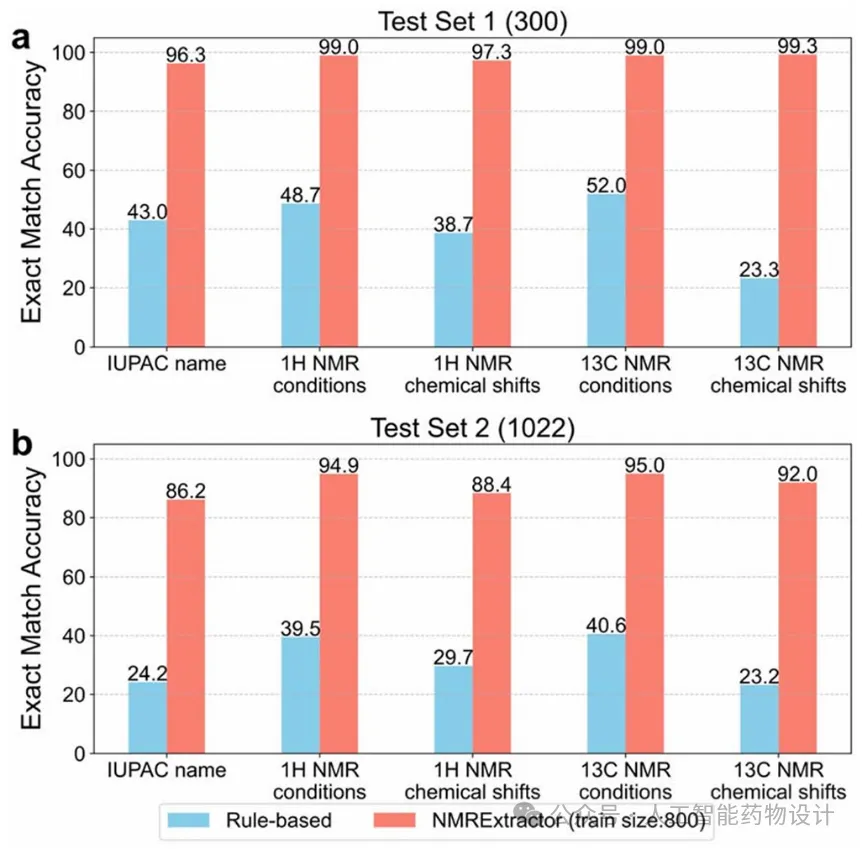
图6. 基于规则的方法(蓝色)与NMRExtractor(红色)在(a)测试集1(300个样本)和(b)测试集2(1022个样本)上的性能对比。
2.6 核磁共振数据提取和分析
研究团队使用NMRExtractor批量处理了380,220条NMR段落。去除13C NMR化学位移为空的记录后,获得约260,000条数据。进一步去除IUPAC名称和NMR化学位移均为空的记录后,最终保留225,809条数据。研究团队使用ChemDraw与OPSIN工具将IUPAC名称转换为SMILES格式,共成功转换156,621条,并使用RDKit对其标准化处理,构建出最终的NMRBank数据集。在这些记录中,共有149,135条为唯一SMILES结构,约总记录的66%。这一过程确保了结构重复项的识别,有助于与现有数据库进行更准确对比。与公开NMRShiftDB2数据库的比较显示,NMRBank在分子量、TPSA、logP等理化性质分布上更广,所覆盖的化学空间更大(图7)。
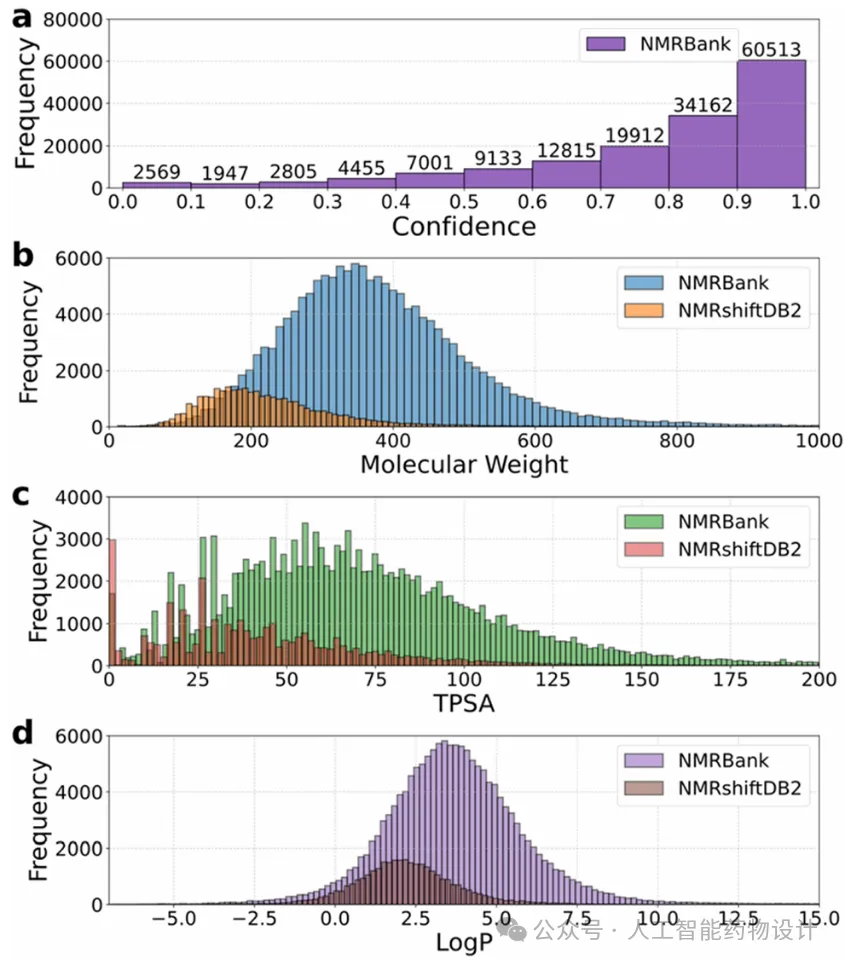
图7. NMRBank与NMRShiftDB2中分子性质的分布对比。(a)NMRBank中数据的置信度分布。(b)NMRBank和NMRShiftDB2 中分子的分子量分布。(c)NMRBank和NMRShiftDB2中分子的拓扑极性表面积(TPSA)分布。(d)NMRBank和NMRShiftDB2 中分子的计算logP分布。
2.7 现有NMR数据库
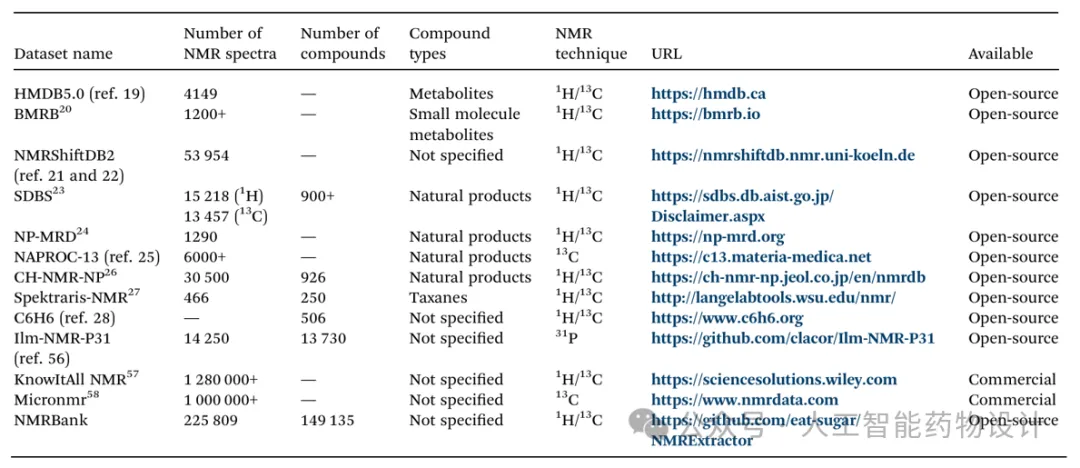
除了分析统计特性外,研究团队还将NMRBank与最常用的开源NMR数据库进行了比较(表1)。结果显示,NMRBank的数据数量显著多于任何现有的开源NMR数据库。此外,该NMR提取流程具备出色的可扩展性。借助NMRExtractor,能够快速自动地处理新文献,大大促进了NMRBank数据库的持续更新。
表1. 主要NMR数据库。
2.8 NMRBank数据集
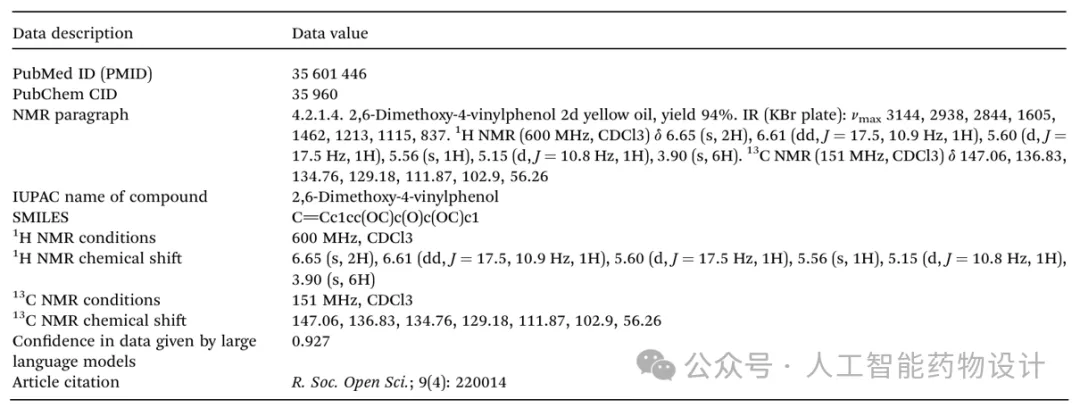
本研究构建的NMRBank 数据集可在https://github.com/eat-sugar/NMRExtractor获取。 NMRBank数据集中的每条记录均包括:文章PMID、NMR段落、化合物IUPAC名称、SMILES、1H NMR 条件、1H NMR化学位移、13C NMR条件、13C NMR化学位移、置信度评分,以及其他如文章信息等元数据。表2概述了NMRBank数据集中的信息项,表3展示了其中一条数据示例。
表2. NMRBank数据集概览。
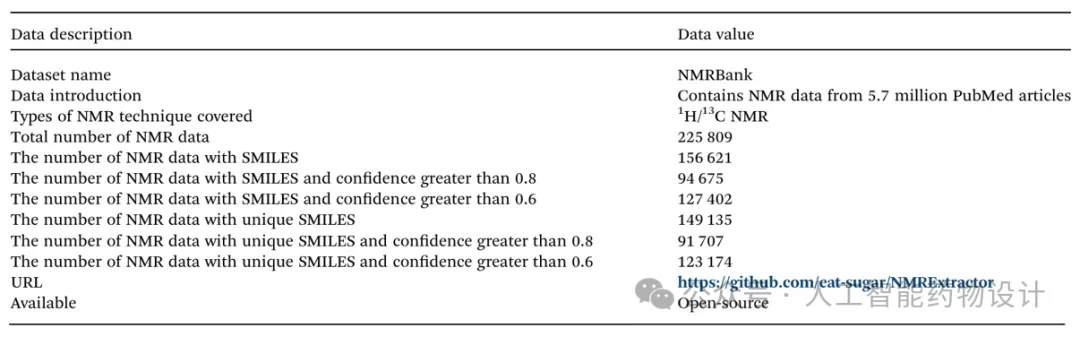
表3. NMRBank数据集数据示例。
2.9 AI 数据提取与算法迭代的未来展望
随着 AI 技术在科研数据提取与分析领域的不断深入,我们正站在一场科学范式变革的门槛之上。类似NMRExtractor这样的AI数据提取技术探索只是这场变革的序章。未来,AI 系统有望实现从文献数据提取到算法自我强化的完整闭环,彻底重塑科学研究的流程与边界。想象一下,AI 模型从海量文献中高效提取数据,构建起庞大而精准的数据库。这些数据不仅是不断积累的知识,更是 AI 算法进一步学习与进化的新养料。随着新数据的持续涌入,AI 算法能够实时自我迭代,不断优化提取与分析的准确性与深度。这种自我强化机制,将使 AI 在科学研究领域的应用潜能呈指数级增长,加速新材料的发现、药物的研发,以及生物或化学反应机理的剖析。
然而,这条充满希望的道路也并非一帆风顺。AI 模型的自我迭代可能带来不可预测性,其决策过程的 “黑箱” 特性或使科研人员难以理解与控制。数据的隐私与安全问题同样不容忽视,如何在开放数据共享与保护知识产权之间寻求平衡,是整个科研界亟待解决的难题。此外,跨学科人才的短缺可能制约这一领域的快速发展,培养既懂领域知识又精通 AI 技术的复合型人才,将是推动这一变革的关键所在。
3. 结论
在本研究中,研究人员基于大型语言模型开发了NMRExtractor,从570万篇PubMed文献中自动提取NMR数据,构建了包含22万余条记录的NMRBank数据库。相比现有开源数据库,NMRBank在规模和化学空间覆盖上更具优势,为基于化学光谱数据的深度学习建模、结构预测和药物研发提供坚实的数据支持。
南京中医药大学与上海药物研究所联合培养硕士研究生王庆功、中国科学院上海药物研究所博士研究生张玮为本文的共同第一作者。中国科学院上海药物研究所郑明月研究员、博士后熊嘉诚、上海科技大学助理研究员付尊蕴为本文通讯作者。本研究得到了国家自然科学基金、国家重点研发专项、上海药物研究所与上海中医药大学中医药创新团队联合研究项目、上海市超级博士后计划、上海市科技重大专项项目的资助。
参考文献
[1]S. Kuhn, H. Kolshorn, C. Steinbeck and N. Schlörer, Twenty years of nmrshiftdb2: A case study of an open database for analytical chemistry, Magn. Reson. Chem., 2024, 62, 74-83.
[2]W. Jia, Z. Yang, M. Yang, L. Cheng, Z. Lei and X. Wang, Machine Learning Enhanced Spectrum Recognition Based on Computer Vision (SRCV) for Intelligent NMR Data Extraction, J. Chem. Inf. Model., 2021, 61, 21-25.
[3]W. Zhang, Q. Wang, X. Kong, J. Xiong, S. Ni, D. Cao, B. Niu, M. Chen, Y. Li, R. Zhang, Y. Wang, L. Zhang, X. Li, Z. Xiong, Q. Shi, Z. Huang, Z. Fu and M. Zheng, Fine-tuning large language models for chemical text mining, Chem. Sci., 2024, 15, 10600-10611.
[4]P. Tiikkainen, L. Bellis, Y. Light and L. Franke, Estimating Error Rates in Bioactivity Databases, J. Chem. Inf. Model., 2013, 53, 2499-2505.
原文链接
Qinggong. Wang, Wei. Zhang, et.al., NMRExtractor: leveraging large language models to construct an experimental NMR database from open-source scientific publications, Chemical Science, 2025, DOI: 10.1039/D4SC08802F.
https://pubs.rsc.org/en/content/articlepdf/2025/SC/D4SC08802F(点击下方阅读原文跳转)

内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢