DRUGAI
分子表示是本研究理解物质世界的关键要素,也是现代分子机器学习的基础。以往的分子机器学习模型通常使用字符串、指纹特征、全局特征以及简单的分子图,这些表示方式本质上信息较为稀疏。然而,随着预测任务复杂度的提升,分子表示需要编码更高保真度的信息。本研究提出了一种新的方法,通过立体电子效应将富含量子化学信息的数据注入分子图中,从而增强其表达能力与可解释性。通过定制的双图神经网络流程学习预测含立体电子信息的分子表示,使该表示能够应用于任何下游的分子机器学习任务,而无需昂贵的量子化学计算。本研究表明,显式地加入立体电子信息能显著提升二维图神经网络模型在分子性质预测任务中的性能。此外,本研究还展示了在小分子上训练得到的分子表示可以准确地外推至更大的分子结构,揭示了先前难以处理的体系(如完整蛋白质)中轨道相互作用的化学机理,为分子设计开辟了新途径。

分子表示是化学中的基石之一。遵循化学家的直觉,骨架结构逐渐成为化学的通用语言。它们在保持简洁性的同时,能够涵盖种类繁多的(主要是有机)分子,并使人类更容易识别其中的共性模式。这类表示不仅影响了本研究思考和描述化学的方式,也推动了分子机器学习的发展,后者已被广泛应用于多个领域。
分子机器学习最成功的应用之一是分子性质的预测,这是化学、生物学和材料科学的核心任务。从太阳能电池材料的发现,到刷新记录的新药开发,分子机器学习通过快速推理显著推动了现代科学的发展。而这些机器学习模型的性能在很大程度上取决于其所基于的分子表示,这可以说是模型成功的最关键因素。
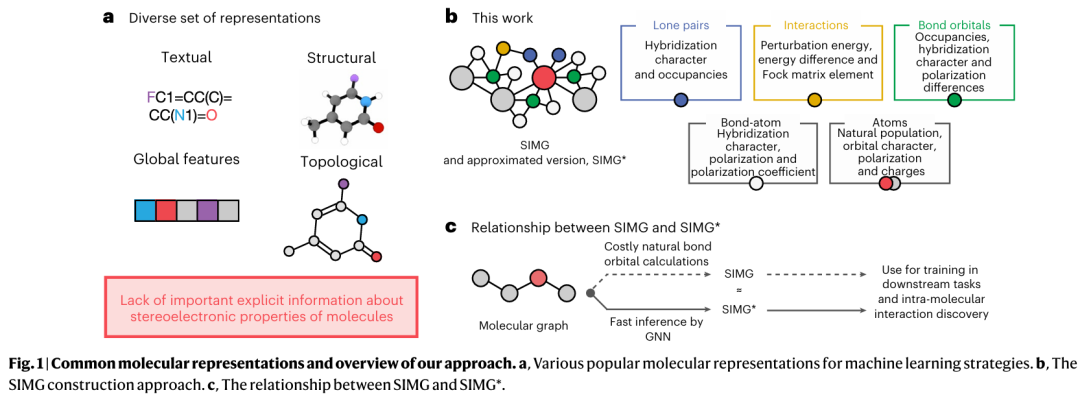
当前标准的分子表示包括多种方式:如全局描述符、将分子结构转化为符号序列的字符串形式、以及编码共价键信息的图结构表示,这些方法提供了分子的拓扑信息(见图1a)。此外,还有一些方法将空间特征注入这些表示中,从而提供结构信息。近期的一些研究还展示了将力场表示与电子结构描述结合,可以同时预测结构性质和电子性质。一些重要的表示方式在库仑矩阵特征值的基础上进行了扩展和改进。最终,表示方式的选择通常也与相应的模型架构和数据处理方式密切相关。许多方法在构建过程中引入了对称性考虑,例如通过多个局部坐标系的平均(类似于 AlphaFold 3 中的方法)或在模型层面上进行调整。
尽管机器学习在分子性质预测中取得了显著成功,目前所使用的分子表示仍存在不完整性。现有广泛使用的图结构表示缺乏来自分子电子结构的量子化学先验信息(见图1a),并且其可解释性十分有限。然而,计算化学领域已经发展出一套强大的技术体系,用于通过量化轨道间的相互作用来描述分子结构的量子力学本质。这类“立体电子”信息提供了宝贵的基础化学见解。例如,成键轨道、非成键轨道与反键轨道之间的相互作用对本研究理解许多化学现象(如蛋白–底物相互作用、有机催化反应)具有重要意义。
立体电子信息的价值表明,只要在下游训练与推理中能避免对输入结构进行高成本的立体电子特征计算,其成功引入有望显著提升机器学习模型的表现。
本研究提出了一种新的分子表示方式,基于分子图结构,并增强引入了表示成键轨道、孤对电子及其相互作用的节点(实质上编码了三维关系信息),称为 SIMGs(stereoelectronics-infused molecular graphs,立体电子注入的分子图)。本研究展示了如何通过自然键轨道(NBO)分析数据构建 SIMG 表示(见图1b),并通过图神经网络进行近似建模(称为 SIMG*),以实现快速预测(见图1c)。本研究系统研究了将 SIMG 表示作为二维消息传递机器学习算法输入的优势,并探讨了 SIMG* 在那些无法直接进行量子化学 NBO 计算的体系中识别立体电子轨道相互作用的能力,从而揭示了先前无法获得的化学机制。
结果
SIMGs
本研究提出的表示方式基于 NBO 分析和一种新的异质图结构拓扑。NBO 分析起始于一个给定分子的三维结构以及其有效波函数的描述。NBO 分析会生成一组局域的自然原子轨道、杂化轨道、成键/非成键轨道以及反键轨道。这一过程涉及对 Hartree–Fock 推导的量子化学方法中的 Fock 矩阵项进行一系列变换,包括杂化密度泛函近似。
此外,NBO 分析还能通过二阶微扰方法量化已占据轨道(供体)与空轨道(受体)之间的相互作用。这种策略为电子密度离域的量子力学本质提供了定量描述,非常适合作为异质分子图表示的一部分。
本研究构建的 SIMG(Stereoelectronics-Infused Molecular Graph)正是基于这些 NBO 分析数据(见图1b)。与传统分子图仅使用原子节点和共价键边不同,SIMG 还引入了多种新的立体电子节点与边类型。除了原子节点,本研究还引入了孤对电子节点(n),成键轨道节点(σ、π)及反键轨道节点(σ*、π*),它们用于表示电子密度轨道,还包括表示轨道间相互作用的节点。虽然已有研究尝试隐式注入此类信息,本研究则选择显式建模以增强可解释性和对图结构的控制力。
此外,本研究还将拓扑扩展到将数值 NBO 分析信息注入 SIMGs 中(见图1b)。例如,**自然电荷分析(natural population analysis)**为每个原子提供了局域电子信息,并作为节点特征被存储在 SIMGs 中,具体包括:原子电荷、核电子数、价电子数和总电子数。对于成键轨道,本研究包含了原子级别的杂化轨道特征、键极化程度及其系数,以及相应的反键轨道特征;对于非成键轨道(如孤对电子),其杂化信息也被包含在内。供体–受体轨道相互作用被表示为相应节点之间的连接,并附带数值特征描述。
使用图神经网络近似 SIMG
虽然 SIMG(立体电子增强分子图)在性能和可解释性方面具有显著优势,但其构建依赖于计算开销极大的 NBO(自然键轨道)分析。对于大规模分子结构数据集或构象集合,这种方法会耗费大量时间;而对于蛋白质等大分子体系,NBO 分析甚至根本无法完成,这是由于 NBO 能处理的基函数数量存在限制。当前主要的替代方案是对大分子进行结构截断,但这种做法会严重限制本研究对其全局立体电子性质的研究能力。因此,本研究提出了一种方法,用于仅从分子的三维结构(例如笛卡尔坐标)快速预测 SIMG 图,称为 SIMG*。这种近似表示可以在不显式进行 NBO 计算的前提下,捕获关键的立体电子信息。
SIMG* 构建的基本流程
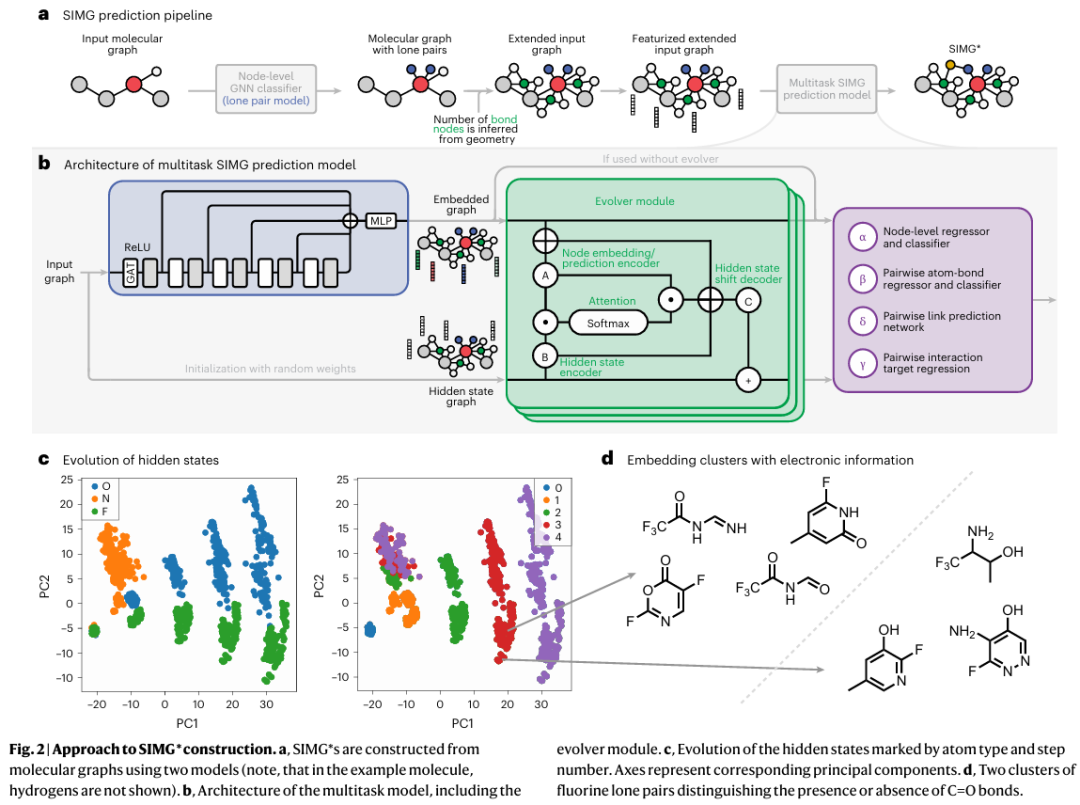
本研究针对传统分子图缺乏非成键轨道(如孤对电子)信息的问题,将其公式化为一个序贯图构建任务,并通过神经网络高效实现。首先,根据分子的三维坐标构建基础分子图,并使用第一个模型预测每个原子的孤对电子数量与类型。随后,基于预测结果生成扩展图,引入孤对电子节点和区分 σ 键与 π 键的轨道节点,提供更丰富的电子结构表达。最终,这一扩展图被输入至第二个图神经网络,通过多任务学习预测轨道间的电子重叠性质,构建出包含立体电子信息的 SIMG* 表示。
多任务模型的架构
多任务模型以扩展分子图作为输入,并将其传入一个基于图神经网络(GNN)的编码器中(见图 2b)。该编码器使用图注意力层,并引入跳跃连接(skip-connection)以解决过度平滑的问题(详见方法部分)。编码器的输出是图中每个节点的一组嵌入表示。
由于存在如下挑战,本研究采用了双重方法从这些嵌入中获得 SIMG*:本研究在输入的图中显式引入了孤对电子节点和键节点,而这些节点可能具有不同的预测目标。例如,在呋喃分子中,氧原子有两个孤对电子:一个为纯 p 轨道,另一个为 spλ 杂化轨道。后者可参与氢键和其他非共价相互作用,但在输入图中它们的特征是相同的。为了解决这一问题,本研究引入了以下两种方法:
赋予可区分的特征:为最初特征相同的节点分配能区分其差异的特征。通过构建一个孤对电子模型,预测其为主要 p 或主要 s 轨道特性(具体见方法部分)。
引入随机隐藏状态并多次更新:采用五个 evolver 模块组成的网络块(见图 2b),在节点目标的中间预测中结合节点嵌入来更新这些隐藏状态,并使用置换不变的损失函数。
evolver 模块的操作基于隐藏状态的迭代更新,因此本研究可以研究嵌入在迭代过程中的变化情况。图 2c 显示了节点隐藏状态的主成分分析(PCA)图,分别按元素种类与时间步长着色。最初几乎无法区分的节点嵌入,随着模型的迭代被逐渐拉开。此外,像氟这种元素在嵌入空间中出现了两个不同的聚类。进一步分析发现(见图 2d),氟原子的两个聚类仅因是否存在 C=O 双键而不同。
基于不确定性估计的 SIMG* 主动学习
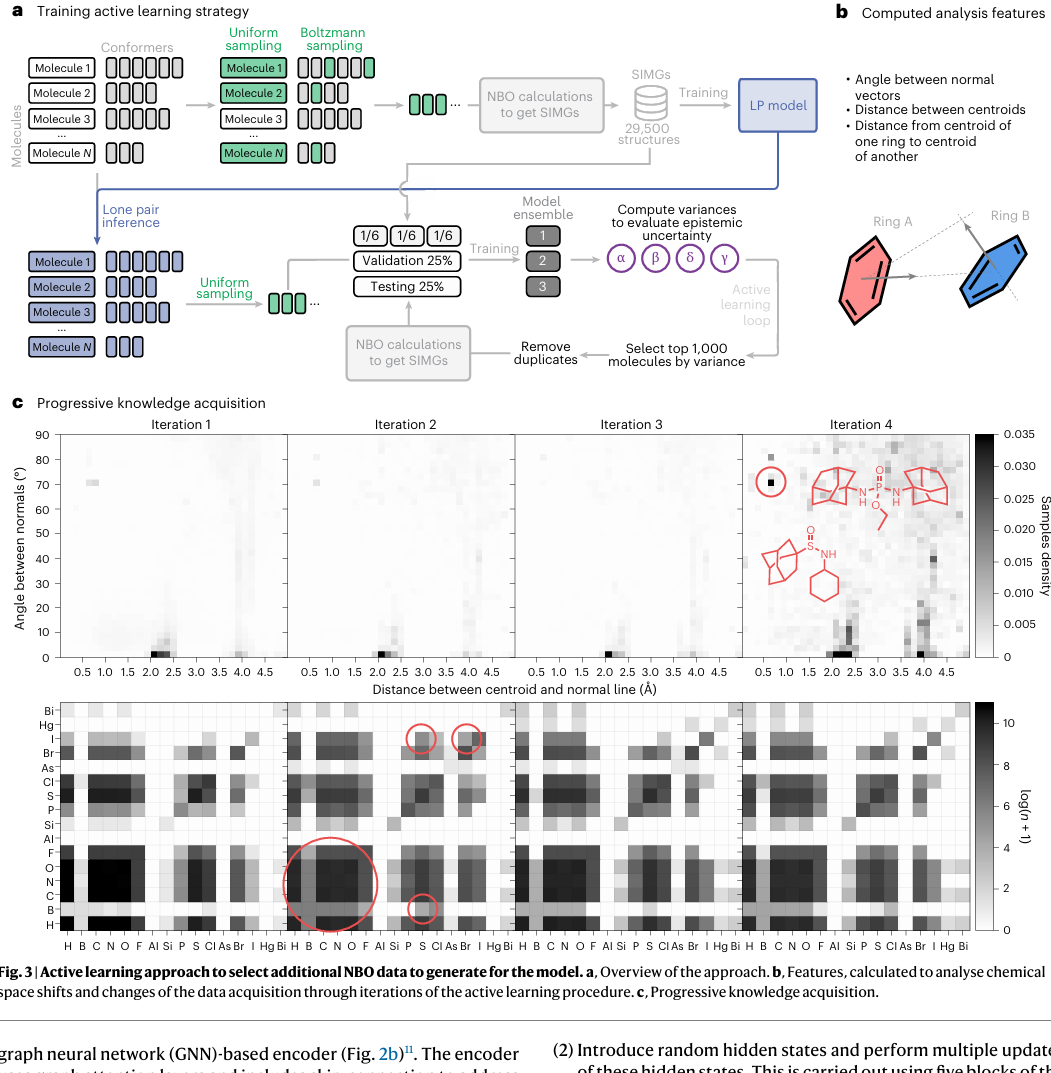
本研究的主要目标之一是实现在涵盖常见生物分子的元素集合上进行高效预测。其中一个具备足够元素种类、多样分子尺寸与构象的代表性数据集是 GEOM 数据集。然而,该数据集规模庞大,使得直接在其上进行 DFT + NBO 计算代价过高。因此,本研究采用了主动学习(Active Learning)策略进行训练(见图 3a)。
该方法的核心在于对 SIMG* 预测中的不确定性进行估计。这些不确定性可以分为两类:偶然不确定性(aleatoric uncertainty)与认知不确定性(epistemic uncertainty),在本研究场景中二者都具有重要意义。尽管如贝叶斯神经网络(Bayesian Neural Networks)等方法在理论上可以严谨地估计不确定性,但其训练难度较高。因此,本研究在实践中采用了**模型集成(ensemble)**的方法,通过多个模型预测结果的方差来估计不确定性,这已被证实具有良好效果。
本研究采用的具体流程如下:
从 GEOM 数据集中均匀采样 29,500 个结构,并对其进行 NBO 计算;用这些数据训练一个孤对电子预测模型以及三个多任务 SIMG* 预测模型的集成;对 GEOM 数据集中的所有结构先运行孤对电子模型,构建扩展分子图;将整个数据集划分为 295 个子集,从每个子集中选取 10,000 个分子输入模型集成中进行预测;对每个分子和预测目标,计算模型集成的预测方差;对每个目标,选出预测方差最大的 1,000 个分子,去重后对新样本执行 DFT + NBO 计算。由于该过程针对每一个目标进行,因此每轮选出的分子数为 20,000–30,000 个。
主动学习过程中的数据添加揭示了许多有趣的观察(见图 3):在第一次迭代中,模型倾向选择数据集中原本代表性不足的 含硼分子;后续迭代中,选出的分子中也包含了 溴、碘,以及 同时含碘和硫的分子;随着迭代推进,模型更多地选择了含 汞 和 砷 的分子;在最后一轮迭代中,模型集中特别挑选了大量具有 B–Cl 键对 的分子;
此外,本研究还分析了分子内环结构的相对位置(见图 3b),模型在最后一轮成功识别并选出了具有 金刚烷(adamantane)结构 的化合物。
这一过程展示了模型在化学空间中主动探索和精准识别特定分子子集的能力。
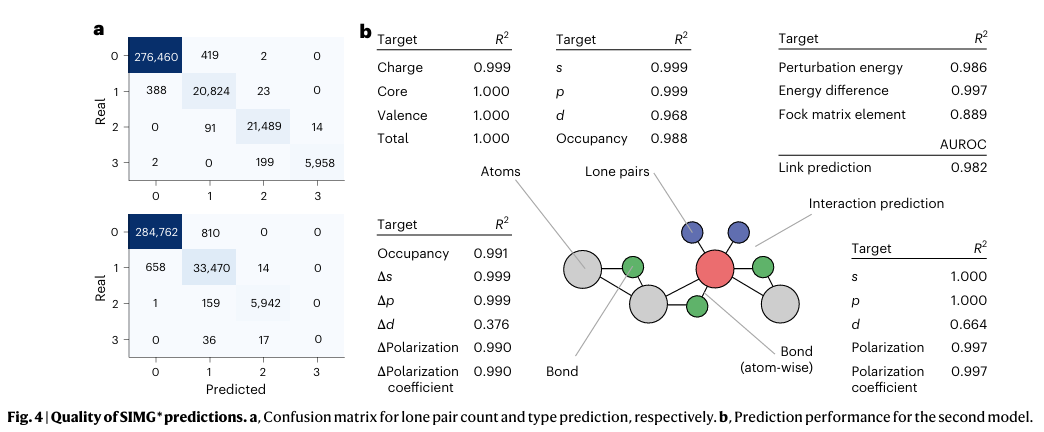
预测性能
图4展示了图神经网络(GNN)在近似预测SIMG*方面的卓越表现。模型在节点层级成功预测了孤对电子的数量与类型,混淆矩阵显示分类准确率较高,并在98%的案例中成功重构了真实的扩展分子图。在节点属性预测任务中,原子相关任务取得极高的R²分数,孤对电子的s和p轨道成分预测效果优秀,尽管d轨道预测稍逊,但考虑到相关样本稀少,其预测结果仍具参考价值。模型还准确预测了孤对电子的占据数和成键轨道的占据数。在成键轨道的预测任务中,杂化轨道特征、极化程度与极化系数等指标在多数原子上均取得优异结果,仅d轨道因样本不足略低。对于二阶轨道相互作用的分类预测,模型表现尤为出色,AUROC达到0.982,平均AUROC为0.979,反映出极强的分类能力,唯一表现略低的是Fock矩阵元素的数值回归,但整体仍维持较高水平。
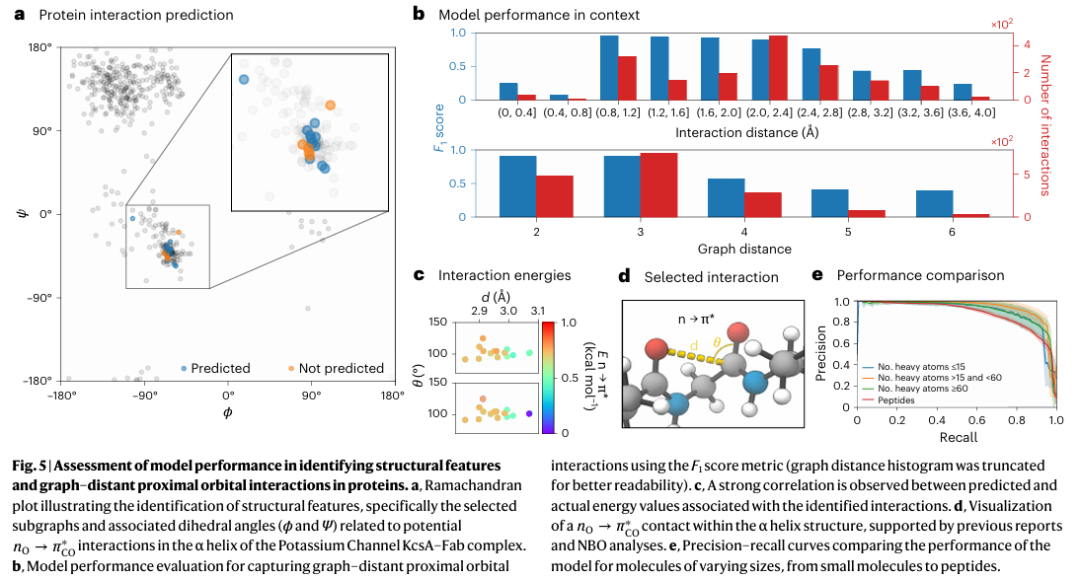
SIMG* 能够实现轨道相互作用的快速预测
由 NBO 数据推导并通过 SIMG* 表示和预测的立体电子相互作用,在大分子的稳定性中发挥着关键作用。值得注意的是,尽管蛋白质的空间结构主要依赖氢键来维持稳定,但各种较弱的轨道相互作用共同显著地贡献了总的稳定能。然而,即使是最新版的 NBO 程序也难以在含有 3000 个以上基函数的 DFT 模拟中进行分析,这使得其在大多数大分子中的应用变得不可行。但由于立体电子效应具有空间局部性,本研究提出可以使用在 GEOM 数据集中小分子的 NBO 数据上训练的 SIMG* 模型,准确预测更大结构中的轨道相互作用,从而实现对以往难以获取的化学信息的挖掘。
为了评估模型在识别大分子,特别是蛋白质中某些特定轨道相互作用的有效性,本研究选取了低 K⁺ 浓度下钾通道 KcsA–Fab 复合物(PDB ID 1K4D)的一个子集作为研究对象。本研究关注的特征是 α 螺旋中酰胺氧原子的富含 p 轨道的孤对电子(nO)与相邻羰基的反键 π 轨道(πCO)之间的轨道相互作用,如图 5d 所示。此前已有研究通过 NBO 分析在该蛋白质中发现了 nO→πCO 相互作用,本研究据此作为模型评估的依据。
在整个蛋白结构的背景下,本研究绘制了 Ramachandran 图(图 5a),并重点分析了可能存在 nO→π*CO 相互作用的子图相关的二面角。这些二面角定义为:ɸ(C′i–1–Ni–Cαi–C′i) 和 ψ(Ni–Cαi–C′i–Ni+1),其中 i 为氨基酸残基的索引。本研究的模型成功预测了大多数情况下的上述轨道相互作用,如图 5a 所示。此外,模型对二阶轨道相互作用的预测与真实值高度一致(图 5c)。
本研究还进一步评估了模型在捕捉和理解图上较远、但在三维空间中邻近的轨道相互作用方面的表现。为此,本研究对 SIMG* 所预测的相互作用进行了量化,并与 NBO 的真实计算结果进行了比较,使用 F1 分数进行评估(图 5b)。结果显示,模型性能与相互作用样本数量呈正相关。当原子间距离小于 2.8 Å,且键图距离小于 4 时,观测到显著增加的相互作用数量,这一趋势提升了模型在该范围内预测的准确性。值得注意的是,训练数据中空间距离大于 2.8 Å 且图距离大于 4 的相互作用实例较为稀少。
为了更广泛评估模型在大结构中的表现,本研究构建了一组额外的大分子(中性、闭壳层、截断肽,接近本研究中量子化学方法的计算极限),并对其进行 NBO 计算以获得 SIMG,再与预测得到的 SIMG* 进行比较。需要强调的是,这些结构的 DFT+NBO 计算通常需要数小时甚至数天,而对同一结构进行 SIMG* 预测仅需几秒。图 5e 显示模型成功捕捉了立体电子相互作用,在预测效果上与小分子无显著差异。此外,大分子与 GEOM 数据集中样本的主要区别仅在于键图距离,而非空间距离。本研究进一步使用 Dijkstra 算法对不同键图距离下的模型性能进行比较,并未观察到显著相关性。汇总指标也未表现出显著差异,验证了 SIMG* 模型预测的外推能力。
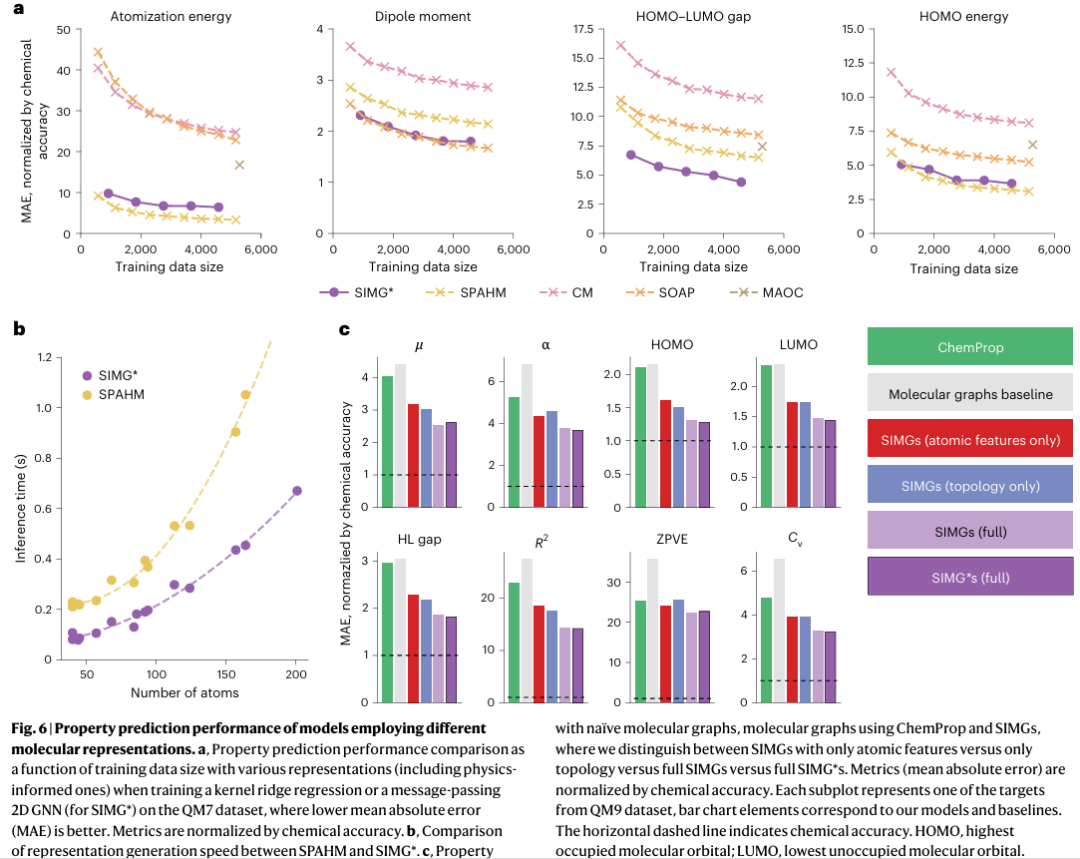
表示方法提升了下游模型的性能
最后,本研究评估了该表示方法在下游任务中的有效性。首先,本研究将 SIMG* 表示用于训练消息传递型二维 GNN 进行性质预测,并与其他物理与几何信息表示训练的核岭回归方法在 QM7 数据集上的表现进行比较(图 6a)。在预测 HOMO–LUMO 能隙方面,本研究的表示效果优于所有其他方法;在偶极矩预测方面与最佳表示(SOAP)持平;在 HOMO 能量和原子化能预测方面仅略逊于最佳表示(SPAHM)。需要指出的是,部分对比方法针对 QM7 任务进行了大量超参数调优,而本研究对 SIMG* 并未进行任何调参。
其次,本研究比较了表示生成速度,与另一种表现优异的物理信息方法 SPAHM 进行对比。如图 6b 所示,SIMG* 的生成速度在不同分子大小下均快于 SPAHM,且分子规模越大,性能差距越显著。
本研究还在更大规模的 QM9 数据集上进行额外比较分析,考察图结构增强的影响,设置了六种图结构(图 6c):(1)包含额外特征的分子图(如 ChemProp 所用,默认超参数);(2)标准分子图;(3)使用 SIMG 表示但保留原始图拓扑结构,仅增强原子特征;(4)使用 SIMG 拓扑但不引入新特征;(5)完整 SIMG;(6)使用本研究模型生成的 SIMG* 表示替代真实 SIMG。通过该比较分析,本研究区分了新增特征与图拓扑变化所带来的效应。
图 6c 显示,在统一的消息传递型 2D GNN 架构下,SIMG 表示无论是增强特征还是拓扑,均显著优于分子图和 ChemProp(除零点振动能以外)。完整的 SIMG 表示在诸如偶极矩预测等任务上获得更大的提升。而使用本研究模型生成的 SIMG* 表示,其性能几乎与真实 SIMG 相当,但无需在下游训练中进行昂贵的 DFT+NBO 计算。此外,在许多目标性质(如偶极矩、HOMO-LUMO 能隙)上,该表示方法能将模型预测结果更接近化学精度标准。
这些结果表明,本研究提出的表示方法在消息传递型 2D GNN 性质预测任务中显著提升了模型性能。然而,也必须指出,2D GNN 在本文任务中的整体表现仍明显不如采用等变架构的 3D GNN。本研究相信,SIMG* 可被直接用作 3D GNN 输入,以提升其在性质预测、原子间势能、分子构象生成和蛋白质折叠等任务中的表现,但这些应用超出了本研究的范围。
整理 | 刘名权
参考资料
Boiko, D.A., Reschützegger, T., Sanchez-Lengeling, B. et al. Advancing molecular machine learning representations with stereoelectronics-infused molecular graphs. Nat Mach Intell 7, 771–781 (2025).
https://doi.org/10.1038/s42256-025-01031-9
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢