DRUGAI
研究人员引入了一种基于transformer的神经网络,在未注释的串联质谱(MS/MS)上进行自监督预训练,从MassIVE GNPS数据库中提取的GNPS实验质谱(GeMS)数据集中挖掘出数百万个质谱图。该模型以预测被掩盖的质谱峰和色谱保留顺序为目标,进而学习分子结构的丰富表示,研究人员称之为DreaMS(Deep Representations Empowering the Annotation of Mass Spectra)。在多个任务中微调该神经网络后,DreaMS表现出优于现有方法的性能。研究人员还发布了DreaMS Atlas——一个由2.01亿个MS/MS谱图组成的分子网络。

小分子和代谢物的发现对药物开发、环境分析和疾病诊断等领域具有广泛影响。然而,人类目前已发现的天然小分子仅占存在于人体或植物王国中的极小一部分,大部分天然化学空间仍未被探索。LC–MS/MS 是研究生物和环境样品分子组成的重要工具,其中MS/MS谱图包含分子结构的关键信息。然而,在一次非靶向代谢组学实验中,能通过参考谱库注释的谱图不足2%,即使借助最新的机器学习方法,也仅能注释不到10%的谱图。
现有的质谱解析方法包括谱图相似度算法、正向注释方法和反向注释方法,但都严重依赖有限的谱图库。即使是最先进的工具如SIRIUS,也依赖复杂的组合优化、人工规则和多个模型,限制了其泛化能力和效率。为了解决这一限制,研究人员设计了一个拥有1.16亿参数的transformer网络,直接在原始、未注释的大规模谱图上进行训练,从而跳过了人工先验知识和昂贵的前处理步骤。
结果
构建大规模MS/MS数据集用于自监督学习
研究人员从MassIVE GNPS数据库中提取了约7亿个MS/MS谱图,并经过质量控制与聚类处理,形成了名为GeMS的高质量数据集。根据质量标准,数据被划分为GeMS-A、B和C子集。研究人员利用局部敏感哈希(LSH)对谱图进行聚类,生成多个用于训练的变体,并以紧凑的格式存储以供深度学习使用。
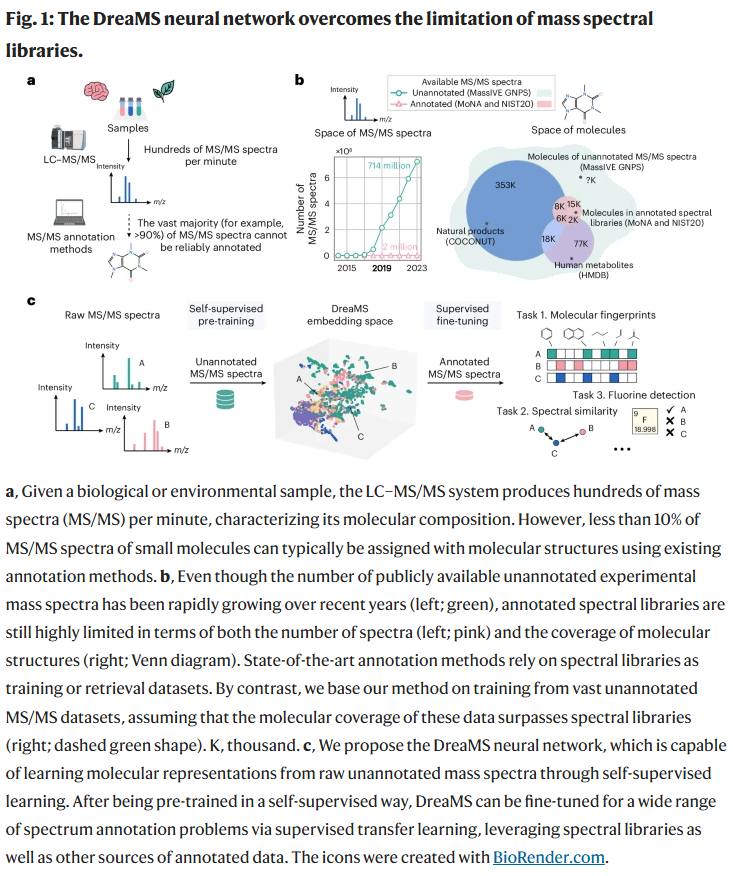
在MS/MS谱图上进行自监督预训练
基于GeMS-A10子集,研究人员构建了DreaMS模型,采用BERT风格的掩码预测策略进行训练。谱图被表示为m/z与强度的连续对,30%的m/z被随机掩盖,并要求模型重构这些值。此外,模型还被训练预测谱图的色谱保留顺序。结果显示,DreaMS学习到了结构相似性驱动的嵌入向量,并能识别代表分子片段的峰值。主成分分析显示,该模型能够在不同电离和碰撞条件下保持结构一致性。
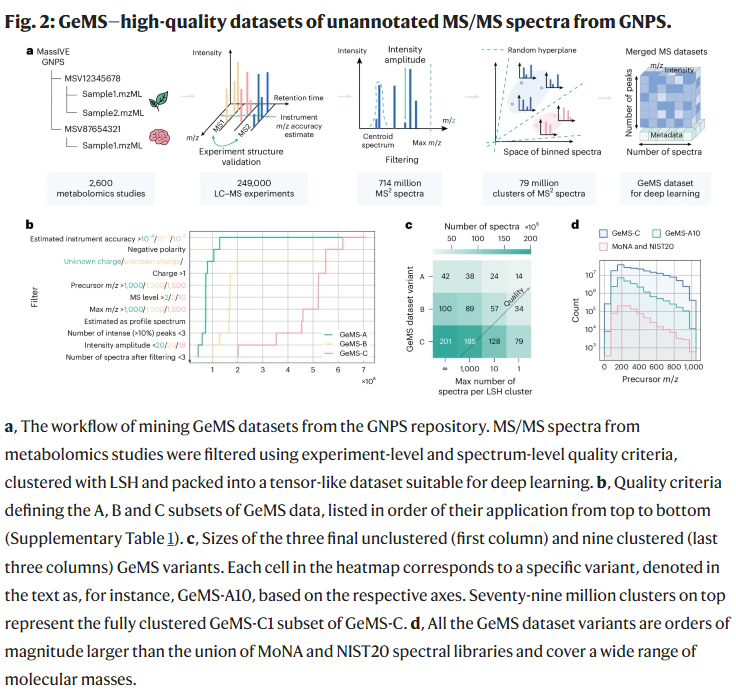
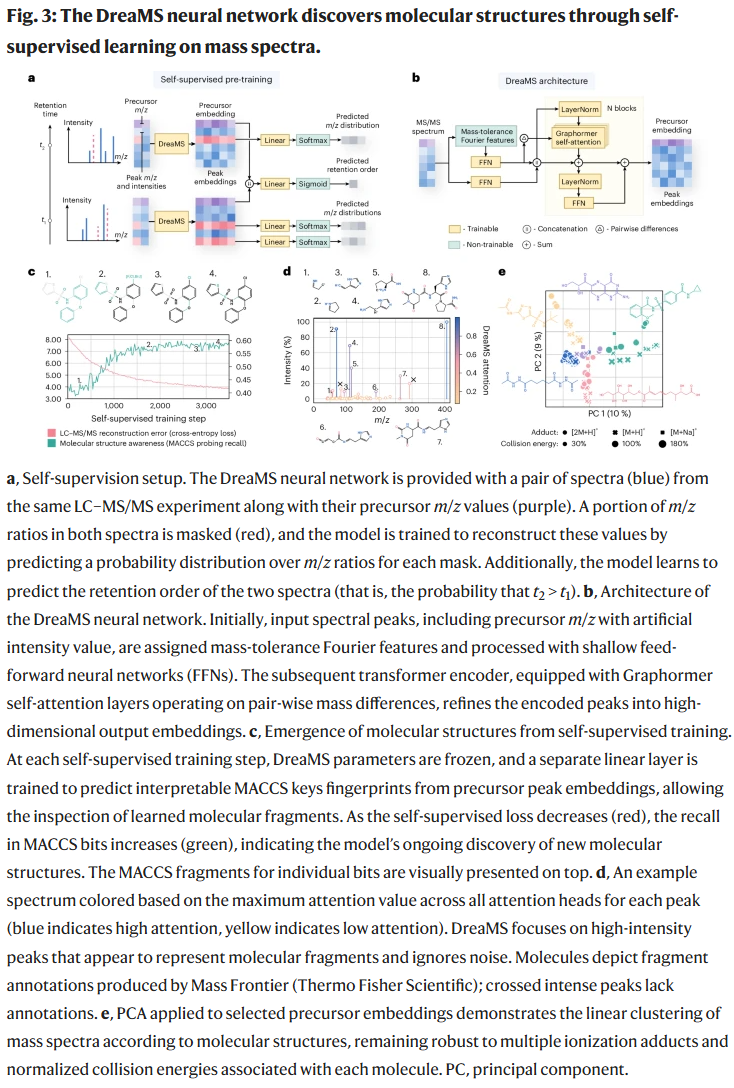
向多个注释任务迁移学习
在自监督训练基础上,研究人员将DreaMS迁移到多个注释任务,包括谱图相似度、分子指纹、化学性质预测和含氟分子识别。即使不经微调,仅计算DreaMS嵌入向量间的余弦相似度,也优于现有的监督模型(如MS2DeepScore)。通过精心设计的对比学习微调,模型进一步增强了对分子微小差异的辨识能力,在相似结构搜索、数据库检索中表现优异。
在指纹预测任务中,DreaMS可直接从原始谱图预测Morgan指纹,实现与MIST相当的检索性能。对于化学性质预测,DreaMS在Lipinski规则参数、Bertz复杂性、合成可行性、QED等多个指标上均优于基准方法。值得一提的是,在含氟分子的预测中,DreaMS达到了0.91的精度,远超SIRIUS,并且能够在无类似训练样本的条件下泛化至新分子。
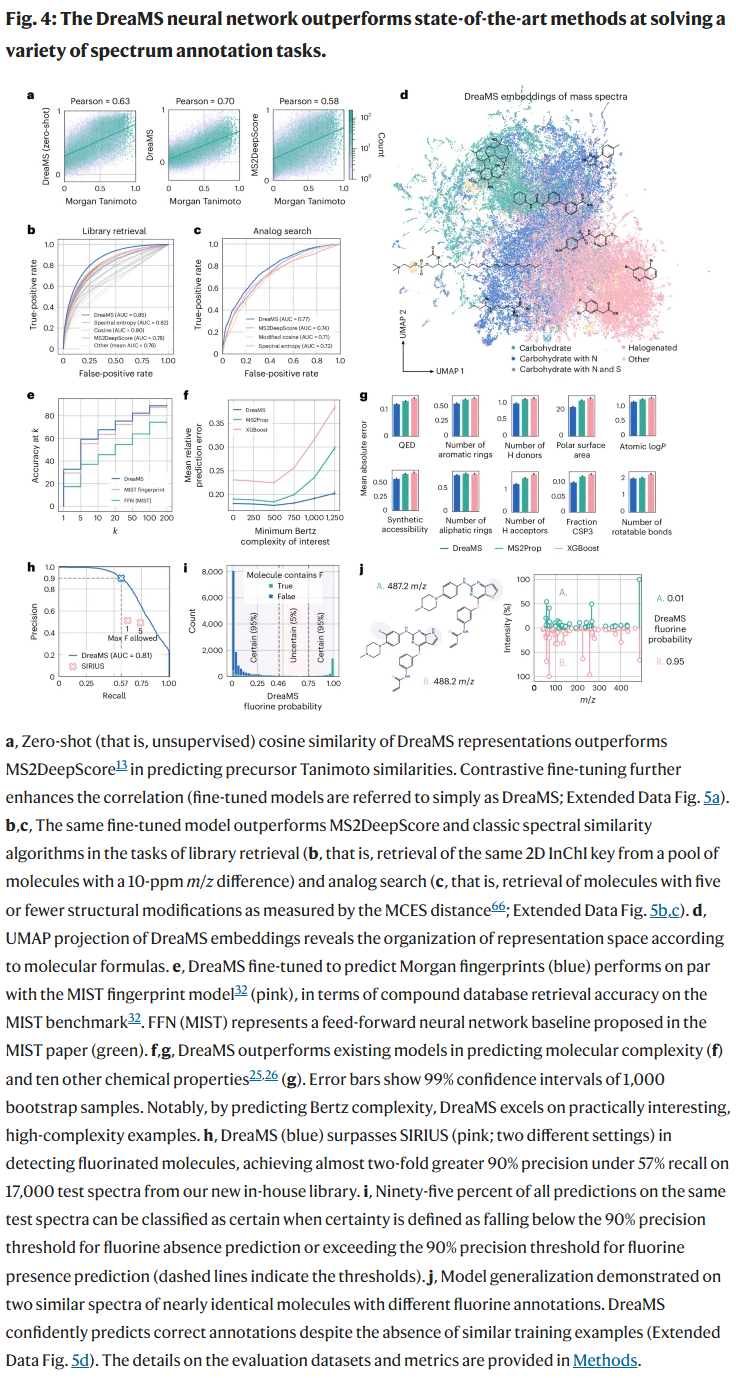
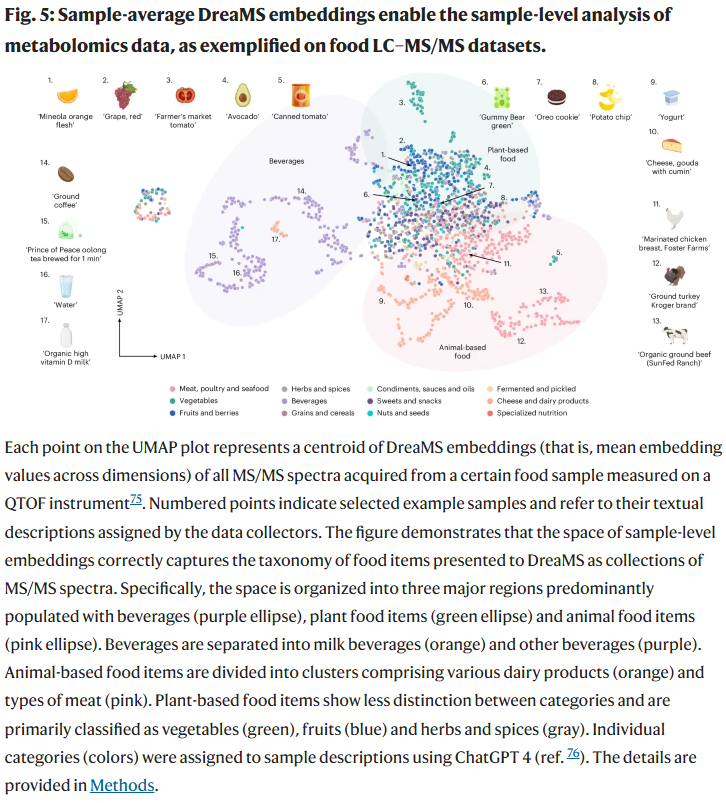
构建DreaMS Atlas
研究人员使用DreaMS对GeMS-C中的2.01亿个谱图进行嵌入,并构建了基于三近邻的图网络,命名为DreaMS Atlas。该图网络中,谱图以节点表示,谱图间的相似度以边表示,图结构紧密,99.7%的节点组成一个主连通分支,谱图库在图中分布均匀。进一步分析发现,Atlas中存在多个连接已知分子和未注释谱图的路径,展示出该图谱在分子结构推断中的巨大潜力。例如,一个与牛皮癣相关的谱图节点与农药谱图高度相似,提示可能的环境暴露机制。
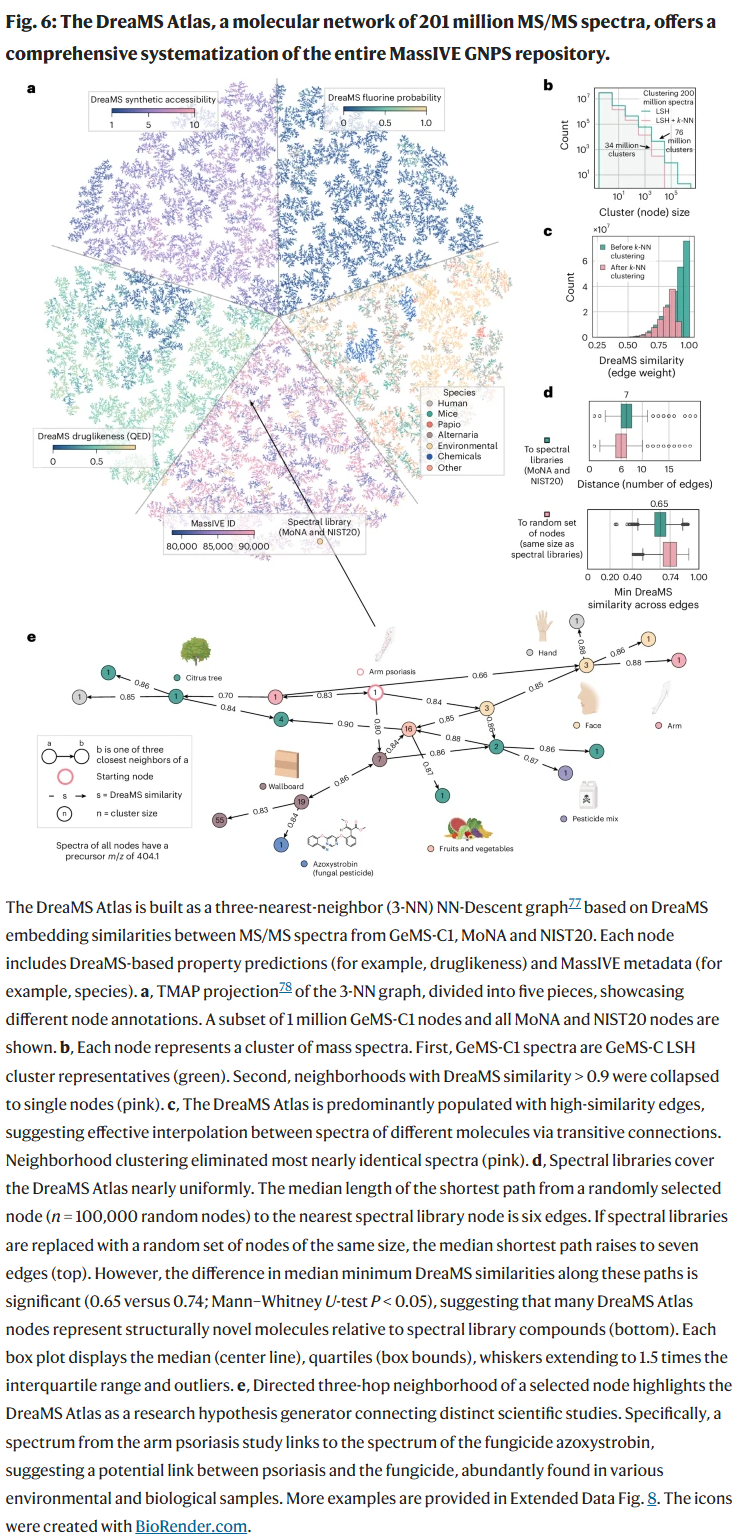
讨论
研究人员提出了DreaMS,一种针对MS/MS谱图构建的transformer模型。通过在大规模未注释谱图上进行自监督训练,DreaMS学会了反映分子结构的谱图表示,并在多项注释任务上达到了先进水平。DreaMS Atlas进一步提供了一个全面的谱图结构网络,可用于代谢组学研究中的结构推断、分子网络分析与药物发现等。
尽管取得了显著成果,研究人员指出DreaMS仍有拓展空间,包括扩展训练数据源至MetaboLights等数据库,纳入负离子模式和MS1谱图信息等。长期来看,DreaMS不仅为质谱分析提供了统一的基础模型,也为化学空间探索、片段药物设计等研究方向带来了全新可能。
整理 | WJM
参考资料
Bushuiev, R., Bushuiev, A., Samusevich, R. et al. Self-supervised learning of molecular representations from millions of tandem mass spectra using DreaMS. Nat Biotechnol (2025).
https://doi.org/10.1038/s41587-025-02663-3
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢