导语


内容简介
内容简介
在过去几十年中,蛋白质折叠问题一直是结构生物学和生物物理学的核心难题。随着深度学习方法,尤其是AlphaFold2的突破,蛋白质结构预测迎来了革命性的飞跃。然而,这些方法主要针对的是“正确折叠”的单体蛋白,对于那些在疾病发生中发挥关键作用、具有高度结构多样性(polymorphism)的淀粉样蛋白(amyloid fibrils)却仍力不从心。
近日,昌平实验室陈明辰团队在PNAS上发表最新研究论文,提出全新的 RibbonFold 方法,通过引入“丝带构象假说”并在深度学习模型中施加结构约束,首次系统性预测并重现了多种淀粉样蛋白的构象多态性,为理解神经退行性疾病等相关病症的分子机制提供了新的窗口。本次读书会将首先介绍蛋白质折叠与能量景观等基础概念,进一步讨论蛋白质错误折叠与淀粉样蛋白,之后介绍“丝带限制”用于预测淀粉样蛋白结构的最新进展,希望与大家一起探讨人工智能、统计物理、蛋白质折叠这一前沿交叉领域。
内容大纲
内容大纲
1. 蛋白质折叠与能量景观 Protein folding & energy landscapes;
2. 最小阻挫原理 Principle of minimal frustration;
3. 蛋白质错误折叠与淀粉样蛋白 Protein misfolding and amyloid;
4. “丝带限制”用于预测淀粉样蛋白结构 Ribbon constraints for predicting amyloid structures;
5. 淀粉样蛋白折叠景观 Amyloid folding landscapes;
核心概念
核心概念
蛋白质折叠的能量景观学说 Energy landscape theory for protein folding
最小阻挫原理 Principle of minimal frustration
蛋白质错误折叠和沉淀 Protein misfolding & Protein aggregation
淀粉样蛋白多态性 Amyloid polymorphism
神经退行 Neurodegeneration
淀粉样纤维 Amyloid fibrils
主讲人
主讲人

陈明辰,2014年在北京大学获得生物学学士学位,2019年在莱斯大学获得计算生物物理博士学位,导师是美国理论化学家和物理学家 Peter Wolynes。2018-2024年期间在北京星亢原生物科技有限公司担任联合创始人& 副总裁。2024年至今任昌平实验室领衔科学家。专注于人工智能/计算驱动的生物分子动态、蛋白质折叠、蛋白质相互作用以及药物设计。重点研究人工智能驱动的抗体分子等生物大分子的设计和成药性优化。
近年主要研究成果包括:1)在蛋白动力学和蛋白相互作用上,在PNAS,Nat Comm,JACS等期刊上以第一作者/共同第一作者发表论文十多篇;2)参与设计的CD24抗体是中国首个人工智能设计优化的单抗拿到了美国FDA的IND批准;设计多个大小分子药物进入到PCC阶段;多个计算设计的候选药物达成总额上亿人民币的授权转让;3)2018年带队参加结构预测大赛 CASP13,获得 free modeling 领域第六名。
参考文献
参考文献
[1] Schafer, Nicholas P., et al. "Learning to fold proteins using energy landscape theory." Israel journal of chemistry 54.8‐9 (2014): 1311-1337.
[2] Chen, Mingchen, et al. "Surveying biomolecular frustration at atomic resolution." Nature communications 11.1 (2020): 5944.
[3] Chen, Mingchen, Nicholas P. Schafer, and Peter G. Wolynes. "Surveying the energy landscapes of Aβ fibril polymorphism." The Journal of Physical Chemistry B 122.49 (2018): 11414-11430.
[4] Eisenberg, David S., and Michael R. Sawaya. "Structural studies of amyloid proteins at the molecular level." Annual review of biochemistry 86.1 (2017): 69-95.
[5] Guo, Liangyue, et al. "Generating the polymorph landscapes of amyloid fibrils using AI: RibbonFold." Proceedings of the National Academy of Sciences 122.16 (2025): e2501321122.
[6] Ferreiro, Diego U., Elizabeth A. Komives, and Peter G. Wolynes. "Frustration in biomolecules." Quarterly reviews of biophysics 47.4 (2014): 285-363.
相关文章:AI+Science新视野:用物理信息引导AlphaFold 2预测蛋白质动力学
参与方式
参与方式
时间:2025年6月5日(周四)晚20:00-22:00

斑图链接:https://pattern.swarma.org/mobile/study_group_issue/917?from=wechat
PNAS 前沿:RibbonFold 揭示淀粉样蛋白多态结构的奥秘

论文题目:Generating the Polymorph Landscapes of Amyloid Fibrils Using Artificial Intelligence: RibbonFold
论文地址:https://www.pnas.org/doi/10.1073/pnas.2501321122
来源:昌平实验室
在过去几十年中,蛋白质折叠问题一直是结构生物学和生物物理学的核心难题。随着深度学习方法,尤其是AlphaFold2的突破,蛋白质结构预测迎来了革命性的飞跃。然而,这些方法主要针对的是“正确折叠”的单体蛋白,对于那些在疾病发生中发挥关键作用、具有高度结构多样性的淀粉样蛋白(amyloid fibrils)却仍力不从心。昌平实验室陈明辰团队在PNAS上发表了题为“Generating the Polymorph Landscapes of Amyloid Fibrils Using Artificial Intelligence: RibbonFold”的研究论文。该研究提出了全新的RibbonFold方法,通过引入“丝带构象假说”并在深度学习模型中施加结构约束,首次系统性预测并重现了多种淀粉样蛋白的构象多态性,为理解神经退行性疾病等相关病症的分子机制提供了新的窗口。
1. 进化的“漏斗”之外:
淀粉样蛋白的错误折叠之路
1. 进化的“漏斗”之外:
淀粉样蛋白的错误折叠之路
众所周知,进化倾向于将天然蛋白的能量景观雕塑为“漏斗”状,确保它们能迅速而准确地折叠至功能性结构。然而,并非所有蛋白都遵循这一法则。越来越多的研究揭示,一类被称为“本征无序(intrinsically disordered protein)”或“模糊(Fuzzy)”的蛋白质,具有高度多样的构象,甚至在无明确结构的情况下完成生物功能。而更极端的是,一些蛋白在特定条件下——比如高浓度、pH变化、突变或金属离子存在时——会自发形成淀粉样纤维。淀粉样纤维的结构特征不仅限于其有序性,更令人惊讶的是其“多态性”(polymorphism):同一条蛋白质序列可以在不同条件下组装成多种三维构型,这种多样性不仅与疾病的发生密切相关,甚至可能决定不同病人的疾病类型和进展速度。例如,Aβ蛋白在阿尔茨海默病中的不同聚集体形式与病理分型相关,α-Synuclein的不同构象则对应于帕金森病与多系统萎缩等不同神经系统疾病。
2. RibbonFold方法:
结构生物学与AI的深度融合
2. RibbonFold方法:
结构生物学与AI的深度融合
面对这些挑战,团队开发了RibbonFold方法,它不仅继承了AlphaFold2在序列到结构预测方面的强大能力,更通过“结构约束注入”策略专为预测淀粉样蛋白结构而设计。
RibbonFold的核心创新在于引入“丝带限制”(Ribbon constraints)——假设在几乎所有淀粉样纤维中,单体蛋白是以平行对齐的β-片层形式重复堆叠。这一假设来源于实验观察,是淀粉样纤维的普遍结构特征。RibbonFold通过将这一结构特征以模板形式硬编码进AlphaFold2的网络框架,从而将搜索空间从全三维折叠简化为二维“丝带折叠”,极大地提高了预测效率和准确性。RibbonFold还引入了训练多样性损失函数,使得模型能学习到尽可能多样的构象而非单一结构,从而探索构象多态性景观。
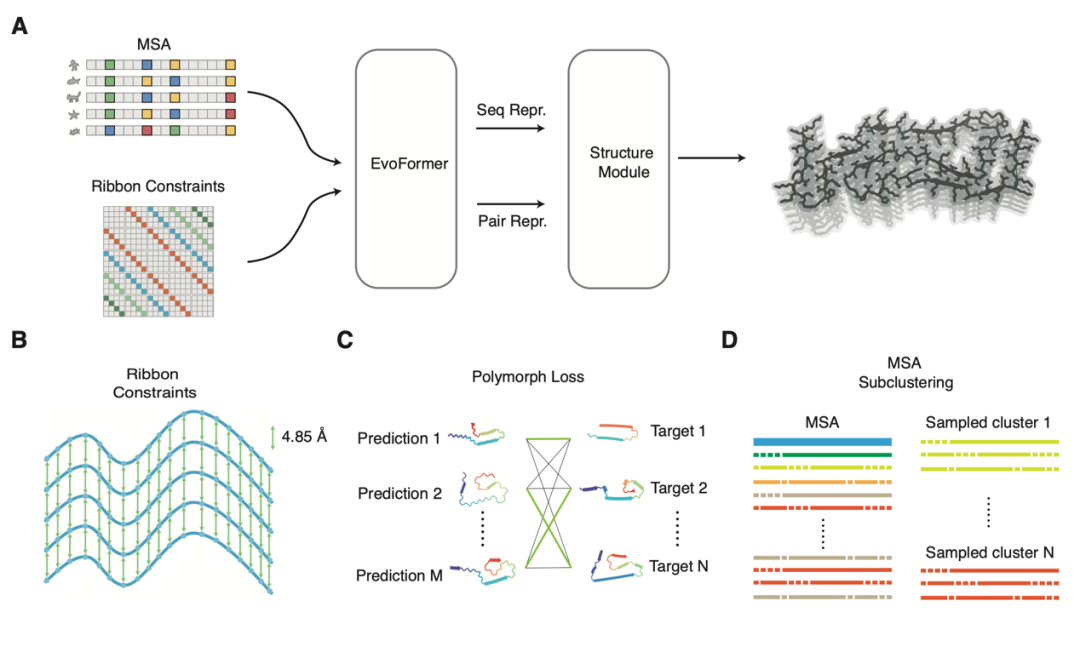
图1. RibbonFold模型框架&训练策略
3. 与实验多态结构吻合,重现多态选择性
3. 与实验多态结构吻合,重现多态选择性
RibbonFold在多个经典淀粉样蛋白上的预测,展示了它不仅能准确预测出已知的多态构象,而且构象之间的相对稳定性排序(基于物理能量打分)也与实验结果一致。例如,在Aβ和Tau蛋白中,RibbonFold能区分出在不同溶解度和生理条件下形成的不同纤维类型,揭示多态结构的形成可能与环境选择压力有关。
更重要的是,团队利用RibbonFold对随机打乱的序列进行了系统性测试,发现尽管它们也能形成某种聚集构象,但这些构象在能量上显著不稳定且分布广泛,缺乏自然序列中观察到的“聚集倾向性”和“有限多样性”。这一发现提示,那更稳定、更不容易被溶解的结构最终会占据主导地位,从而也导致了淀粉样蛋白有限的多态性。
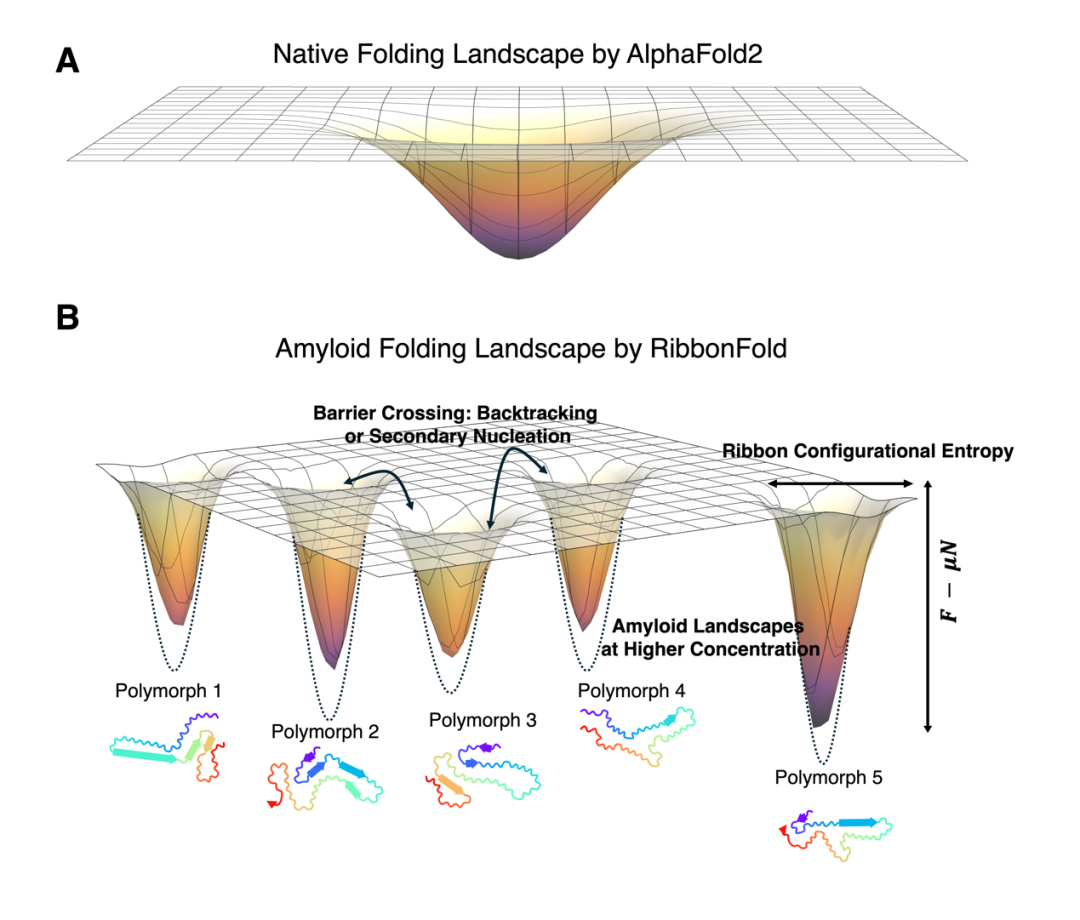
图2.RibbonFold结构预测重现淀粉样蛋白多态选择性
4. 未来展望:从结构预测到功能发现
4. 未来展望:从结构预测到功能发现
RibbonFold不仅仅是一个预测工具,它疾病机制探索和药物发现开启了多种新的可能性。
首先,它为揭示淀粉样疾病的分子机制提供了新手段。多态构象之间的转换可能是疾病进展中的关键步骤,RibbonFold提供了预测这些构象变换的可能通路。其次,它有潜力用于寻找药物作用位点。传统药物设计侧重于稳定天然折叠态,而RibbonFold提供了探索淀粉样蛋白构象空间的能力,可能帮助识别在某些多态结构中暴露但在其他多态结构中隐藏的“构象特异”位点,从而实现高特异性的靶向干预。第三,近期研究显示某些淀粉样蛋白具有催化功能,甚至在神经突触中参与记忆形成。RibbonFold能预测功能性淀粉样构象,有望推动功能性“类酶”材料设计和合成生物学的发展。最后,RibbonFold为将深度学习与物理知识结合提供了典范。通过引入结构约束、合理简化搜索空间,并关注多样性而非唯一结构,RibbonFold的理念也可推广至其他大尺度蛋白复合体的预测,甚至扩展至液-液相分离等动态聚集过程的建模中。
昌平实验室工程师郭良越,助理研究员余其林为共同第一作者;助理工程师王迪和吴小雨为本工作做出重要贡献;莱斯大学Peter Wolynes和昌平实验室领衔科学家陈明辰为该工作的共同通讯作者。
生命复杂性读书会:
生命复杂系统的构成原理
在生物学中心法则的起点,基因作为生命复杂系统的遗传信息载体,在生命周期内稳定存在;而位于中心法则末端的蛋白质,其组织构成和时空变化的复杂性呈指数式增长。随着分子生物学数十年来的突飞猛进,尤其是生命组学(基因组学、转录组学、蛋白质组学和代谢组学等的集合)等领域的日新月异,当代生命科学临近爆发的边缘。如此海量的数据如何帮助我们揭示宇宙中最复杂的物质系统——“人体”的构成原理和设计原理?阐释人类发育、衰老和重大疾病的发生机制?
集智俱乐部联合西湖大学理学院及交叉科学中心讲席教授汤雷翰,国家蛋白质科学中心(北京)副研究员常乘、李杨,香港浸会大学助理教授唐乾元,北京大学前沿交叉学科研究院研究员林一瀚,中国科学院分子细胞科学卓越创新中心博士后唐诗婕,共同发起「生命复杂性:生命复杂系统的构成原理」读书会,从微观细胞尺度、介观组织器官尺度到宏观人体尺度,梳理生命科学领域中的重要问题及重要数据,由生物学家提问,希望促进统计物理、机器学习方法研究者和生命科学研究者之间的深度交流,建立跨学科合作关系,激发新的研究思路和合作项目。读书会目前共进行10期,现在报名参与读书会可以加入读书会社群,观看视频回放,解锁完整读书会权限。

非平衡统计物理读书会
2024年诺贝尔物理学奖授予人工神经网络,这是一场统计物理引发的机器学习革命。统计物理学不仅能解释热学现象,还能帮助我们理解从微观粒子到宏观宇宙的各个层级如何联系起来,复杂现象如何涌现。它通过研究大量粒子的集体行为,成功地将微观世界的随机性与宏观世界的确定性联系起来,为我们理解自然界提供了强大的工具,也为机器学习和人工智能领域的发展提供了重要推动力。
为了深入探索统计物理前沿进展,集智俱乐部联合西湖大学理学院及交叉科学中心讲席教授汤雷翰、纽约州立大学石溪分校化学和物理学系教授汪劲、德累斯顿系统生物学中心博士后研究员梁师翎、香港浸会大学物理系助理教授唐乾元,以及多位国内外知名学者共同发起「非平衡统计物理」读书会。读书会旨在探讨统计物理学的最新理论突破,统计物理在复杂系统和生命科学中的应用,以及与机器学习等前沿领域的交叉研究。读书会已完结,现在报名可加入社群并解锁回放视频权限。
详情请见:从热力学、生命到人工智能的统计物理之路:非平衡统计物理读书会启动!
点击“阅读原文”,报名读书会
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢