DRUGAI
今天为大家介绍的是来自耶鲁大学生物统计学系的赵宏宇教授团队的一篇论文。药物协同效应的预测在癌症等复杂疾病的治疗中具有重要意义,但这一任务仍存在诸多挑战。本文提出了一个名为BAITSAO的统一模型,旨在通过标准化流程处理不同数据集中的协同效应预测任务。研究团队首先基于大语言模型(Large Language Models)生成的上下文嵌入信息,构建了药物和细胞系的初始表示,并据此形成训练数据集。在验证这些嵌入表示具有实际价值后,研究者采用多任务学习(multi-task learning)框架对BAITSAO进行大规模预训练,训练任务经过精心筛选。通过一系列基准测试,BAITSAO在模型结构和预训练策略上均表现出优于现有方法的性能。此外,研究还探讨了模型对输入变化的敏感性,并展示了其在药物发现、药物与基因相互作用预测以及多药组合效果预测等方面的广阔应用潜力。

联合用药已成为治疗HIV和癌症等复杂疾病的重要手段,其中关键在于实现药物的协同效应——即联合使用多种药物所产生的治疗效果超过它们单独使用的总和。这种策略不仅能增强疗效,还能降低单药所需剂量,减少耐药性。传统上,研究人员依赖高通量筛选实验来寻找有效的药物组合,但面对成千上万的可能组合,实验成本和时间代价高昂,特别是涉及三种或更多药物时更为困难。因此,发展高效的计算预测方法,结合大规模实验数据和生物学先验知识,成为提升协同效应识别效率的关键。
针对这一需求,许多基于深度学习的算法相继提出,如DeepSynergy、TreeComb、MatchMarker等。这些方法主要分为两类:一类直接预测协同得分,另一类则将连续得分转换为二元分类任务。然而,大部分方法未能充分利用公开数据库中的丰富信息,且在处理药物同分异构体或不完整数据集时泛化能力有限。此外,随着数据库持续更新,如何追踪和管理训练数据版本也成为影响模型可信度的重要因素。
近年来,大语言模型(Large Language Models, LLM)作为基础模型的代表,已在自然语言处理领域取得显著突破。部分研究尝试将药物协同预测问题转化为问答任务,通过LLM获取药物和细胞系的先验知识来预测未知组合的协同效果。虽然这种方法在一定程度上提高了可解释性,但依赖预设阈值的分类方式可能引入偏差,而且所使用的模型未开源,不利于学术界的进一步验证和拓展。
为此,研究团队提出了BAITSAO,一个统一且可扩展的药物协同预测模型。该模型充分利用LLM提供的上下文嵌入信息,作为药物与细胞系的输入表示,在大规模协同效应数据库上通过多任务学习(Multi-Task Learning)进行预训练,兼顾分类与回归任务。更重要的是,BAITSAO支持零样本学习与模型微调,能够对从未见过的药物组合进行有效预测,同时支持多药组合分析及元信息的引入,展现出强大的扩展性和应用潜力。
BAITSAO总览
BAITSAO模型有两项关键创新。首先,研究团队构建了一套统一的数据处理流程,将药物和细胞系的信息转换为适用于机器学习的表格数据形式,并据此生成可用于多种任务的训练数据集。实验证明,这些由大语言模型生成的嵌入表示中包含了有助于模型预测的生物功能信息。其次,BAITSAO在多任务学习框架下,对不同类型的协同效应预测任务进行统一建模和训练,通过系统实验验证了其模型架构和预训练策略在准确性和泛化能力上的显著优势。
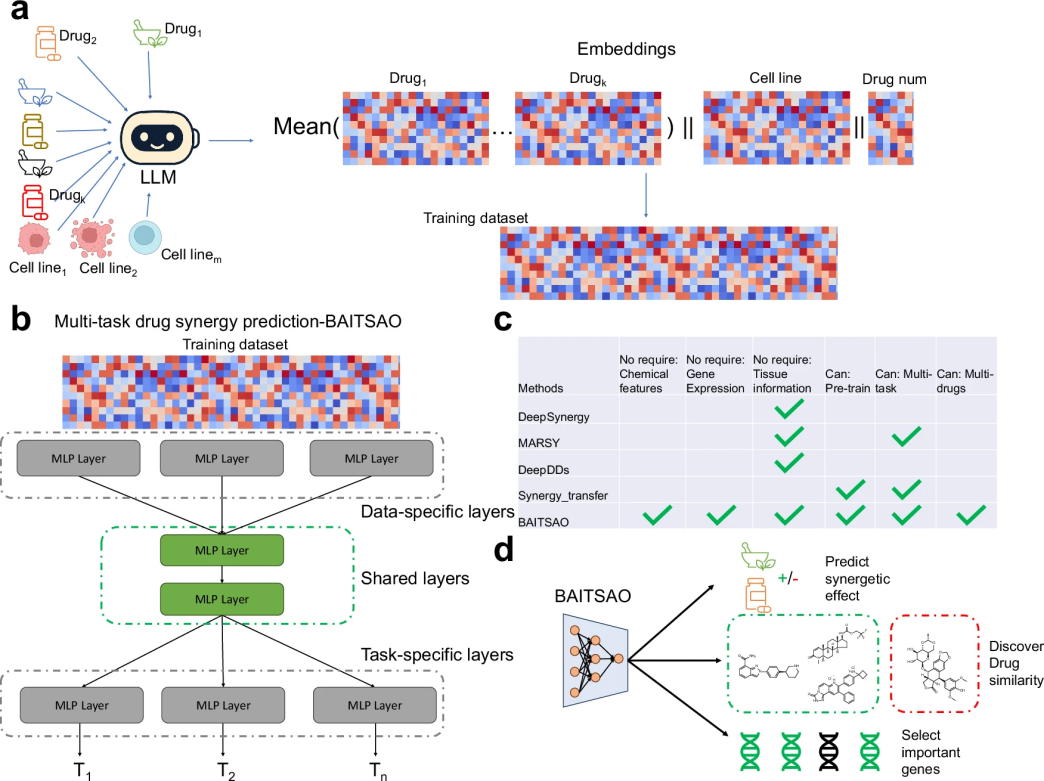
图 1
此外,BAITSAO支持灵活迁移,能够广泛应用于各类药物协同相关的下游分析任务。图1a 和1b展示了 BAITSAO的整体架构与运行流程,图1c则对比总结了BAITSAO与其他药物协同预测方法之间的主要差异,而图1d概览了BAITSAO支持的核心功能,包括任务扩展、多药组合分析等实际应用场景。
来自LLM的药物嵌入反映了功能相似性与细胞级反应
本研究利用大语言模型(LLM)生成药物与细胞系的嵌入信息,揭示其在功能识别与细胞响应预测中的潜力。研究团队基于GPT-3.5提取药物和细胞系描述的嵌入向量,并验证其在表达功能信息上的有效性。与GPT-4和Claude 3.5相比,GPT-3.5嵌入在效率与准确性之间取得平衡,且显著优于Gemini。UMAP可视化和相似度分析显示,同一药物在不同描述下的嵌入相似度远高于不同药物之间,说明其具备稳定性与辨别力。
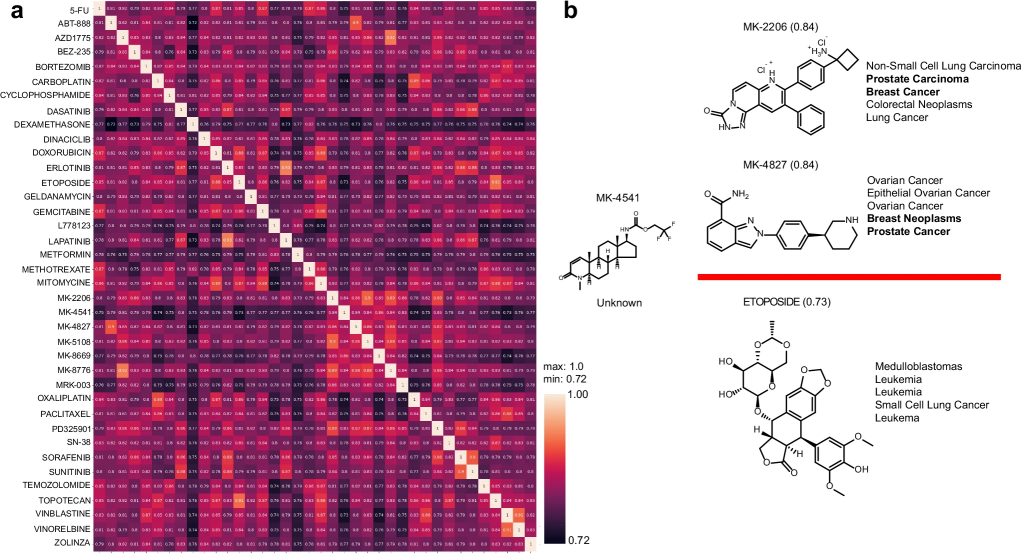
图 2
进一步比较GPT-3.5嵌入与DrugBank的原始描述,发现三类说明(指征、摘要、背景)的相似度均高于0.87,皮尔森相关系数也显著偏高,验证了嵌入的功能保真性。图2b展示了如何通过嵌入推测未知药物(如MK-4541)的功能。
此外,研究将该嵌入用于单细胞转录组(scRNA-seq)中药物扰动的基因表达预测。与传统基于SMILES的方法相比,GPT-3.5嵌入及其与SMILES的联合使用显著提升了预测性能(p < 0.05)。结果表明,LLM嵌入不仅能表征药物功能,还可连接药物与细胞响应,有助于构建更强的药物协同效应预测模型。
通过无需预训练的评估来展示强大的嵌入和架构
为了验证大语言模型(LLM)嵌入表示与BAITSAO架构本身的有效性,研究团队分别在药物协同预测的回归与分类任务中进行了无预训练评估。结果表明,GPT-3.5默认生成的药物嵌入无需依赖额外的提示工程(如MetaPrompt或Chain-of-Thought),已具备充分功能信息。此外,在使用相同嵌入作为输入的条件下,BAITSAO架构相较DeepSynergy表现更优(p = 0.002),说明模型结构优化对性能提升具有显著贡献。
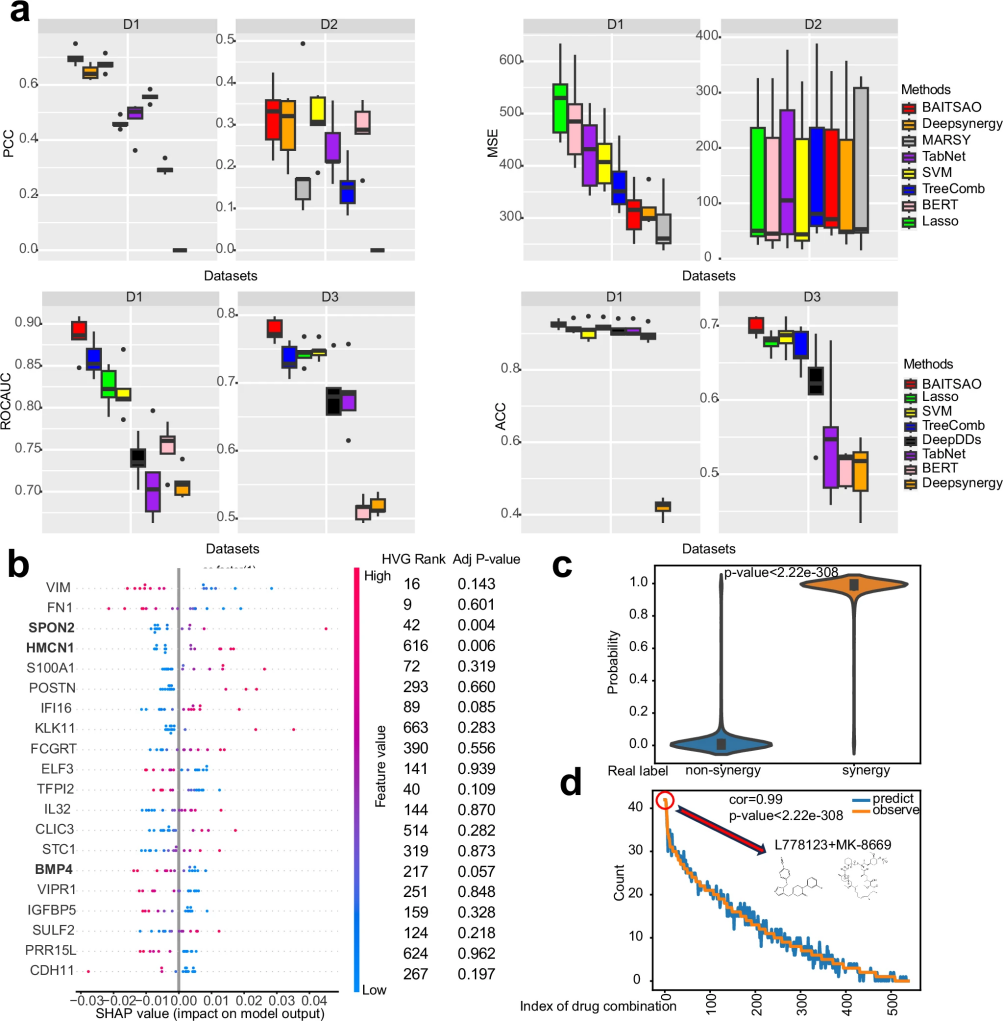
图 3
在回归任务中,团队选取D1和D2数据集,分别用皮尔森相关系数(PCC)与均方误差(MSE)作为评估指标;在分类任务中,评估D1和D3数据集,使用ROCAUC和准确率(ACC)指标。图3显示,BAITSAO在四项评估中有三项排名第一,且在深度学习方法中表现最为稳定。相比之下,BERT方法整体表现不如BAITSAO,验证了使用嵌入向量作为输入的优势。实验结果表明,将高质量嵌入与合理架构结合,能够构建出性能更优、泛化能力更强的药物协同预测框架。
通过多模态学习,展现BAITSAO对药物基因相互作用和药物细胞系相互作用的可解释性
本研究展示了BAITSAO如何通过整合基因表达数据与SHAP解释方法,实现药物与基因、药物与细胞系之间交互机制的可解释性分析。在D3数据集中,作者将细胞系的基因表达信息作为额外模态输入,并通过SHAP评估不同基因对药物协同预测的贡献。以DEXAMETHASONE–DINACICLIB药物组合为例,分析结果显示VIM基因在多个细胞系中具有最高重要性,且该基因已知与多种癌症相关。
进一步分析发现,部分高SHAP值基因(如BMP4)在协同与非协同样本间呈显著表达差异(p = 0.0062),并验证为药物靶点。同时,这些基因在表达水平和方差上表现出明显异质性,说明其反映了细胞对药物响应的多样性。GO和MsigDB富集分析亦表明,这些基因涉及多个癌症相关通路。
在药物组合层面,图3c显示BAITSAO可区分不同细胞系中药物组合的协同潜力(p < 2.22e-308)。图3d进一步证实预测标签数量与真实标签高度相关,表明模型具备准确识别高协同覆盖药物组合的能力。例如,L778123–MK-8669被预测在最多细胞系中具有协同效应,展现了BAITSAO在药物筛选中的应用潜力。
预训练数据的统计特征
研究团队使用DrugComb数据库构建了BAITSAO的预训练数据集。DrugComb是当前规模最大的药物协同数据库,包含多个细胞系与药物组合的协同效应信息。经清洗后,数据集中共收录超过70万条药物-细胞系组合。图4a和图4b分别显示这些组合及细胞系在不同组织类型中的分布,以皮肤、淋巴和肺组织最为常见,代表了多种重要的生理组织。
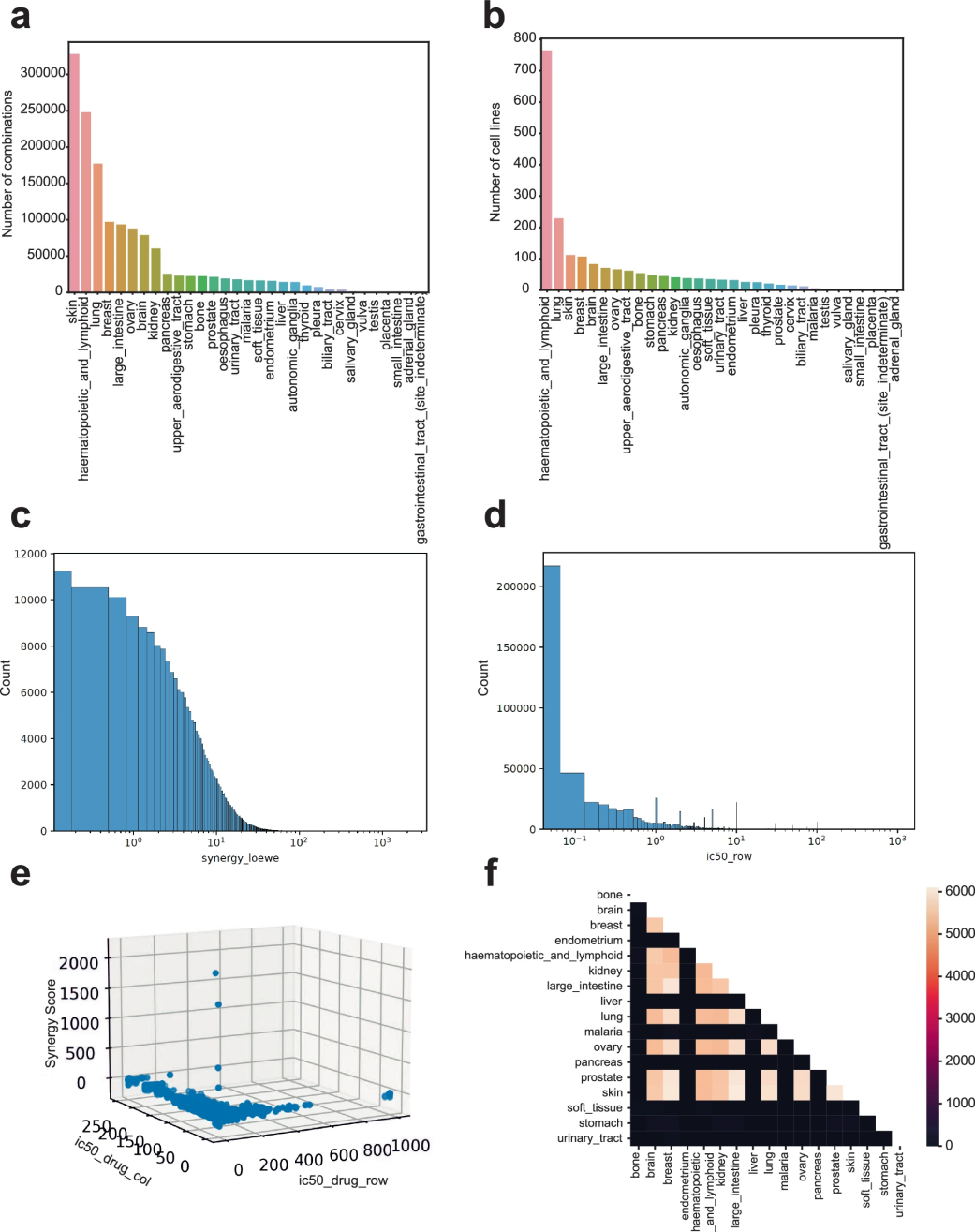
图 4
图4c展示协同评分的分布,整体偏向低分,说明协同效应具有稀疏性。图4d显示药物的IC₅₀分布,也呈非对称形态。图4e揭示IC₅₀与协同评分之间存在非线性关系,进一步验证了采用神经网络等非线性模型的合理性。图4f分析了不同组织间组合的重叠情况,显示组织来源较为多样,有助于模型在多组织背景下泛化。
此外,研究对药物和细胞系的嵌入向量进行了Leiden聚类分析,同一聚类内对象具有功能相似性,进一步验证嵌入在生物学属性上的表示能力。由于DrugComb发布于2021年6月,而GPT-3.5的训练数据截至同年9月,因此可以合理推断本研究使用的数据未出现在GPT-3.5训练集中,确保了模型分析的独立性与可靠性。
预训练的BAITSAO有助于多任务条件下的药物协同效应预测
研究团队进一步优化了BAITSAO的结构,并在多任务学习框架下进行大规模预训练,显著提升模型在药物协同预测方面的表现。模型设计包括四项任务:协同效应的回归与分类预测,以及两种单药抑制效应的回归预测。最终选定其中三项用于训练,并采用不确定性加权策略平衡各任务损失,提升稳定性。图5a所示的Help-Harm矩阵帮助判断各任务之间的协同关系,发现分类任务能提升其他任务表现,而某些冗余信息(如单药 RI_col)反而削弱分类效果,因此在训练中被剔除。
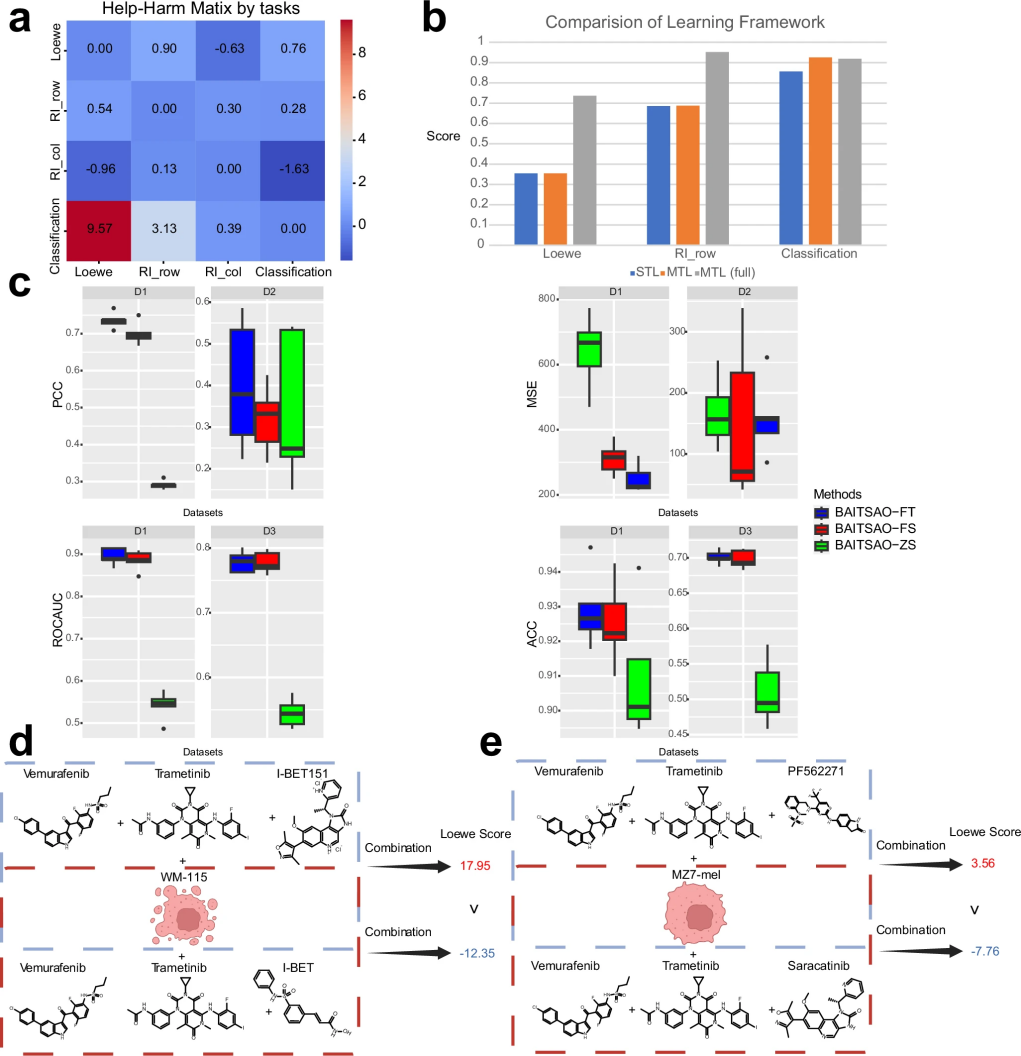
图 5
通过图5b与图5c的对比实验证明,多任务预训练下的BAITSAO(BAITSAO-FT)在多个数据集上均优于零样本版本(BAITSAO-ZS)。即使在预训练组合未包含的测试样本上,BAITSAO-ZS依然表现出超过随机水平的分类能力(ACC > 0.5),显示良好的泛化性与效率。
此外,研究还展示了BAITSAO在预测三药组合中的应用潜力(图5d, e)。利用Monte Carlo Dropout方法生成置信区间,评估不同第三药物对协同效应的影响。例如,I-BET151相比I-BET显著提高协同得分,PF562271相比Saracatinib也具有更强的抑制效果。这些预测与实验结果高度一致,表明BAITSAO可为复杂药物组合优化和临床设计提供科学支持。
灵敏度分析
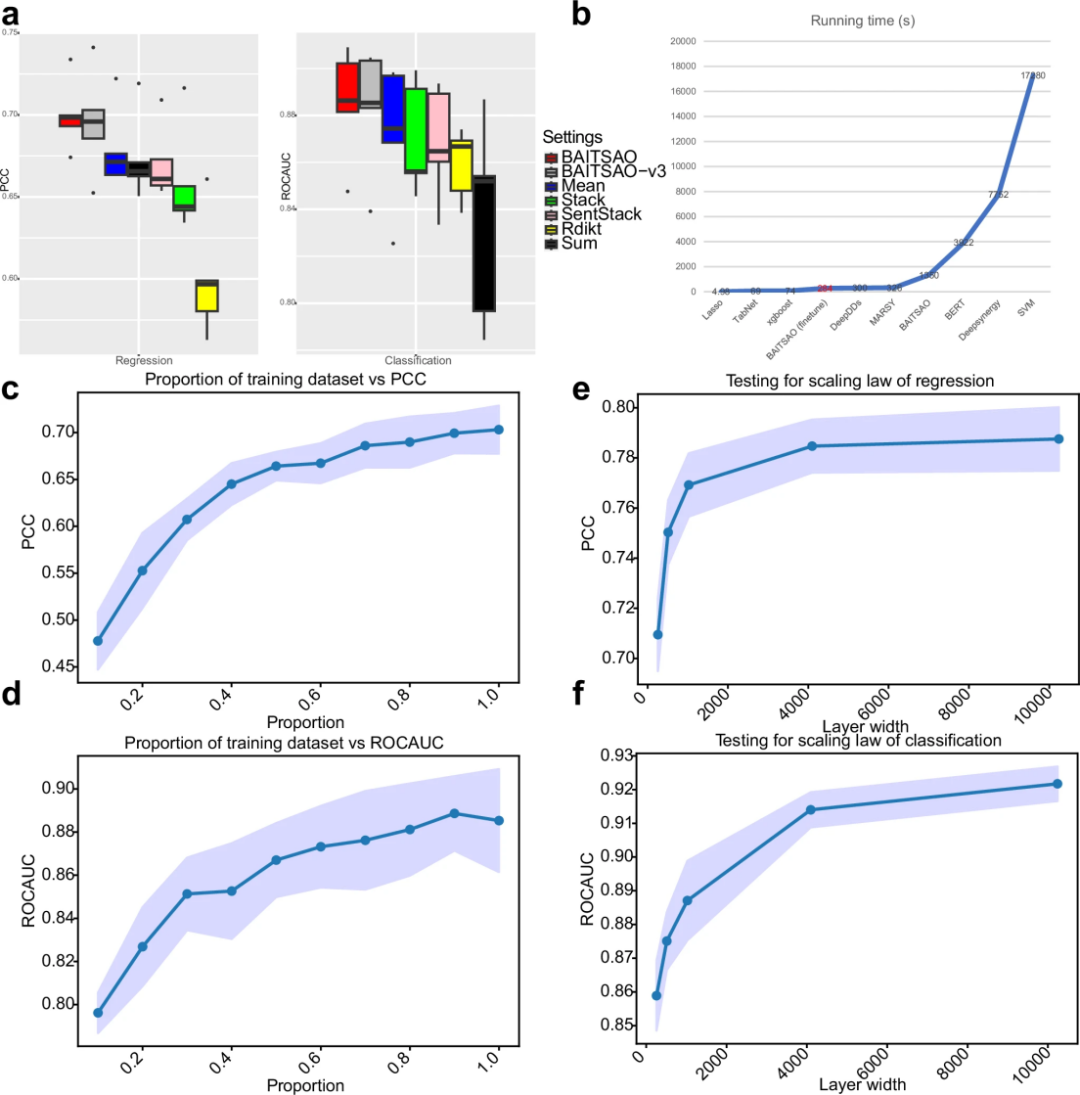
图 6
研究通过系统性敏感性分析,深入评估了BAITSAO模型在不同设置下的性能变化与资源适应性。首先,图6a显示,在多种药物与细胞系嵌入组合策略中,将药物嵌入取平均并与细胞嵌入按行堆叠,可在所有任务中取得最优表现,兼顾效率与效果。图6b表明,无需预训练的BAITSAO比传统方法如DeepSynergy和SVM更高效,而经预训练并微调后的模型运行速度更快,且全流程可在单个GPU上完成,具备良好部署潜力。
在多任务策略方面,辅助图14和15的消融实验显示,BAITSAO采用的修正版不确定性加权(UW)在分类任务中表现最稳健,相较其他梯度匹配方法更具优势。最终选择每个任务配置一个专属层,达到最佳性能与资源平衡。
图6c–d进一步揭示,训练数据占比越高,模型性能越好,分类任务即使在仅使用0.1%数据时仍能获得较高准确率(ROCAUC)。图6e–f表明,模型参数宽度越大,表现越优,符合深度学习中“扩展规律(Scaling Law)”。因此,若计算资源有限,也可选择宽度较小的模型(如 4096)进行部署,在维持性能的同时控制资源消耗,为实际应用提供灵活的配置参考。
讨论
BAITSAO是一种面向药物协同效应预测的统一模型,结合大语言模型(LLM)嵌入表示与多任务学习(MTL)框架,能够同时完成药物组合的协同预测与单药抑制效果分析。通过统一的数据构建流程,BAITSAO有效解决了跨数据集药物与细胞系名称不一致的问题,并在多项基准测试中表现出领先的分类与回归预测能力。此外,预训练策略显著提升了模型的泛化能力,使其在零样本分类任务中亦能维持良好性能。
研究还通过敏感性分析优化了模型结构和参数设置,验证了在小规模数据条件下模型依然具备稳定表现,并展示了模型在参数扩展下的性能提升趋势。尽管如此,BAITSAO在缺乏功能或结构描述的新药物上预测效果有限,未来有望通过整合单细胞数据与全基因组关联研究(GWAS)进一步增强模型在早期药物开发场景中的适用性。
编译|于洲
审稿|王梓旭
参考资料
Liu, T., Chu, T., Luo, X. et al. Building a unified model for drug synergy analysis powered by large language models. Nat Commun 16, 4537 (2025).
https://doi.org/10.1038/s41467-025-59822-y
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢