DRUGAI
迄今为止,计算设计的酶通常表现出较低的催化效率,并且需要大量实验优化才能达到天然酶的活性水平。这些结果暴露了设计方法的局限性,并暗示了对生物催化基本原理理解存在关键空白。研究人员提出了一种完全基于计算的工作流程,在不依赖突变库筛选的情况下,使用天然蛋白骨架片段在TIM-barrel折叠结构中设计高效酶。三种Kemp消除酶的设计显示出高于2000 M⁻¹s⁻¹的效率。其中最优设计与任何天然蛋白相比含有超过140个突变,包括一个全新活性位点,具有极高的稳定性(超过85°C)和催化效率(12,700 M⁻¹s⁻¹)及速率(2.8 s⁻¹),相比先前设计提高了两个数量级。进一步地,对以往Kemp消除酶设计中被认为必需的残基进行再设计,使得效率提升至超过10⁵ M⁻¹s⁻¹,速率达30 s⁻¹,其催化参数已与天然酶相当。这一策略克服了设计方法的限制,实现了通过最小实验努力设计稳定且高效的非天然酶。

天然酶具有极高的催化效率和选择性。然而,特别是对非天然反应,计算设计的酶仍难以匹敌。尽管近期进展提升了天然酶的稳定性、表达性和选择性,但完全脱离天然模板进行设计的酶,其活性往往低于天然酶多个数量级。因此,过去的研究通常需反复进行实验进化和高通量筛选,才能获得有效酶。这种方法效率低,且限制于可被筛选的反应类型。此外,依赖大规模随机突变库也反映出对生物催化的控制能力尚不充分。
Kemp消除反应是研究de novo酶设计的经典模型,因为自然界尚未发现专门进化用于此反应的酶。虽然设计方法不断改进,但Kemp消除酶的计算设计通常表现出较低的催化效率和速率,并依赖进一步优化才能接近或超过天然酶的中位值。
影响效率低下的原因包括:活性位点构象偏离设计意图;催化受分子细节高度敏感;设计产物稳定性和表达性不足;固定骨架设计方法难以精确定位非天然催化基团;过渡态模型不准确;蛋白动力学与长程静电相互作用在设计中未被考虑。因此,有观点认为,要克服这些问题可能需要更先进的人工智能技术或高通量数据支持。研究人员验证了是否可通过精确控制骨架和序列设计,在不依赖实验筛选或大数据分析的情况下,解决de novo酶设计面临的限制。
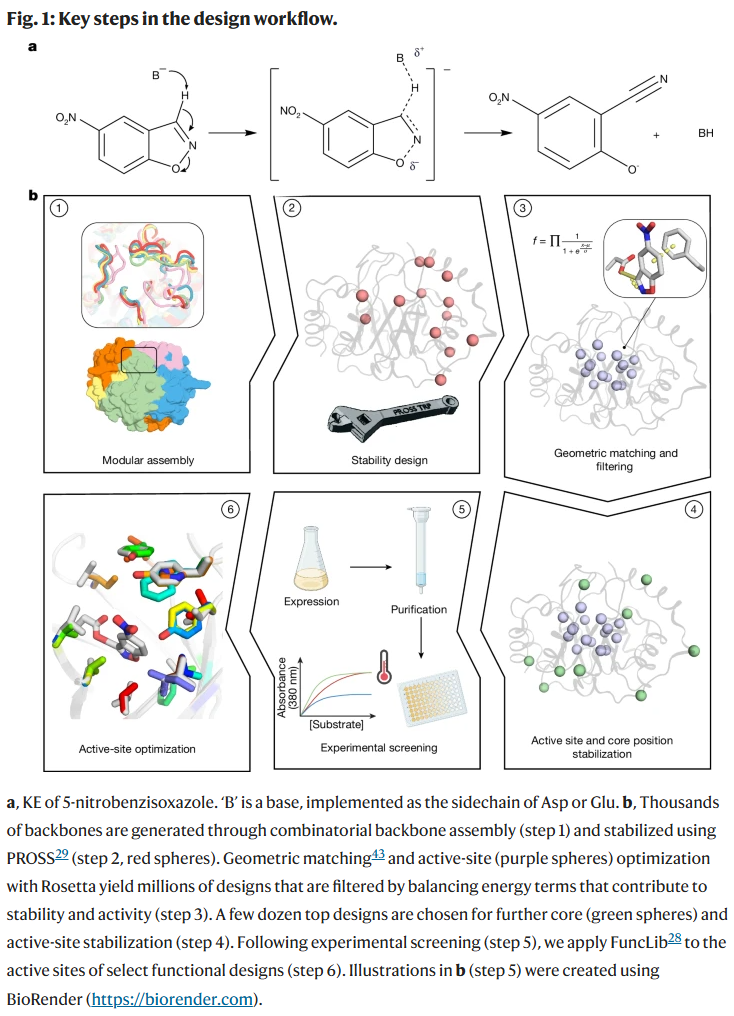
结果
稳定性、可折叠性与活性设计
研究人员的假设是,要设计高效酶,需对蛋白的各个自由度进行控制,以确保稳定性、可折叠性和催化位点的精确定位。尽管过去的设计方法追求完美的可折叠性,但缺乏支持复杂功能所需的非理想结构元素。研究人员选用TIM-barrel这一广泛存在于天然酶中的折叠类型,其中心β桶面向活性腔,适合精确放置催化基团。
所开发的工作流程基于片段组装生成数千种骨架,并通过稳定性设计工具进行优化,随后利用几何匹配和Rosetta原子级能量优化活性位点,得到数百万种设计方案,并通过多目标“模糊逻辑”筛选,最终选出几十种用于进一步活性位点和蛋白核心的优化。整个流程强调对全蛋白稳定性的系统设计。
高效稳定的Kemp消除酶
研究人员将流程应用于IGPS酶家族。共测试了73种设计,其中66种可溶表达,14种表现出协同热变性行为,3种在初筛中检测到Kemp消除活性。进一步使用FuncLib对活性位点进行计算优化后,获得多个突变体,其催化效率提高10–70倍,其中Des27.7达到12,700 M⁻¹s⁻¹、kcat为2.85 s⁻¹,超过此前任何计算设计。
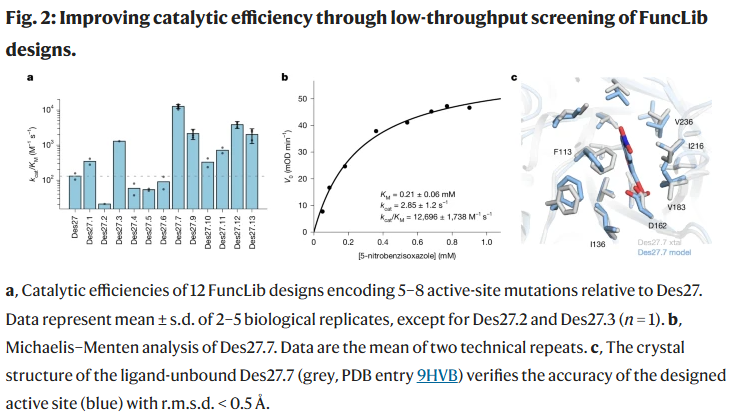
通过结构和动力学分析,研究人员发现一系列保守性突变(如Ile136Val、Ile216Val和Val183Ile)增强了活性位点疏水堆积,提高了前组织性和去溶剂化程度,从而提升催化效率。分子动力学模拟表明,优化后的设计能更稳定地结合底物,并且底物可采用两种互为反向的有效结合方式进行催化,表明其活性腔具备较强的结构灵活性和前组织性。
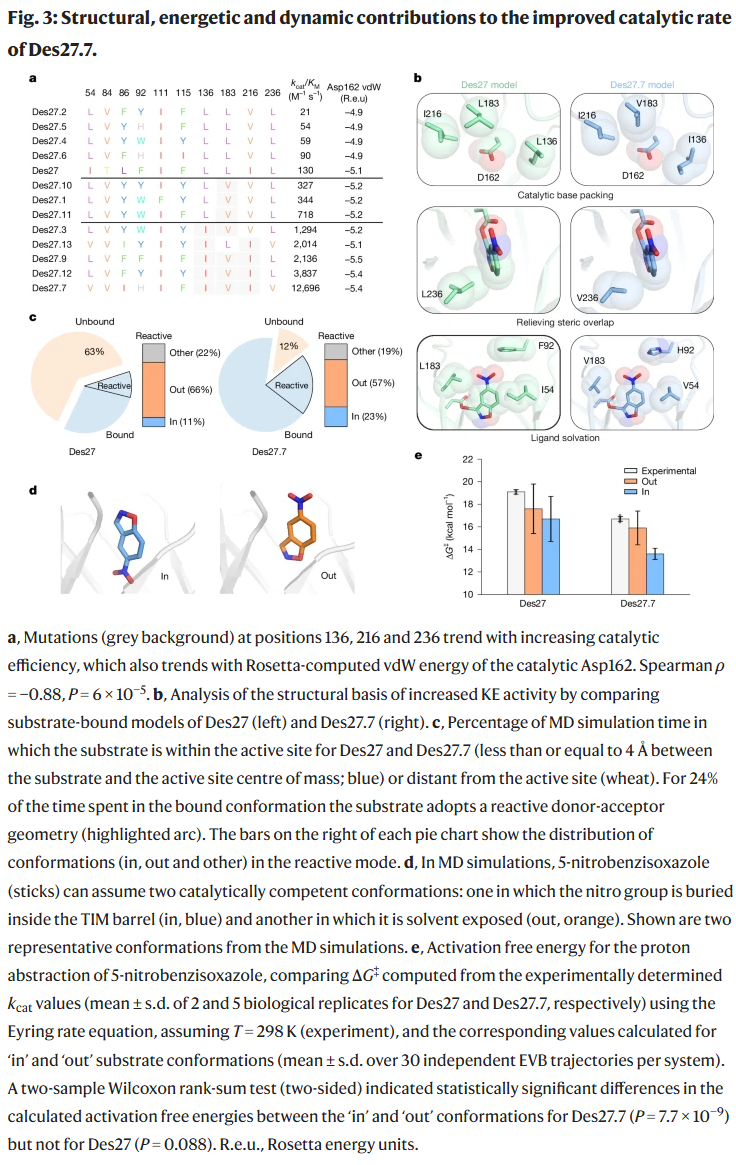
验证设计准确性
通过晶体学结构验证,Des27.7的活性位点与设计模型高度一致(20个残基全原子均方根偏差低于0.7 Å),支持该设计流程可在不依赖实验筛选的前提下构建准确的de novo活性位点并生成高效酶。
探讨设计的必要与充分条件
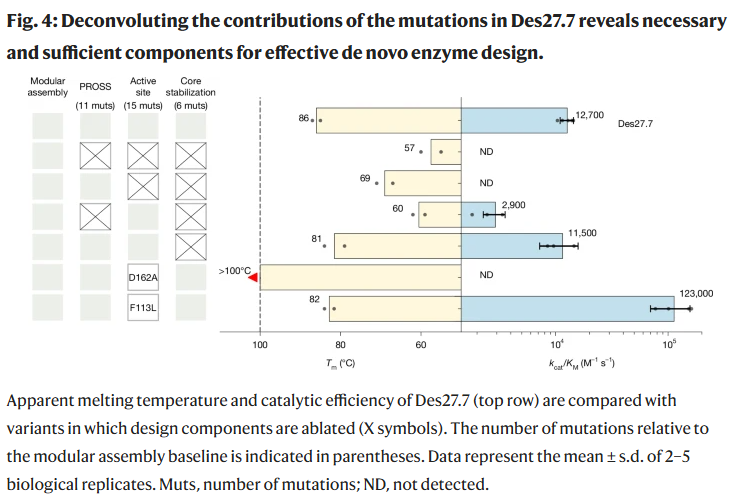
研究人员解构了设计流程各组成部分对最终酶性能的贡献。去除稳定性设计会显著降低表达量与热稳定性;去除活性位点设计则完全丧失催化活性;完整组合三者可协同显著提升稳定性和活性。此外,对催化基Asp162进行突变会完全失去活性,同时使蛋白稳定性大幅提升,说明其活性核心的构建带来了显著稳定性代价。
更值得注意的是,移除以往被认为必要的芳香残基后(如Phe113→Leu),不仅未降低活性,反而使催化效率提升至123,000 M⁻¹s⁻¹,速率达到30 s⁻¹,显示活性位点的设计可以突破原有的结构假设,挑战了长期以来对芳香基团在Kemp反应中作用的共识。
讨论
该研究展示了一个无需高通量筛选和实验进化即可实现的、完整的、计算驱动的酶设计流程。设计出的TIM-barrel折叠结构具有高度稳定性和多样性,并能在一次设计中获得覆盖三个数量级活性的候选。结果表明,只要具备骨架多样性、高蛋白稳定性和活性位点前组织性,即可实现高效的de novo酶催化。
更重要的是,该策略表明,当前原子级别的蛋白设计方法已足够可靠,无需人工智能生成结构或大规模数据分析即可实现高效催化,为未来完全可编程的生物催化奠定基础。
整理 | WJM
参考资料
Listov, D., Vos, E., Hoffka, G. et al. Complete computational design of high-efficiency Kemp elimination enzymes. Nature (2025).
https://doi.org/10.1038/s41586-025-09136-2
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢