DRUGAI
除了承担蛋白编码功能外,mRNA在特定细胞环境中还具有调控作用,参与多种关键生物过程。然而,传统的生化实验与计算方法在探索新的mRNA功能方面仍面临诸多限制。为应对这一挑战,研究人员提出人工智能(AI)可以通过加强RNA生物学家与计算科学家之间的协作,推动该领域的深层创新。在本文中,研究人员强调了mRNA中的非编码区域,包括内含子、5′与3′非翻译区,在调控mRNA代谢与相互作用网络中的关键作用,并指出当前在表征这些区域方面仍存在显著挑战。研究人员进一步探讨了大语言模型(LLMs)在学习RNA序列语义表示方面的潜力,并提出了一种结合图神经网络(GNNs)的方法框架,旨在整合公开可用的测序数据与知识图谱资源。通过采纳这一技术路线,未来有望实现RNA与多种分子之间互作的预测,并构建出具有上下文依赖特性的mRNA相互作用图谱。

RNA是一种古老而核心的生物分子,负责将染色质中的信息传递至各个细胞亚区。RNA世界假说认为,RNA在进化早期可能先于DNA出现,具备执行多种无需蛋白辅助的功能,如自我剪接以及逆转录等酶促反应。在过去的三十年中,RNA的功能认知发生了根本性变化。RNA不再被视为简单的信息中介,而被认为在多种广泛且特异的细胞过程中扮演关键角色。越来越多的研究发现,前体mRNA中的调控区域,如内含子、5′非翻译区和3′非翻译区,可能具有未被充分认识的功能,例如合成短肽等。由于多种人类疾病(包括癌症与神经退行性疾病)均与RNA调控紊乱密切相关,RNA已成为解析复杂疾病机制和开发新疗法的重要突破口。
然而,相较于DNA,RNA结构更加动态和复杂,因而其功能机制难以预测。此外,RNA与其结合分子之间的互作受到结构可达性、修饰状态以及二级三维结构等多重因素的影响。这些挑战决定了传统方法难以从少量RNA分子中总结出通用规律。因此,为揭示RNA功能与互作机制,亟需更先进的计算工具辅助探索。
在此背景下,研究人员提出利用人工智能(尤其是LLMs、GNNs和融合专家知识的建模策略)提升RNA序列的语义建模能力,从而实现更精确的互作预测与结构功能解析。这种方法不仅有助于阐明非编码区域的调控作用,还有望推动RNA互作网络的系统性建模与定量化分析。
mRNA非编码区的多样性功能
研究人员指出,mRNA的非编码区(如内含子、5′UTR与3′UTR)并非仅仅作为“非功能性序列”存在,而是参与了稳定性、定位、翻译调控等多重层面。这些区域在神经元中尤为复杂且功能独特。例如,3′UTR序列可决定mRNA是否被定位至轴突或树突末端。此外,保守性高的内含子序列也可富集RNA结合蛋白(RBPs)与microRNA的识别位点,进而调控剪接、运输和翻译过程。更进一步,一些非编码区域甚至可被翻译为功能性短肽,打破了“非编码即无翻译潜能”的传统观念。研究人员还指出,mRNA的非编码部分可通过剪接变体或位点选择产生新的非编码RNA,这些转录本在不同组织和发育阶段中可能具备特异性调控功能。
RNA结构与互作网络的动态构建
mRNA可通过与蛋白质、DNA、其他RNA(如miRNA和circRNA)等分子形成复杂的多层次互作网络。研究人员强调了RNA序列、二级结构、修饰(如m6A和ψ)以及RBP识别位点在决定互作强度与选择性方面的关键作用。这种互作网络具有高度的动态性与模块化特征。例如,RNA可在特定条件下通过结构变化释放或屏蔽结合位点,调节其与RBPs的亲和力。研究人员还指出,细胞类型、发育阶段和亚细胞位置等因素均可影响互作模式,使RNA在不同环境中表现出上下文特异性的功能。为此,有必要开发能表征结构动态变化的AI模型,并将修饰信息整合至预测框架中。
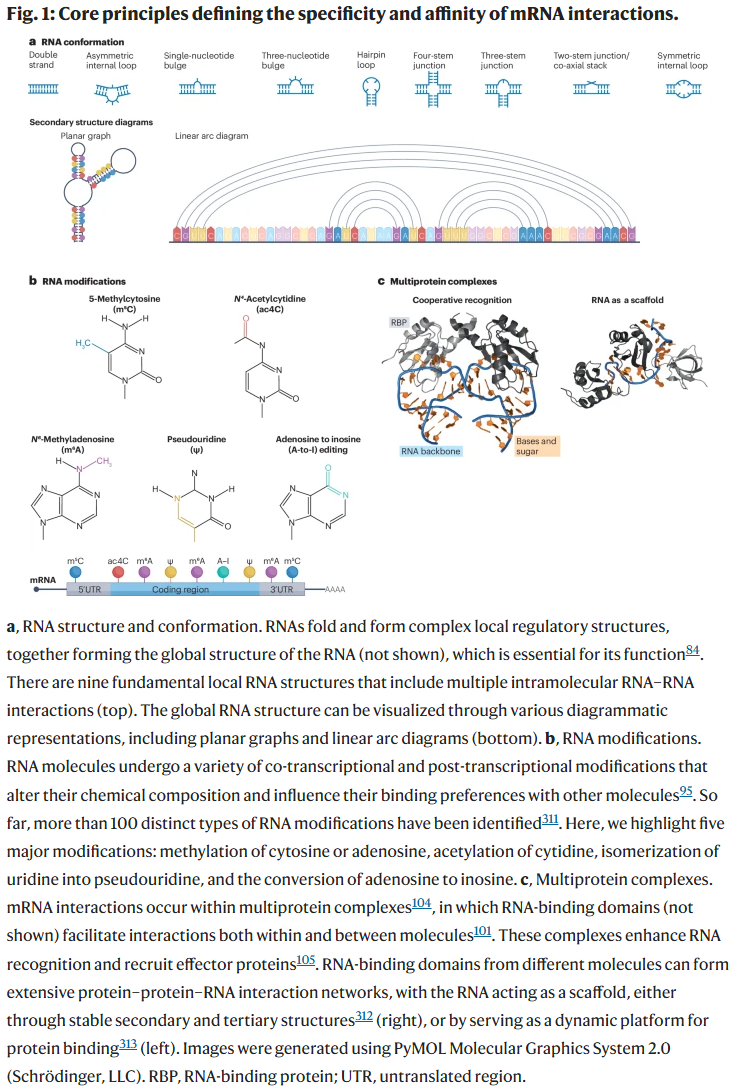
利用大语言模型(LLMs)解析RNA序列功能
传统的监督学习模型虽在特定任务中表现良好,但其训练依赖标注数据,泛化能力受限。相比之下,RNA语言模型(RNA-LMs)通过自监督学习可从海量未标注RNA序列中学习潜在语义特征。这些模型生成的嵌入表示不仅能被用于预测RNA翻译效率、剪接位点及修饰位点,还在建模RNA–RBP互作方面表现优异,甚至在某些任务中超过基于实验结构的模型。研究人员指出,RNA-LMs能够从序列中隐式学习结构信息,展示出类比于蛋白语言模型的泛化能力。
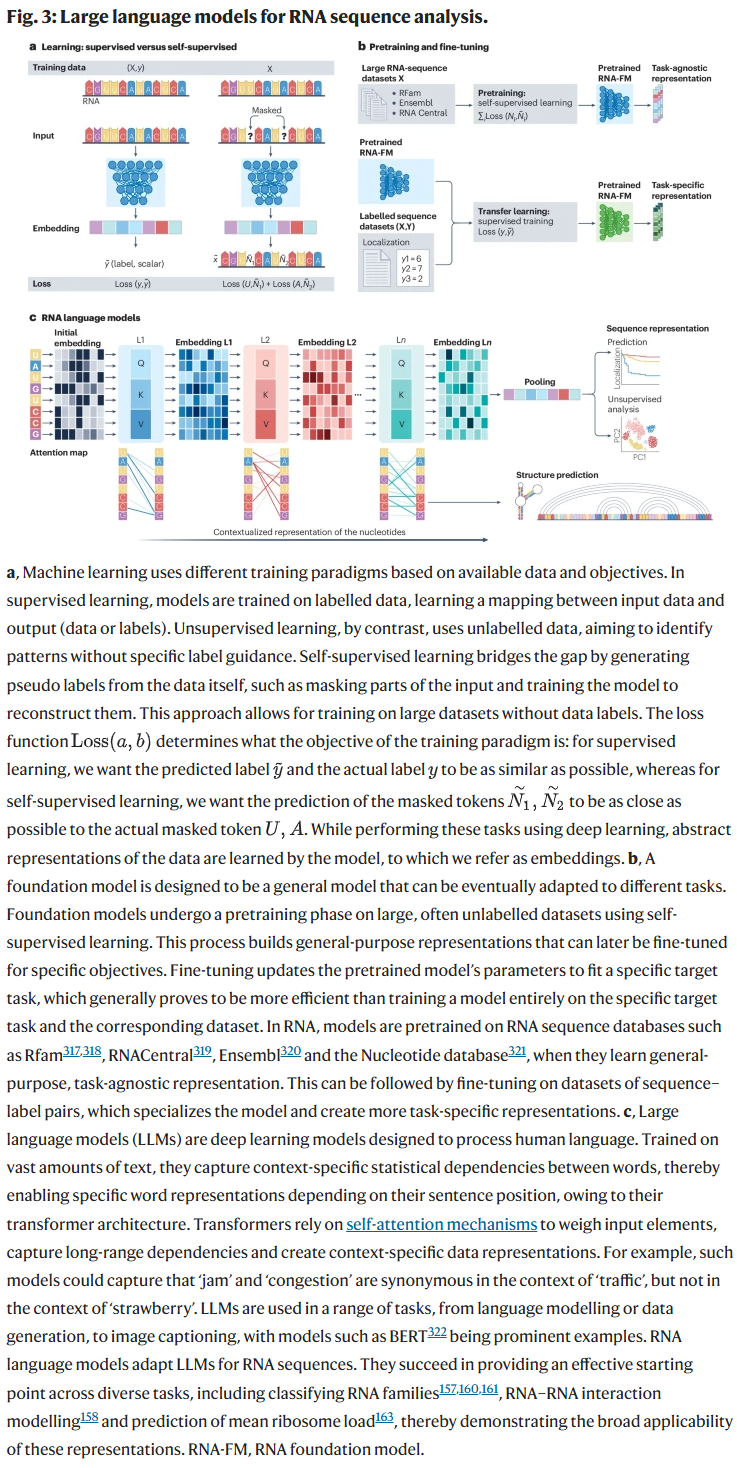
未来改进方向:RNA-LMs与GNNs的融合
尽管RNA-LMs展现出强大能力,但其仍面临可解释性差、输入长度受限和结构信息缺失等挑战。研究人员提出,可通过多任务训练、结构感知的掩码机制、跨模态融合(如结合RNA结构图或化学图谱)等方式对模型进行优化。同时,标准化的模型评估基准也是推动模型持续改进的重要前提。为更全面捕捉RNA与蛋白、染色质及其他RNA的互作,研究人员建议将LLMs与GNNs结合,构建跨分子层级的网络模型,从而生成细胞环境特异的RNA互作图谱,并实现功能预测。

讨论
本研究强调,RNA的非编码区域具有超越传统认知的多重调控功能,且这些功能高度依赖序列、结构、修饰状态与互作环境。在实验技术尚难以全面解析RNA动态互作网络的背景下,人工智能提供了一条具备可行性的新路径。
RNA语言模型已在多个任务中取得进展,但要实现RNA功能的全面建模,仍需考虑互作网络的结构特征与多模态信息整合。研究人员提出,应在保持RNA序列表示通用性的同时,引入结构推断、多源数据融合及上下文感知机制,以增强模型的泛化与解释能力。
通过与图神经网络结合,RNA-LMs可进一步拓展其应用边界,实现跨分子、跨层级、跨环境的RNA功能建模。这种组合策略不仅可用于构建RNA–RNA与RNA–蛋白互作图谱,还可对不同细胞类型中的RNA行为差异提供预测依据,为RNA功能研究与靶向疗法开发提供新工具。
最终,研究人员呼吁建立统一的RNA建模评估标准,鼓励数据与模型的开放共享,以推动RNA人工智能研究迈向新阶段。
整理 | WJM
参考资料
Jung, V., Vincent-Cuaz, C., Tumescheit, C. et al. Decoding the interactions and functions of non-coding RNA with artificial intelligence. Nat Rev Mol Cell Biol (2025).
https://doi.org/10.1038/s41580-025-00857-w
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢