DRUGAI
基因的转录由启动子和增强子等DNA调控元件控制,而这些元件的活性又受到多种转录因子的调节。由于其中涉及高度复杂的组合逻辑,长期以来一直难以构建能从DNA序列预测基因活性的计算模型。近年来,深度学习技术在表观基因组图谱和高通量报告基因实验中的应用取得了重要进展。这些模型能够以显著精度捕捉调控语法,并在预测非编码变异效应、揭示基因调控机制以及设计生物技术用合成调控元件方面展现出巨大潜力。本文系统讨论了此类方法的原理、可用的训练数据类型以及各类方法的优势与局限性。

基因表达的调控是细胞类型多样性与对外界信号响应能力的基础。表达模式主要由基因周围的调控序列决定,这些调控元件通过识别特定DNA序列短片段的转录因子实现功能。多数基因受到多个调控元件与转录因子的共同作用,形成高度复杂的调控网络。在基础生物学中,基因调控主导了发育与信号转导全过程;在生物技术中,提升基因表达调控能力有助于改善产品与农作物的质量与产量;在临床领域,了解非编码变异对表达的影响可推动新型诊断与治疗策略的发展。
传统机器学习方法广泛用于建模基因表达,部分算法还考虑了染色质三维结构。然而,这些模型的预测能力依然有限。近年来,深度学习的进展为破解调控基因组提供了新思路。尤其是“序列到表达”(S2E)模型能够仅依据DNA序列预测表达水平,有助于揭示调控语法。相较传统方法,S2E模型的一大优势是其可推广能力,能够预测未知序列的表达效应,或设计自然基因组中不存在的合成调控元件。
本文重点介绍了近期发展并应用于S2E任务的深度学习模型,涵盖建模原理、训练数据类型、验证策略以及方法优劣分析。此外,还展示了S2E模型不再是“黑箱”,而可被用来解码调控语法,最终阐述其在遗传学与合成生物学中的应用前景。
结果
深度学习在基因调控中的应用
深度学习能够通过多层操作处理长DNA序列,捕捉复杂的、层级化的非线性模式,使其在S2E建模中表现卓越。典型的模型包括卷积神经网络(CNN)与Transformer等架构,能够识别转录因子结合位点、DNA结构特性及其交互关系。Transformer模型如Enformer和Borzoi已成功整合长距离调控信息,提升了基因表达预测的精度。
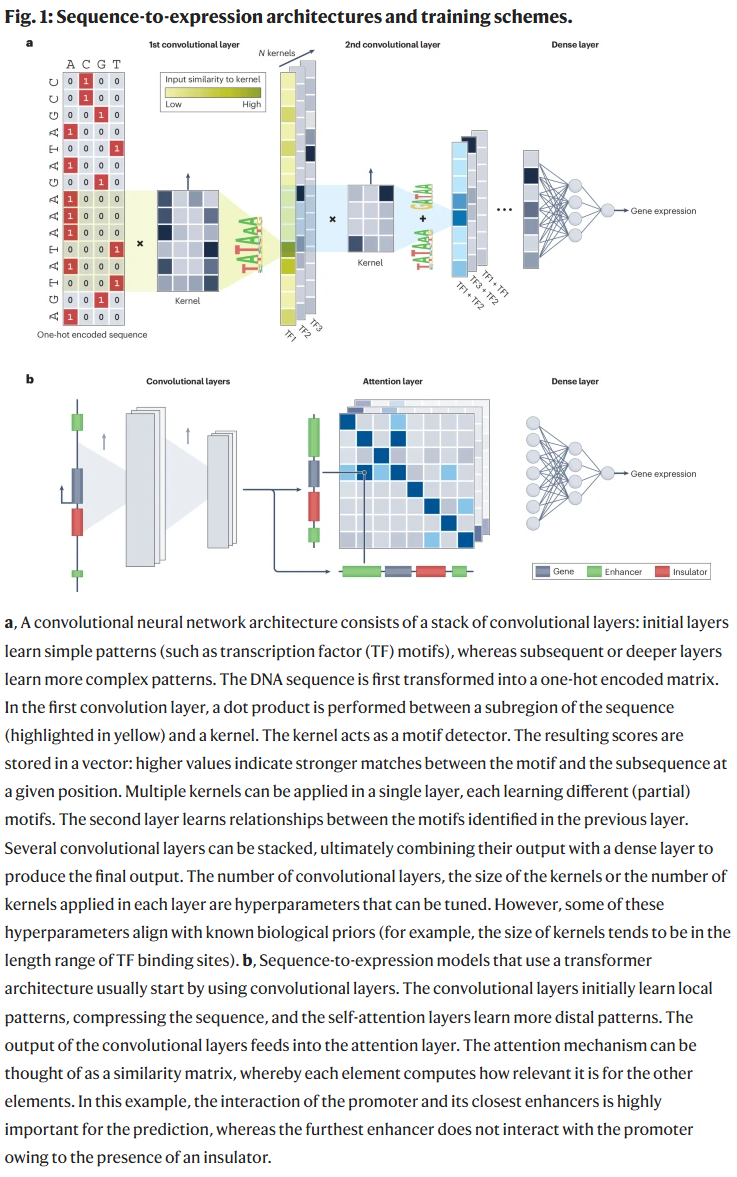
从单任务到多任务建模
S2E模型可训练于多种数据类型,包括表达量测定数据(如RNA-seq)、表观组图谱(如ChIP-seq、ATAC-seq)以及MPRA等。尽管单一任务模型具备更高的解释性和效率,多任务学习有助于充分利用不同数据源的相似性,提升模型泛化能力。转移学习也被广泛采用,即先在大数据集上预训练,再针对特定任务进行微调,提高性能。
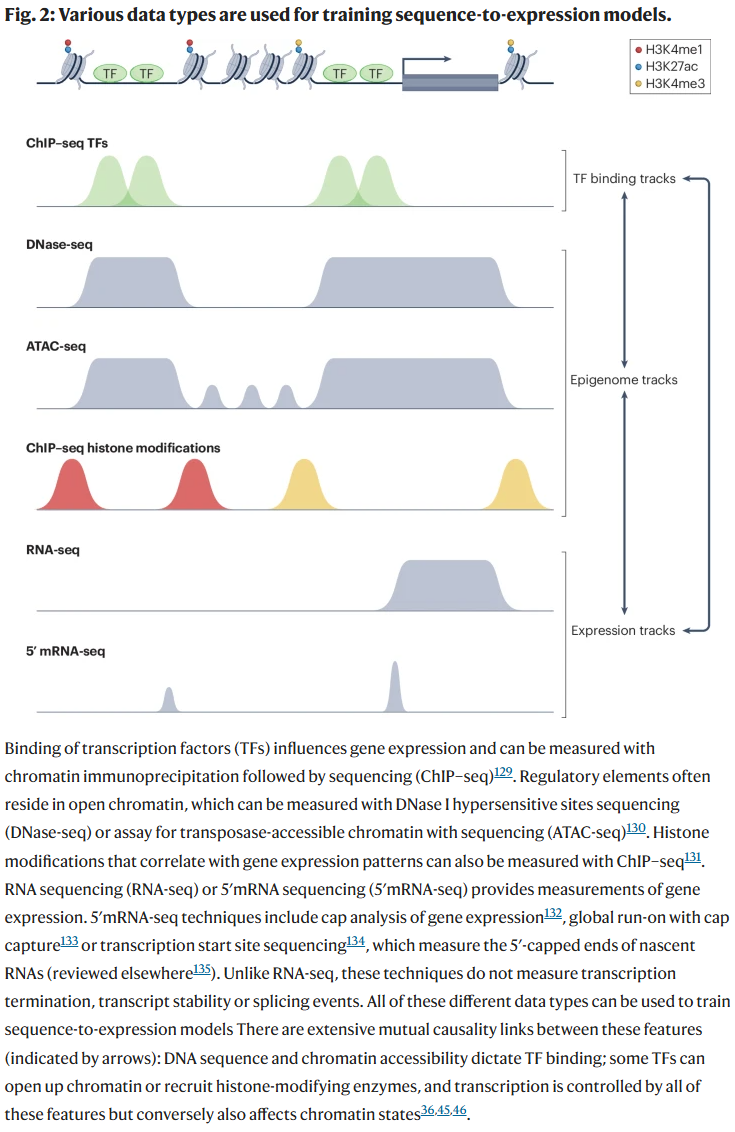
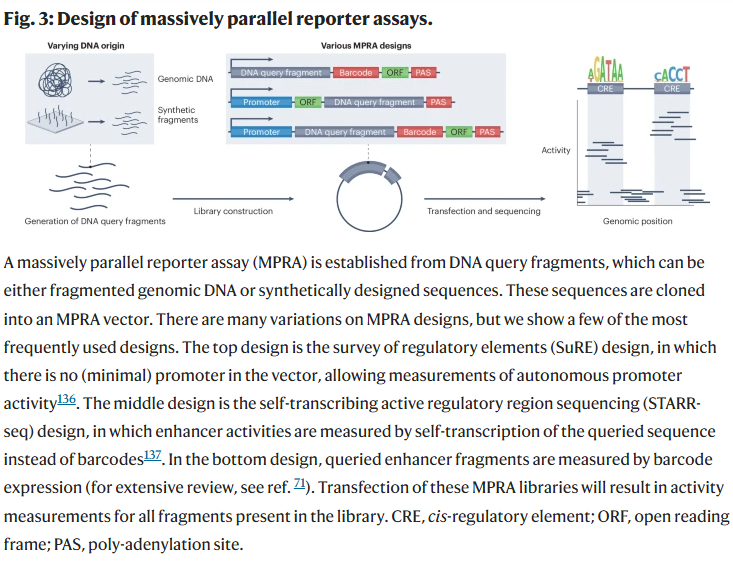
MPRA实验的价值
MPRA可直接测量数以千计短序列的增强子或启动子活性,为模型提供高分辨率的监督信息,弥补传统表观组数据的相关性限制。通过结合CNN与MPRA,研究人员解码了果蝇、哺乳动物等多种物种的调控语法,并在转录因子位置偏好、协同调控机制等方面取得新发现。
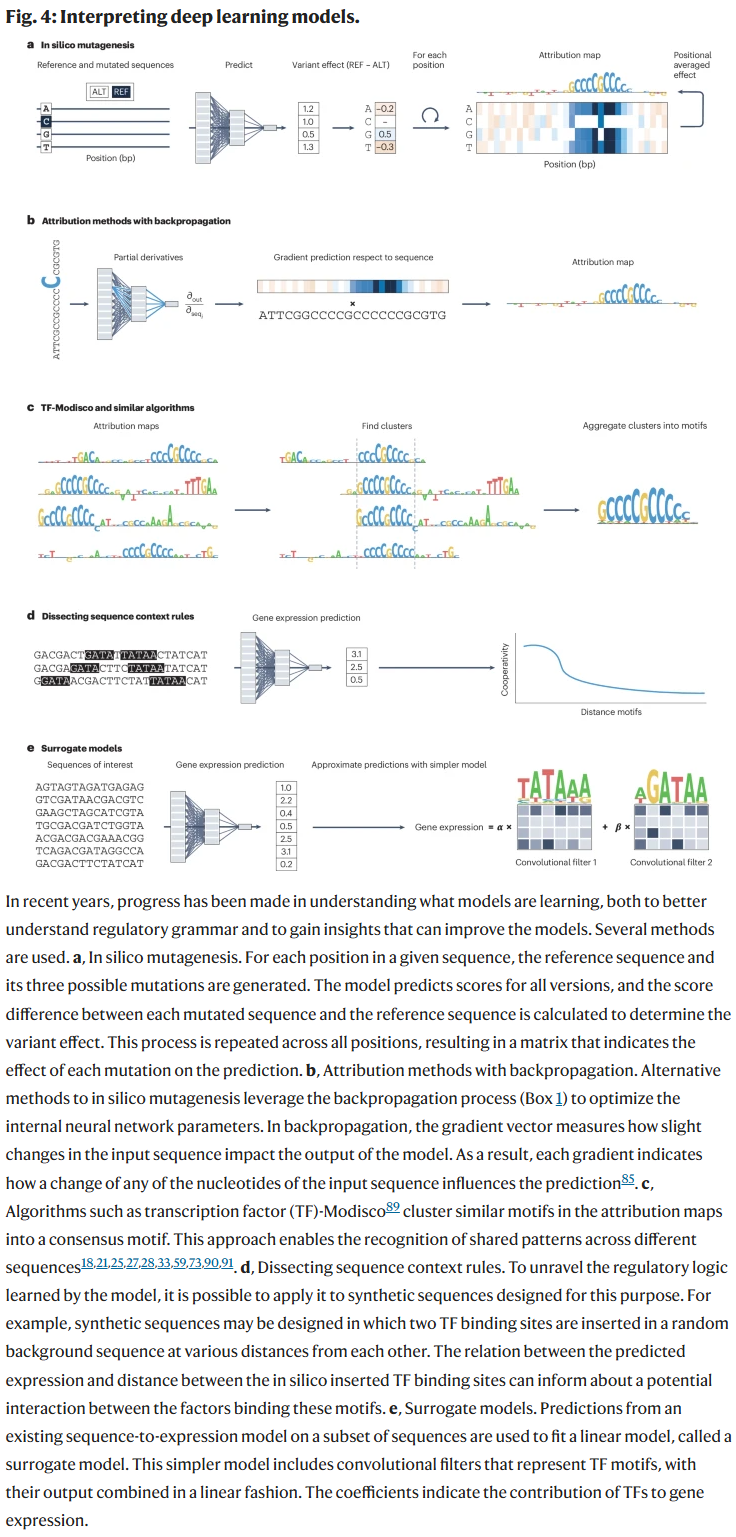
打开“黑箱”:模型解释性
S2E模型的解释性逐渐增强。方法如in silico饱和突变、反向传播归因图(attribution map)、TF-MoDISco聚类算法等,帮助识别关键调控序列及其功能上下文。此外,研究人员还设计了合成序列用于测试调控逻辑,发展了可解释模型(如ExplaiNN)和代理模型(如SQUID)以提升可读性。
独立验证策略
高通量实验验证如MPRA被用于大规模测试模型预测能力。尽管eQTL数据受限于分辨率,结合精细定位策略仍可用于模型基准测试。新兴方法如CRISPRi、Targeted Perturb-seq与序列重定位策略等,为功能验证提供更多手段。
S2E模型的应用进展
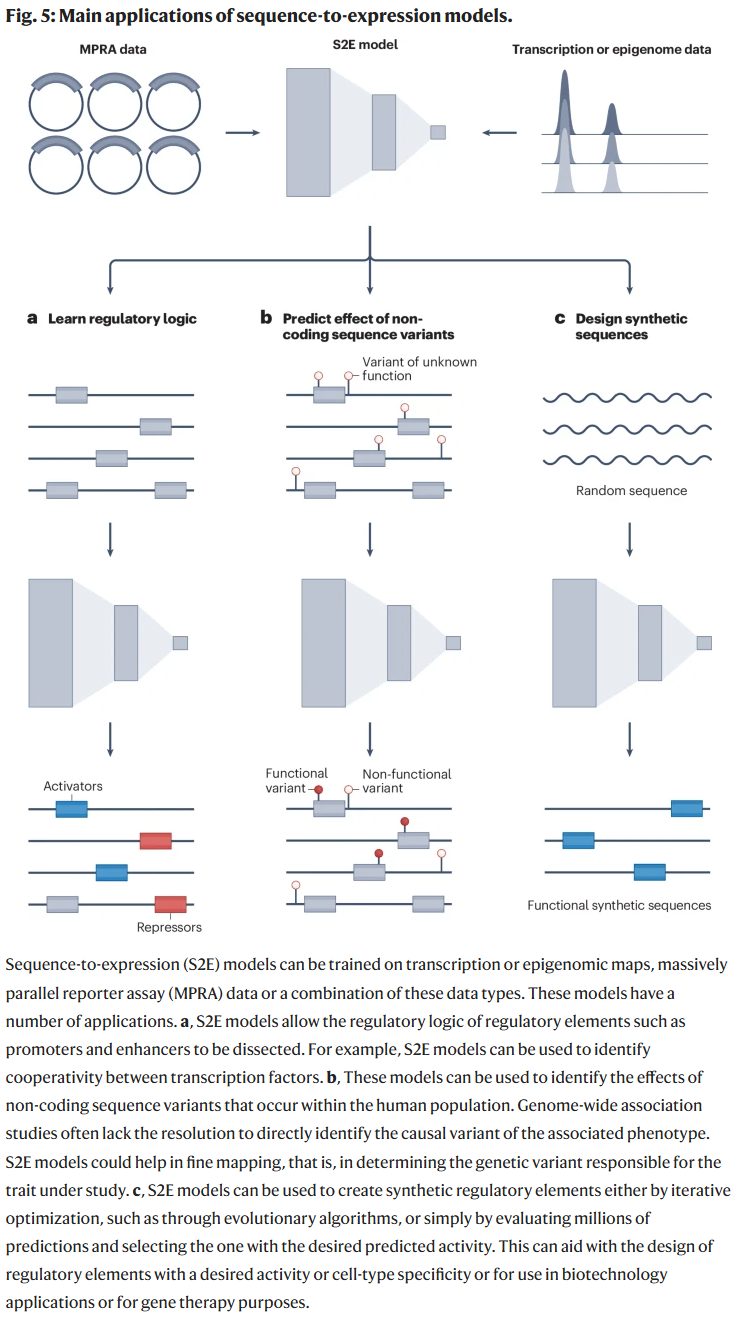
理解顺式调控逻辑:模型揭示了转录起始规律、motif协同作用、低亲和力motif在染色质开放中的作用等关键机制。
预测非编码变异效应:S2E模型能帮助精细定位GWAS中相关的非编码变异,解析其功能机制。
设计合成调控序列:通过S2E模型与进化优化算法结合,研究人员成功设计出高活性的合成启动子或增强子,用于特定细胞类型或生物技术应用。
讨论
S2E模型正逐步向大规模、多任务、跨物种学习方向发展,但在捕捉长距离调控作用、泛化至未知细胞类型或条件方面仍面临挑战。引入基因组先验知识、结合个体化基因组训练、扩大多样化训练数据是未来提升性能的关键方向。
此外,尽管Transformer模型能力强大,但其高计算需求限制了实际应用。构建可解释、紧凑、高效的“小模型”,并制定低成本高质量的数据采集策略,是当前更为可行的路径。值得注意的是,深度学习模型可能产生“幻觉预测”,因此必须辅以严格的实验验证,特别是在临床与工业场景中。
整理 | WJM
参考资料
Barbadilla-Martínez, L., Klaassen, N., van Steensel, B. et al. Predicting gene expression from DNA sequence using deep learning models. Nat Rev Genet (2025).
https://doi.org/10.1038/s41576-025-00841-2
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢