DRUGAI
联合用药的快速兴起为临床治疗带来了显著优势,但同时也增加了不良药物反应(ADRs)的风险。为了确保精准医疗与合理用药管理,亟需一种高效、可解释的计算方法来预测ADRs。然而,传统的机器学习和近年来的发展中的深度学习架构,往往难以从器官视角解释ADR所涉及的关键蛋白互作,也缺乏对ADR之间关联性的显式建模。为此,研究人员提出了一个增强关联学习的模型OrganADR,能够在器官层面预测新兴联合用药可能导致的ADRs。该模型融合了器官层级的ADR信息、分子层级的药物数据以及基于图网络的生物医学知识,通过多模块协同建模,实现了从微观分子到宏观器官的跨尺度信息整合。对15个器官的评估显示,OrganADR不仅在预测准确率方面达到领先水平,还提供了可解释的预测结果和网络机制洞察,为精准医疗中ADR的规避和药物开发提供了重要支持。

联合用药已成为现代临床治疗中的重要策略,能提升疗效、减少耐药性与毒性。然而,由于药物之间的相互作用,其所引发的ADR问题日益严重。现有研究显示,全球范围内因ADR引发的住院率介于3.6%至15.6%之间,严重影响患者健康并增加医疗负担。
由于ADR可能由多种生物机制引起,加之缺乏大规模的临床试验数据,尤其是在新兴药物组合缺乏历史记录的情境下(即冷启动场景),使得准确预测ADR成为一大挑战。面对实验手段的局限性,研究人员逐步转向使用计算模型进行ADR预测。早期模型通常依赖于药物对与其对应的ADR标签训练分类器,而后续深度学习方法虽增强了特征提取能力,但常忽视了药物间交互机制与器官层面的信息关联。
此外,现有知识图谱(KG)方法虽提供了跨域生物医学知识整合能力,但往往将多标签ADR预测任务转化为独立的二分类问题,忽略了器官之间在ADR上的内在联系。事实上,许多免疫相关ADR会同时影响肠道、肝脏和神经系统,因此建立一种能够建模器官间ADR关联性的模型具有重要临床意义。
研究人员提出的OrganADR正是针对上述问题设计而成。其将药物分子信息与生物医学KG知识进行整合,并引入器官层面的ADR关联矩阵,引导图神经网络通过端到端的学习方式进行跨尺度建模,实现了对新兴联合用药ADR的精确预测和机制解释。
结果
器官层级ADR预测任务设计
研究人员将药物划分为“新兴药物”与“已知药物”两类,提出两个任务:一是预测两个新兴药物组合产生的ADR,二是预测一个新兴药物与一个已知药物组合的ADR。模型输入为两个药物的分子特征、一个生物医学KG和一个预构建的器官级ADR关联矩阵,目标是对15个器官层面的ADR进行多标签预测,并解释可能的蛋白互作路径。
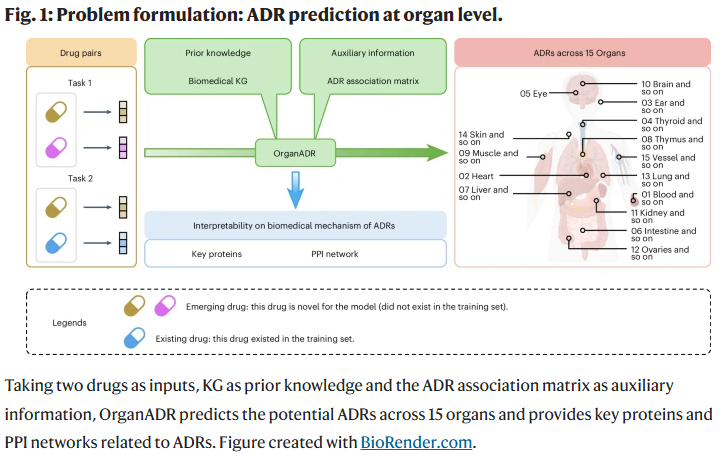
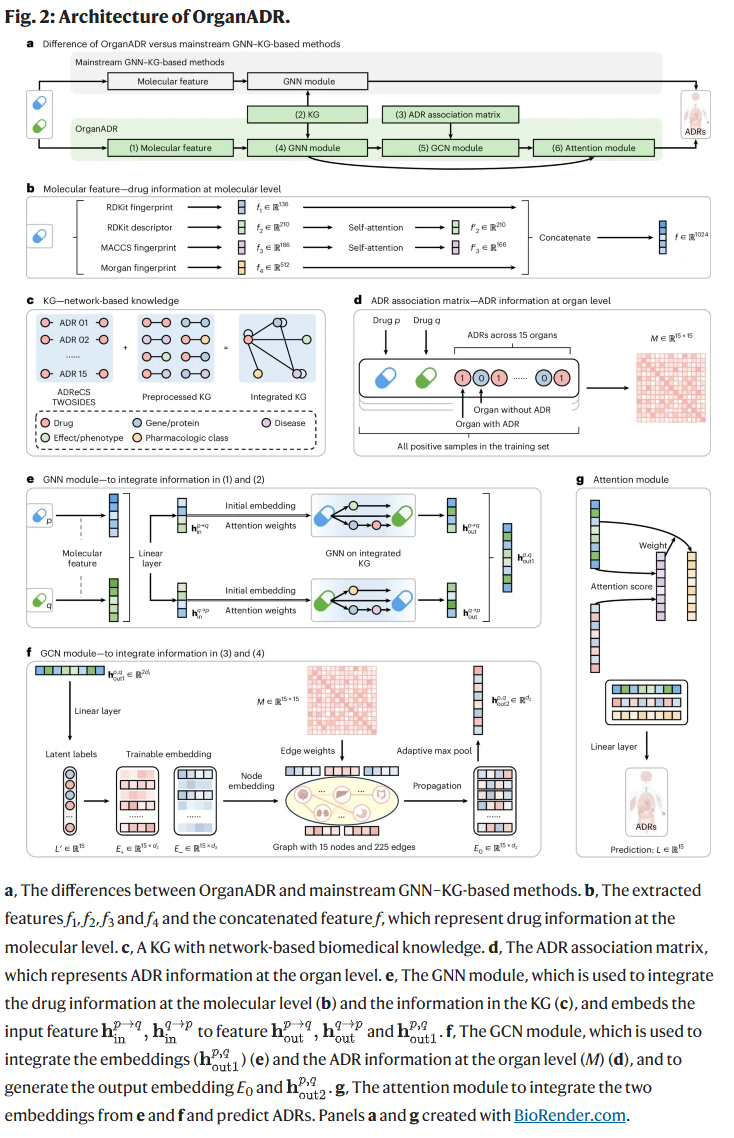
OrganADR模型架构
OrganADR 模型由六个核心模块构成,分别为:分子特征提取模块、知识图谱(KG)模块、ADR 关联矩阵模块、图神经网络(GNN)模块、图卷积网络(GCN)模块以及注意力机制模块。这一架构旨在统一整合药物的分子层级信息、器官层级的ADR关联性,以及生物医学网络中的先验知识,以实现对新兴联合用药在器官层面可能引发的不良反应进行准确预测。
首先,在分子特征层面,模型从每个药物中提取三种分子指纹(RDKit、MACCS、Morgan)以及一组连续化学描述符(如分子量、极性表面积等),并对其中的 RDKit 描述符与 MACCS 指纹分别引入可训练的自注意力模块,从而增强对具有明确物理化学意义特征的感知能力。每个药物最终被编码为一个长度为1024的向量。
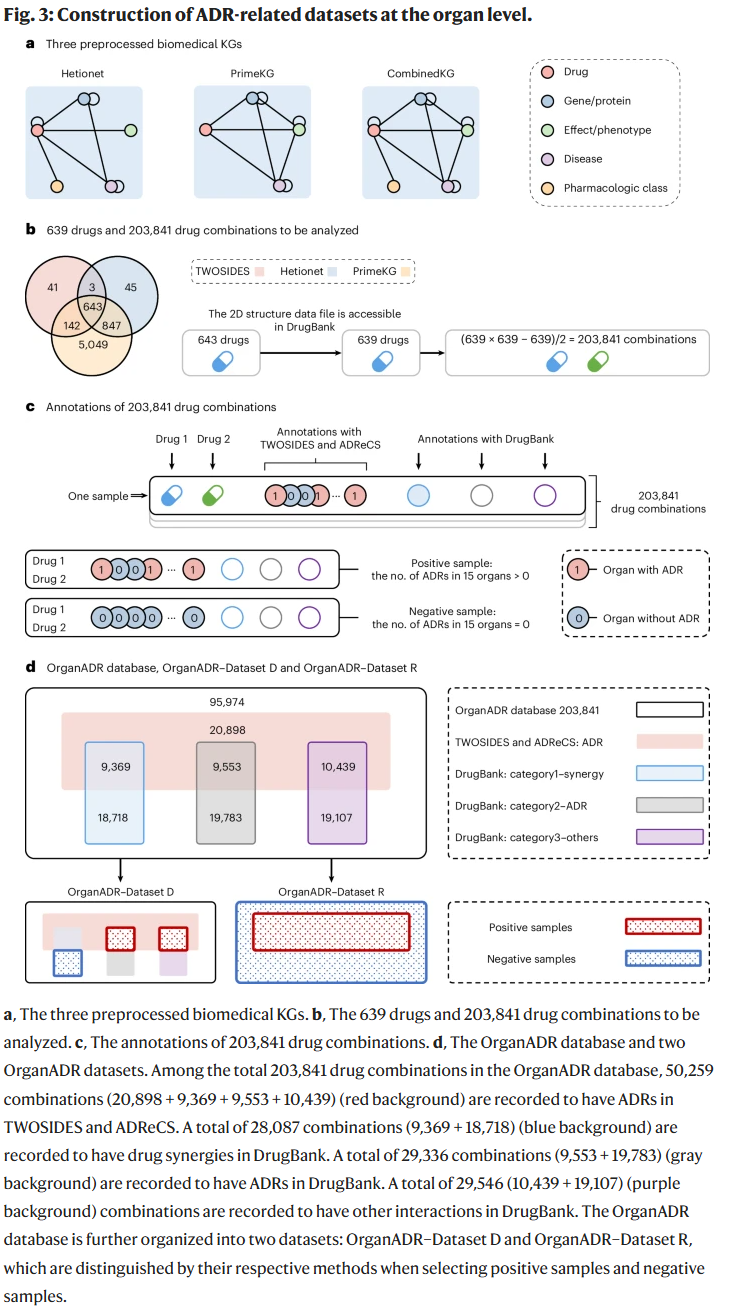
其次,模型引入预处理后的生物医学知识图谱,包括Hetionet、PrimeKG 及其融合版 CombinedKG,用以提供网络层级的药物、靶点、疾病等实体间关联信息。为避免信息泄露,KG 中关于药物协同作用的边在训练前被移除,仅保留合理的单向生物学连接。
为了表达不同器官间ADR发生的统计关联性,模型在训练集基础上构建了器官级ADR共现矩阵,并通过归一化处理获得对称的关联矩阵。该矩阵在训练、验证与测试过程中保持不变,用作先验辅助信息,引导模型从器官视角识别ADR的潜在传播模式。
在GNN模块中,研究人员采用了流动式图神经网络架构,逐步从药物节点向外传播信息,整合其在知识图谱中可达的生物实体路径,从而获得每对药物组合的嵌入表示。随后,这些表示通过一个多层感知机被转换为“潜在ADR标签”,这些标签被映射至15个器官的嵌入空间,并进一步构建为图结构节点,以器官间的关联矩阵为边权,送入GCN模块进行迭代更新,捕捉潜在标签之间的语义关系。
在最终的注意力模块中,药物嵌入向量与器官级标签嵌入被融合,通过注意力权重计算两者之间的语义对应性,生成融合后的高维表示,最终通过线性多标签分类器对每个器官是否发生ADR进行预测。
整体而言,OrganADR 的模型架构实现了从分子层级到器官层级,再到生物医学知识网络的跨尺度集成,并通过可解释模块协同协作,为预测联合用药引发的不良反应提供了高性能且具可解释性的解决方案。
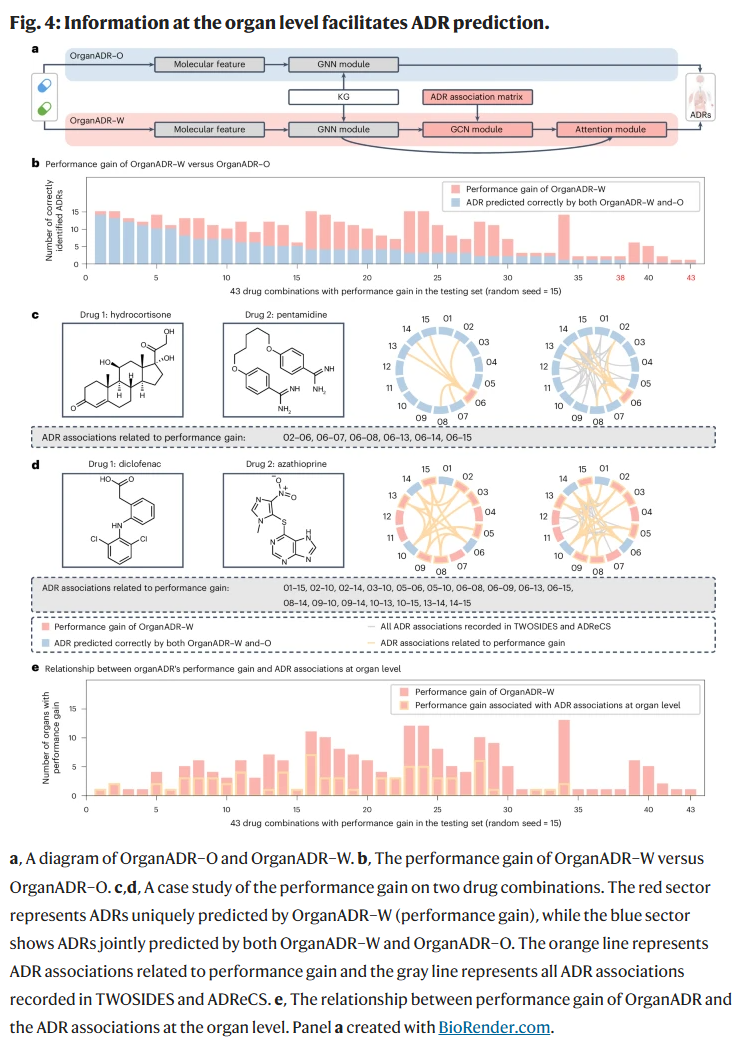
融合器官信息提升预测性能
OrganADR采用注意力机制融合器官层级的ADR关联信息。研究人员构建了一个移除器官信息的对照版本(OrganADR–O),与完整模型(OrganADR–W)进行了对比。在测试集中,OrganADR–W在43个药物组合中实现了预测性能的显著提升,尤其是在17个药物组合中完全复现了临床数据库中的ADR记录。
进一步的因果分析和个案研究(如氢化可的松-喷他脒、双氯芬酸-硫唑嘌呤组合)显示,这些性能提升主要归功于器官层级ADR信息的引入,且与已知的临床禁忌相符,验证了模型的合理性与解释力。
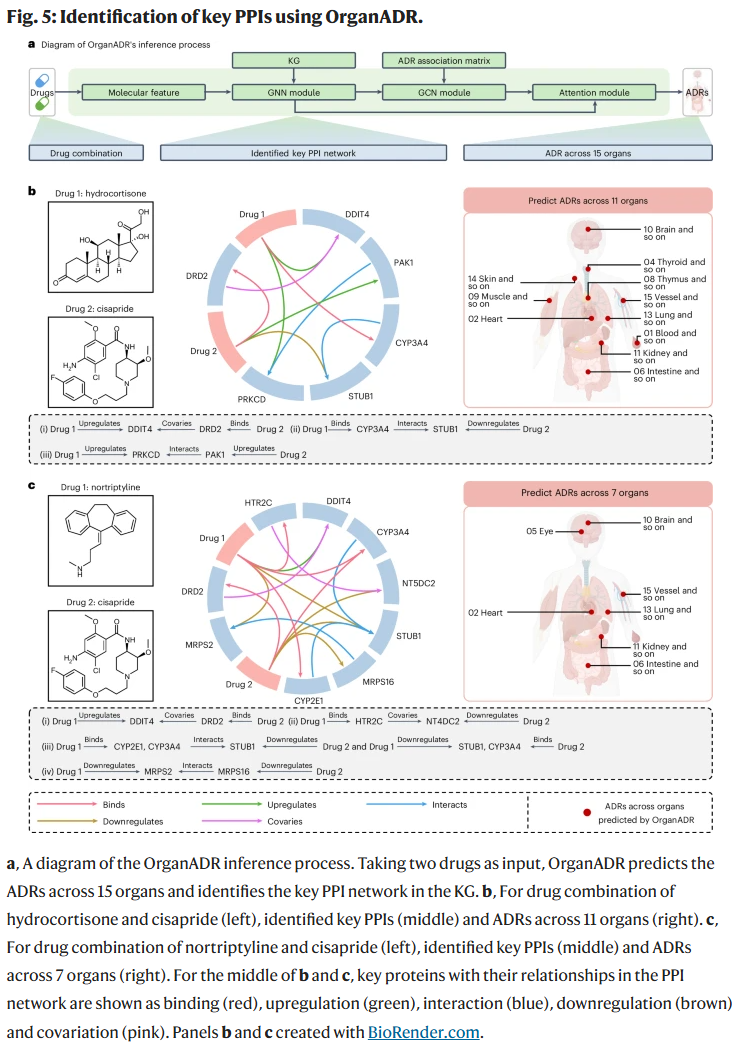
识别关键蛋白互作
OrganADR能够基于KG识别出与ADR相关的关键PPI路径与关键蛋白。例如,在氢化可的松与西沙必利的组合中,模型成功识别出与心脏毒性相关的关键蛋白如DRD2和CYP3A4。在西沙必利与去甲替林的组合中,模型则识别出MRPS2与MRPS16等与线粒体功能和心脏ADR密切相关的蛋白。该能力展示了OrganADR对ADR生物机制的深层捕捉能力。
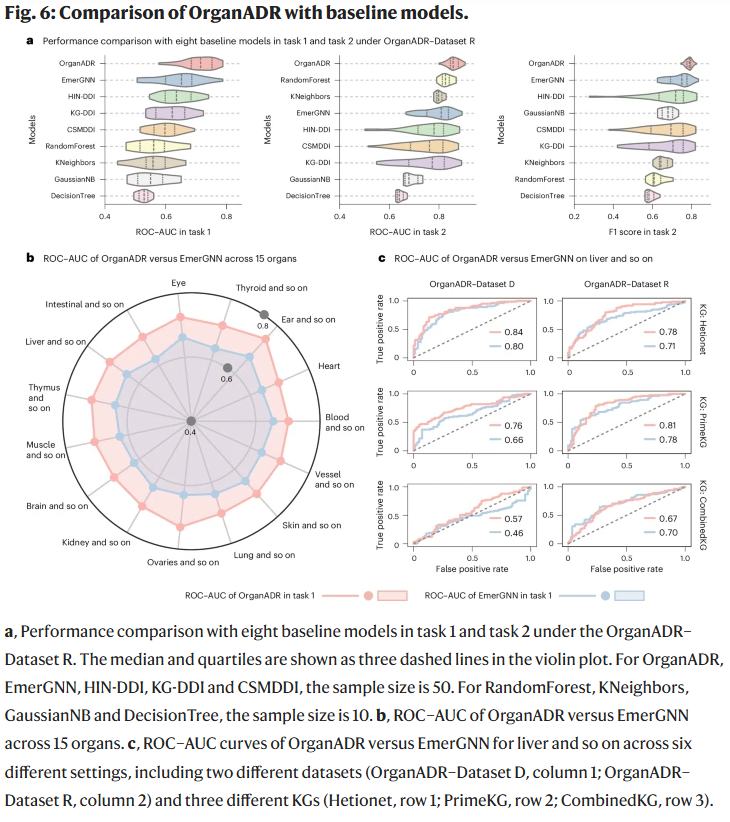
与其他模型的性能对比
OrganADR在两个任务、两种数据集和多种KG设定下,均显著优于八种主流对比模型。在新兴药物组合预测任务中,其ROC–AUC达到71.14%,而最佳对比模型仅为64.19%。在混合组合预测任务中,其F1分数达到78.71%。整体上,OrganADR在准确性、稳定性和跨数据源的鲁棒性方面展现了全面优势。
消融实验验证关联矩阵贡献
通过引入或去除器官级ADR关联矩阵,研究人员系统评估了其在模型性能中的作用。在多个特征设定和数据集条件下,引入该矩阵均带来显著性能提升。例如在任务1中,OrganADR–W比OrganADR–O在ROC–AUC上高出4–6个百分点,表明器官信息在预测中发挥了关键作用。
不同类型关联矩阵的影响
研究人员比较了基于不同方法(Spearman相关性、余弦相似度、Theil’s U系数等)构建的ADR关联矩阵,发现OrganADR自定义的归一化共现矩阵在所有指标上均表现最佳,验证了其构建策略的有效性。
不平衡数据下的鲁棒性
模型在不同正负样本比例下仍表现出较强鲁棒性。在任务1中,即使数据集倾斜至1:4,模型的AUC仍保持在80%左右,说明其具备良好的泛化能力和对数据分布变化的适应性。
讨论
从临床角度看,ADR的发生具有显著的跨尺度与跨器官关联性。若忽略这些联系,模型的可解释性与实际应用价值将大打折扣。OrganADR打破了传统仅关注药物相似性的预测框架,通过整合分子、器官与网络三大层级信息,建立起跨尺度的统一建模方式。
此外,模型的性能不仅依赖于架构本身,还受到数据质量、KG选择、负样本构建等因素影响。研究人员的策略在这些方面进行了系统优化,为后续基于更大规模KG、融合多模态语言模型、或结合更多临床数据的ADR预测研究奠定了基础。
OrganADR以其可解释性强、性能稳健的优势,有望在未来的多病共治、精准医疗和药物组合推荐等实际场景中发挥关键作用。
整理 | WJM
参考资料
Li, B., Qi, Y., Li, B. et al. Predicting adverse drug reactions for combination pharmacotherapy with cross-scale associative learning via attention modules. Nat Comput Sci (2025).
https://doi.org/10.1038/s43588-025-00816-7
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢