DRUGAI
抗体–抗原的结合亲和力是治疗性抗体开发的核心:药效依赖于高特异性的结合能力以及亲和力的可控性。研究人员在此提出了Graphinity,一种基于抗体–抗原结构构建的等变图神经网络架构,可实现实验结合亲和力变化(ΔΔG)预测的Pearson相关系数高达0.87。然而,正如以往方法所示,该模型在目前可用的少量实验数据上存在过拟合现象,且在训练–测试划分上表现出较差的稳健性。
为了探究实现ΔΔG泛化预测所需的数据量与类型,研究人员构建了两个大规模合成数据集:近100万个由FoldX生成的ΔΔG样本,以及超过2万个由Rosetta Flex ddG生成的样本。结果表明,当前的实验数据量不足以支撑准确稳健的ΔΔG预测,或许还需数量级上的增长。此外,除了数据体量,数据的多样性也是影响模型预测性能的重要因素。
本研究为未来预测方法的开发和数据采集工作提供了数据需求的下限估计,强调了推动“可泛化的亲和力预测”迈向现实所需的关键基础。

抗体通过与靶抗原的特异性结合发挥其生理与治疗功能。因而,在治疗性抗体的开发过程中,控制其结合亲和力始终是筛选与优化候选分子的核心目标。
除了亲和力本身,抗体的“可开发性”(developability)也受到多种其他性质的影响,包括自聚集倾向、人源性、多反应性与特异性等。近年来,机器学习方法已广泛应用于这些性质的预测任务,如抗体自聚集、人源性评分、抗原交叉反应性等,取得了显著进展。然而,任何为改善这些性质而引入的抗体序列变异,都必须以不显著损害其靶抗原结合能力为前提。这使得治疗性抗体的设计问题本质上成为一个复杂的多目标优化任务。
传统的亲和力测定实验通常缓慢而繁琐,成为抗体设计流程中的瓶颈。因此,开发一种快速且准确的亲和力变化(ΔΔG)预测模型,可极大加速抗体设计过程。同时,计算预测方法还可结合多个性质评分器,实现多属性联合优化,同时保持高结合力。
尽管如此,抗体–抗原亲和力的计算预测仍然是一个重大挑战。传统方法如 FoldX 和 Rosetta Flex ddG 主要基于物理方程与经验能量项构建,虽然在特定场景中取得良好表现,但在准确性和计算速度上仍存在局限。
近年来,研究重点逐渐转向机器学习(ML)方法,这些方法大致可分为两类:基于序列的方法和基于结构的方法。
序列方法在数据充足的情况下,对特定抗原的预测性能较好,但往往缺乏泛化能力。它们主要学习的是抗原特异性信息,因此难以迁移至其他抗原的预测任务中。
结构方法则试图从多个抗体–抗原复合物中捕捉通用的相互作用模式,更有潜力实现泛化。然而,它们通常依赖于人工特征提取,如结合表面积、原子间相互作用与能量项等,导致效率低下,且容易引入偏差。
因此,如何构建一个结构感知、泛化能力强、可扩展的深度学习架构,用于泛化的抗体–抗原ΔΔG预测,成为该领域尚未解决的核心问题。
结果
Graphinity 模型架构
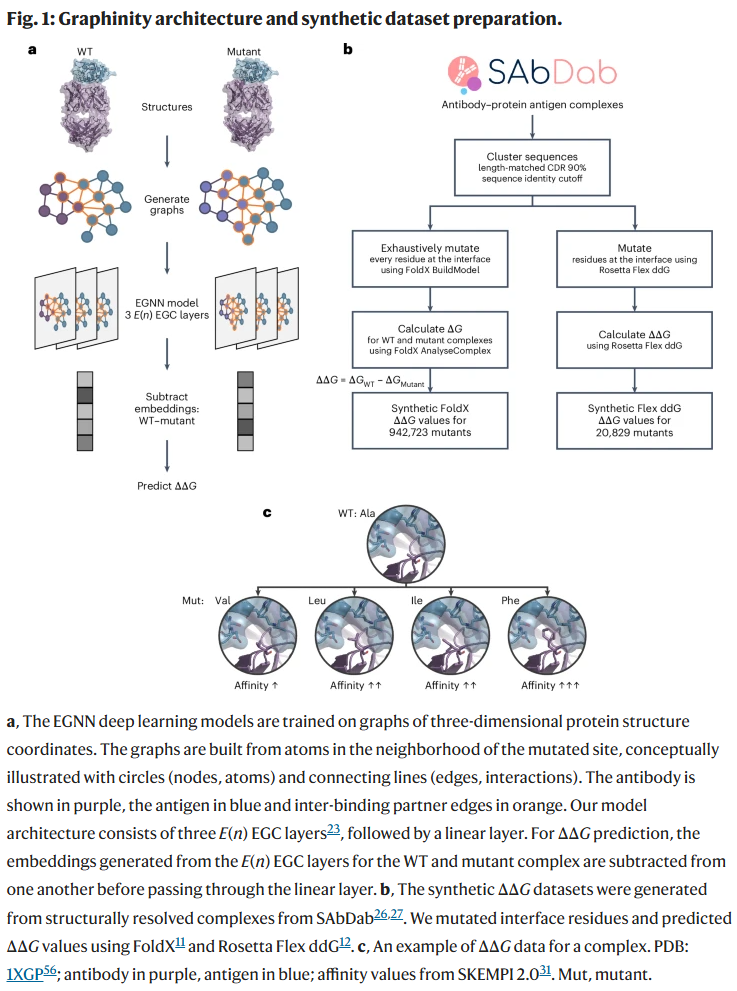
Graphinity 是一个基于等变图神经网络(Equivariant Graph Neural Network, EGNN)的模型,输入为抗体–抗原复合物的野生型(WT)和突变型三维结构。两种结构分别被编码为图,其中非氢原子作为节点,节点间小于4Å的距离被视为边。图结构被限制在突变位点邻域,以提高计算效率。
Graphinity 模型由两个部分组成:
图构建模块:以突变位点为中心,提取其周围邻域形成原子级图表示。
Siamese EGNN 主体网络:将WT与突变体图结构分别输入三层EGNN模块中,得到其嵌入(embedding),随后做向量差(WT减去Mutant)并通过线性层输出ΔΔG预测值。
该架构模块化设计,适配回归与分类任务,支持单图或多图输入,并具备灵活扩展能力。
Graphinity 在实验 ΔΔG 数据集上的预测表现
研究人员首先在 AB-Bind 数据集上评估 Graphinity 的性能。该数据集包含29个抗体–抗原复合物的645个单点突变及其对应的ΔΔG实验值,构成了“Experimental_ΔΔG_645”数据集。
在10折交叉验证下,Graphinity 可实现最高Pearson相关系数达0.87,优于以往已报道方法。然而,进一步分析发现,该高性能可能并不反映真实的泛化能力,而是模型对训练数据的过拟合结果。
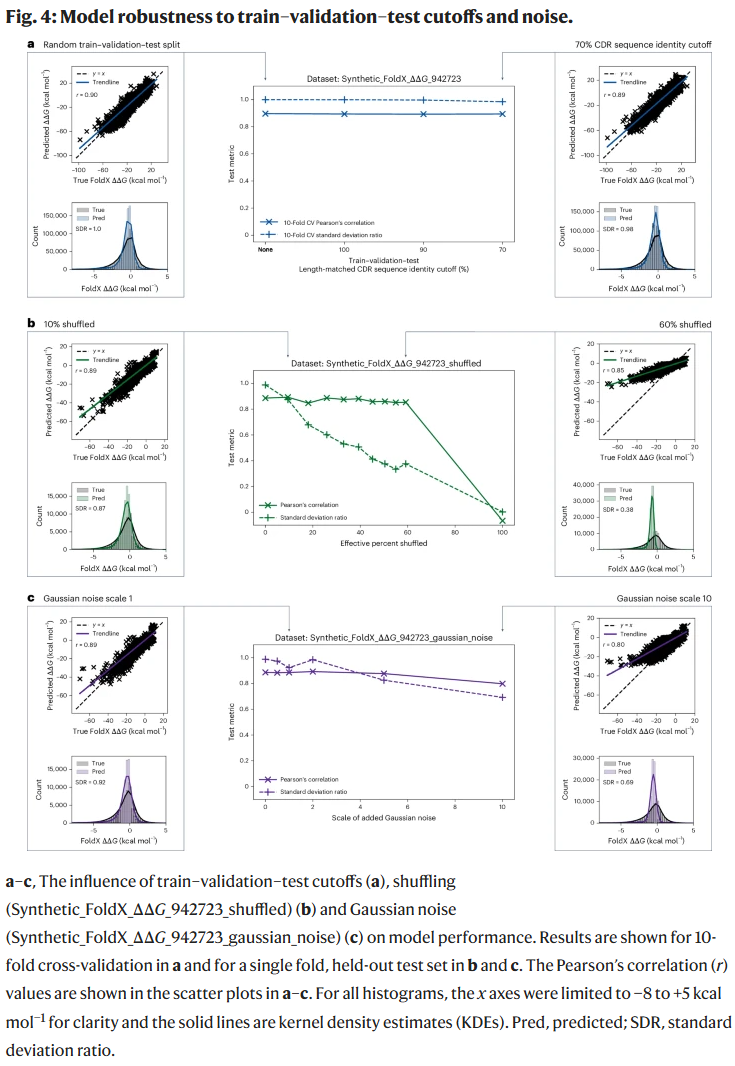
为验证模型是否存在信息泄露,研究人员引入了更严格的训练–测试数据划分策略:
引入CDR区(互补决定区)序列同源性约束(如90%、70%、100%相似性以下不能在同一划分中)。
移除非结合突变样本(ΔΔG被设为固定值-8)以及假设的反向突变。
结果显示,在引入CDR序列划分约束后,模型性能显著下降,平均相关性降低约63%,表明之前的高相关度来自于对同源结构的记忆,而非真实泛化能力。
此外,模型在不同数据划分折之间的表现差异较大,进一步证实其对训练数据分布较为敏感。

利用近百万个突变构建合成 ΔΔG 数据集
实验数据稀缺且分布集中,限制了泛化能力的评估与训练。为此,研究人员基于 FoldX 工具,利用 SAbDab 数据库中解析结构的大量抗体–抗原复合物,系统性生成近百万个ΔΔG预测样本(Synthetic_FoldX_ΔΔG_942723)。
具体流程包括:
对每个复合物界面残基进行穷举式单点突变。
使用 FoldX 分别计算WT与突变体的结合能,得出ΔΔG值。
最终获得94万余个样本,分布广泛,能量范围覆盖广。
尽管这些数据为模拟生成,但FoldX能较好地重现结合变化趋势,特别是对高影响突变(ΔΔG绝对值大于1 kcal/mol)具有良好区分能力,因此可用于大规模训练与性能测试。
在该数据集上,Graphinity 模型在10折交叉验证中取得0.89的Pearson相关性,若延长训练轮数至100轮,可进一步提升至0.92,说明在数据充足情境下,该模型具备较强的拟合能力。
此外,与其他方法(如基于结构接触数变化的简单基线模型、CNN、Rotamer Density Estimate、Equiformer等)相比,Graphinity 达到最优表现,且计算开销较低,是当前性能与效率兼备的结构感知预测架构。
严格的序列同源性划分验证 Graphinity 的泛化能力
研究人员进一步检验了 Graphinity 在不同序列同源性划分条件下的性能。即便施加最严格的数据划分条件(CDR序列和抗原序列同源性均限制在70%以下),Graphinity 仍能保持 0.89 的 Pearson 相关性,显示出较强的泛化能力。
然而,当数据划分依据为 ΔΔG 值本身(即按结合能级别划分训练与测试集)时,相关性降至 0.52,提示模型在处理跨亲和力分布区间的样本时仍面临挑战。
此外,Spearman 排名相关性(衡量预测排序的一致性)在全体数据上约为 0.64,但在去除绝对值低于 1 kcal/mol 的小变化样本后提升至约 0.74。说明模型在预测微弱亲和力变化方面仍存在不足。
大规模合成数据可实现高精度预测,但实验数据仍远远不足
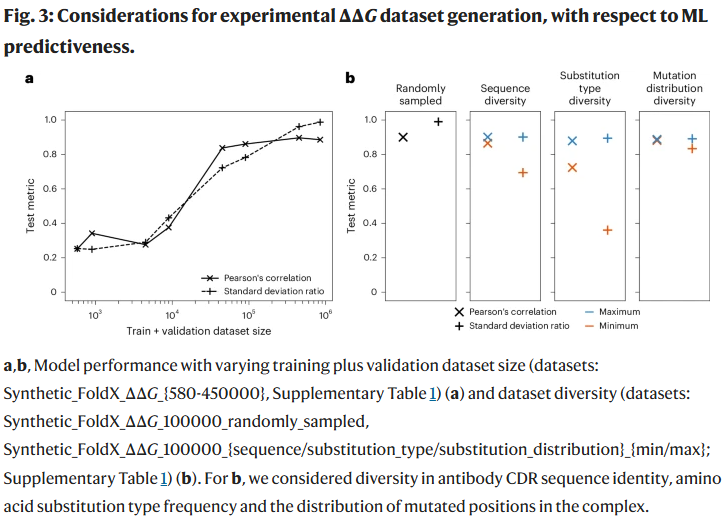
为系统评估数据体量对模型性能的影响,研究人员以合成 FoldX 数据为基础构建了多个不同规模的数据子集(从580至45万条不等),并在统一的测试集(94,126个突变)上评估其性能。
结果显示:
Pearson 相关性在数据量达到 90,000 条后趋于平台期,最高可达 0.85;
标准差比值(真实值与预测值方差之比)在数据量达到 45 万时才接近理想值 1.0,说明较小数据集虽能获得较高相关性,但预测值分布较窄,低估了极端样本;
使用另一种合成工具 Rosetta Flex ddG 构建的 2 万样本数据集,验证了趋势的稳健性,即更大规模的数据显著提升泛化能力;
这些分析表明,当前公开实验 ΔΔG 数据体量远远不足,若希望实现真正可泛化的预测,可能至少需 9 万条实验突变数据。
数据多样性显著影响模型性能
除了数据量,研究人员还系统分析了以下三种多样性对模型的影响:
抗体序列多样性(CDR序列聚类数)
氨基酸突变类型多样性(如Arg→Lys, Ala→Phe等)
突变位点在界面区域的空间分布多样性
研究人员构建了多样性“最大”与“最小”的数据子集(每组10万条),发现:
抗体序列与突变类型的多样性对性能影响最显著。最低多样性数据集的标准差比值分别比最高多样性集低 23% 和 60%;
位点分布多样性对性能影响较小,可能由于模型仅使用突变邻域信息。
这说明:即使数据量相同,缺乏多样性也会显著限制模型的泛化能力,强调未来数据采集策略应注重样本分布的广度。
Graphinity 对标签噪声具备一定鲁棒性
由于真实实验数据常伴随测定误差与实验条件差异,研究人员对合成数据引入了两种类型的噪声:
标签打乱(shuffling)
高斯随机噪声(Gaussian noise)
测试结果显示:
在60%标签被打乱的情况下,Pearson相关性仍可维持在0.85左右,但预测值分布变得集中,模型逐渐丧失识别能力;
当噪声标准差控制在实验数据常见范围内(如0.2–1 kcal/mol),模型性能几乎不受影响;
即便引入高达5的高斯噪声,模型仍可保持约0.8的相关性,说明 Graphinity 具备较强抗噪性;
在SKEMPI中重复突变样本间的标准差为0.19–0.9 kcal/mol,说明Graphinity 的鲁棒性已达实验测定误差范围。
模型能学习特定氨基酸突变的结构趋势
研究人员进一步分析了模型对特定氨基酸替换(如Gly→Phe、Arg→Lys等)的预测能力。结果显示:
模型能较好重现不同突变类型对应的 ΔΔG 均值和标准差分布;
替换为大体积残基(如Trp、Phe、Tyr)往往导致结合力下降,且模型预测更加准确;
替换为小体积或柔性残基(如Gly→Ala)则更难建模,建议未来实验应重点采集这些难预测突变。
整理 | WJM
参考资料
抗原结合亲和力对于抗体功能与药效至关重要,但其计算预测极具挑战性。虽然已有模型在少量实验数据上取得较高性能,但多数因过拟合训练数据而无法泛化至不同抗原或不同抗体–抗原组合。
为探索是否可通过增加数据量实现稳健预测,研究人员构建了近百万条FoldX生成的ΔΔG合成数据,并评估多种结构与序列输入模型。结果表明,图神经网络(如Graphinity)在数据充足条件下具备较强的泛化能力,其性能在抗体与抗原序列差异较大、标签存在实验级别噪声时依然稳定。
不过,应注意这些合成数据来自单一工具(FoldX),相比实验数据更一致、噪声更低,预测值分布可能不同。但FoldX对影响显著的突变仍具合理性,说明其在训练阶段仍具信号价值。
此外,研究人员在 trastuzumab 大规模突变体数据集上的验证说明,Graphinity 并非只能学习合成数据,而确实能够识别实验分布中的结合规律。
本研究表明,实现可泛化ΔΔG预测的最大障碍在于实验数据不足,而非模型架构本身。目前公开实验数据量(如SKEMPI 2.0)远不足以训练高性能模型。根据EGNN与Equiformer等模型在合成数据上的学习曲线,研究人员初步估算:若希望测试集相关性超过0.85,至少需要约9万条突变的ΔΔG值。
这种数据规模在实验通量提升后是可达成的。但具体所需样本量仍受数据多样性与模型类型影响,因此未来也可通过模型优化(如小样本学习、迁移学习、策略采样)进一步降低需求。
除数据量外,数据多样性(特别是抗体序列与突变类型)同样关键。目前实验数据存在明显偏倚,例如SKEMPI 2.0 中多数突变为Ala替换,且来源于不到50个复合物,限制了泛化能力。
因此,研究人员建议朝着“机器学习适用级别”(machine learning-grade)的数据体系发展,未来应聚焦于以下几个方面:
提升实验亲和力测定通量;
设计结构多样、均衡的数据集;
建立标准化、持续更新的ΔΔG与元数据数据库;
实施盲测评估机制,系统验证模型稳健性。
通过这些努力,抗体–抗原亲和力的计算预测有望迈向可泛化、可落地的阶段,为抗体药物的理性设计注入更多确定性。
整理 | WJM
参考资料
Hummer, A.M., Schneider, C., Chinery, L. et al. Investigating the volume and diversity of data needed for generalizable antibody–antigen ΔΔG prediction. Nat Comput Sci (2025).
https://doi.org/10.1038/s43588-025-00823-8
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢