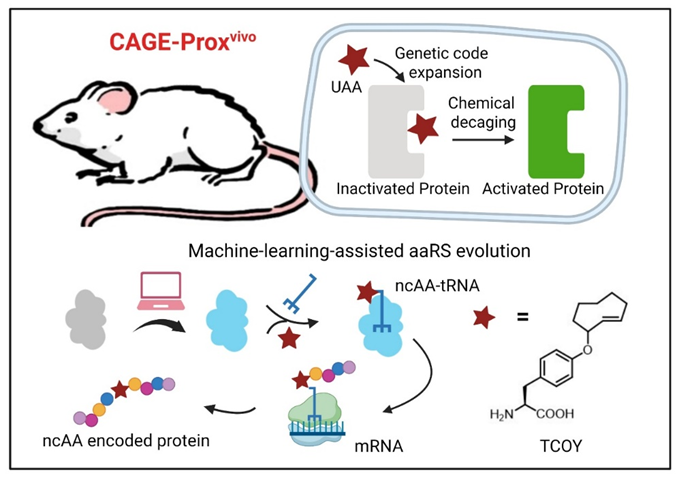
近日,本课题组与北京大学化学与分子工程学院、北大清华生命科学联合中心陈鹏课题组合作,在Cell杂志上发表题为“Machine-learning-assisted universal protein activation in living mice”的研究论文,通过人工智能与蛋白质结构建模辅助开发了一种在活体动物内通用的蛋白质特异-可控激活技术CAGE-Proxvivo。

缘起
六年前,两个课题组就在Nature杂志合作发表了基于“邻近脱笼”理念的蛋白质原位激活技术CAGE-Prox,能以高时间和空间分辨率激活目标蛋白质的酶活性,为动态解析蛋白质作用机制提供了全新策略。但由于依赖紫外光照,该方法存在组织穿透力不足等缺点,应用范围受到限制。
升级
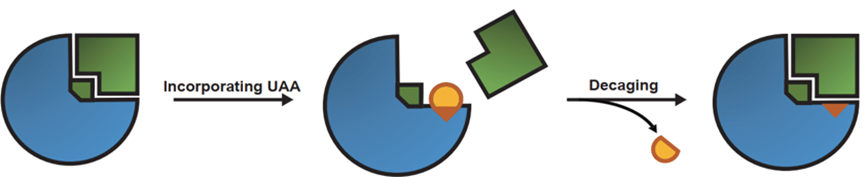
于是我们想问,“邻近脱笼”是否适用于其它的蛋白质功能调控场景?紫外光控脱笼能否改进为应用范围更广的化学脱笼?
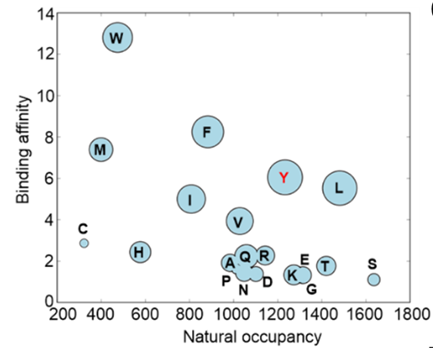
答案是可以!通过统计分析,我们发现酪氨酸除了在蛋白-小分子结合口袋之外,在蛋白-蛋白的界面也具有较高的丰度,基于酪氨酸类似物的非天然氨基酸ONBY,在蛋白-蛋白互作界面有良好的调控表现,经过简单的流程改造,成功将CAGE-Prox技术扩展到蛋白-蛋白互作场景。
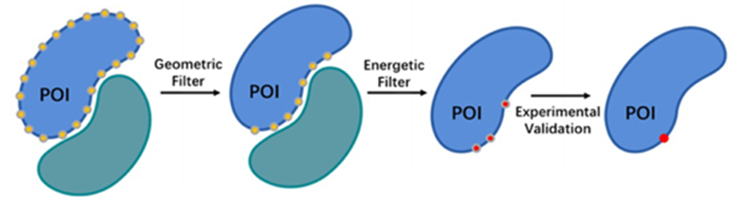
为了进一步实现化学脱笼,我们合成了可以在活体内被小分子正交脱笼的非天然氨基酸TCOY,并希望把它遗传编码到目标蛋白中。但遗憾的是,已知的上百个tRNA合成酶(PylRS)突变体都无法识别这个新型的非天然氨基酸。尝试使用基于结构的蛋白质与小分子建模方案Funclib对PylRS与TCOY的复合物进行能量优化,筛选结合更强的突变体,后续实验验证也都没有成功。我们通过对已知的PylRS突变体与对应的底物建模发现,可以识别的配对在总结合能上并没有明显的优势。因此我们猜测:过度优化蛋白与底物间的作用可能适得其反,PylRS由于无法释放产物而被抑制。
正在研究陷入困境时,基于大量天然蛋白质序列的“蛋白语言模型”随着人工智能领域的技术突破取得了重要进展,各类蛋白语言模型如UniRep、ESM等(注:此时为2021年左右,后续又出现很多新模型)的出现,展示出用基于序列的语言模型可以很好地完成小样本甚至零样本的下游预测任务,这提示我们语言模型可以学到与酶活性相关的关键信息,只是这些信息与底物类型无关,难以直接迁移到全新的底物类型。于是我们采取了一种简单粗暴的整合策略,把基于结构的各种能量信息和基于序列的编码以及打分信息拼接在一起,衔接传统的监督学习。

监督学习最重要的是训练数据。我们挑选了若干已经报道过可以被PylRS或其突变体识别的类似酪氨酸的非天然氨基酸,用已有的上百个PylRS突变体库进行识别效力的全面表征,整理得到近1000对突变体与底物识别效力的实验数据。调用自动化机器学习库AutoGluon,训练出一个简单的监督学习模型,可以对任意PylRS突变体和底物的配对进行打分。我们以可以识别ONBY的突变体为起点,挑选了9个保守较低的易变位点,把每个位置突变成其它可能的19种氨基酸类型,共得到171种可能的单突变体。用机器学习模型进行打分和排序,挑选排名前12的突变体进行实验验证,成功地验证出两个具有识别能力的突变体。

为了进一步挑战模型的边界,我们又合成了半胱氨酸类似的TCOC,直接采用前述单突变体库进行预测,没有验证出有效的突变体。经过观察,我们发现TCOC的侧链结构与TCOK具有更大的相似性,于是重新选择可以识别TCOK的PylRS突变体作为起点,重新构造了171个单突变,这次在排名靠前的突变体中,再次成功验证出两个具有识别能力的突变体。
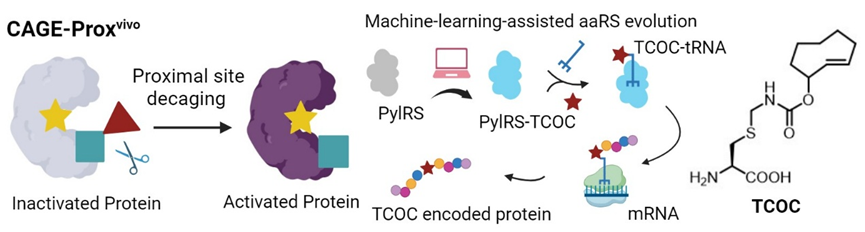
由此看来,虽然该机器学习的训练集是针对酪氨酸类似物构建的,但目前的结果显示该机器学习模型也具有一定的推广能力,这可能来自于两点:蛋白语言模型偏好于保持酶基本活性的信息,而能量模型可以排除破坏与底物结合的突变。如果未来可以进一步增加训练集中非天然氨基酸的类型,增加训练集数量,并引入更先进的机器学习模型训练方式(如Lora)以及更先进的突变体优化策略(如贝叶斯优化),本方法具有拓展到更广泛的非天然氨基酸识别场景的潜力。(延伸阅读:机器学习辅助酶工程)
应用
在建立CAGE-Proxvivo标准流程后,我们首先将炭疽毒素致死因子(LF)设计为可化学激活的"蛋白质前药"(LF-Y659TCOY),并利用表皮生长因子(EGF)修饰的保护抗原(PA)进行EGFR靶向递送。实验证实,该系统能选择性杀伤EGFR高表达的A431肿瘤细胞,而对EGFR缺陷的A375细胞无效。

在动物模型中,通过注射Me2Tz化学激活肿瘤部位的LF前药,我们成功实现了MEK3激酶的特异性切割,诱导癌细胞凋亡并显著抑制实体瘤生长。该研究首次在活体动物中实现了蛋白质药物的时空特异性激活与靶向治疗,为精准癌症治疗提供了新策略。

接着利用CAGE-Proxvivo系统,我们在活体动物中实现了肿瘤特异性焦亡(pyroptosis)的可控诱导。焦亡是一种由Caspase-3介导的炎症性细胞死亡方式,能释放免疫刺激信号,激活抗肿瘤免疫应答。尽管传统化疗可在正常组织中诱导焦亡,但由于焦亡关键蛋白GSDME在肿瘤细胞中表达较低,实现肿瘤特异性焦亡诱导仍具挑战。通过EGFR靶向递送系统,将可化学激活的炭疽致死因子(LF)前药递送至EGFR高表达的小鼠肺癌细胞(LLC),随后注射Me2Tz触发LF活化。实验证实,LF激活后诱导了Caspase-3依赖性GSDME切割,成功在肿瘤内引发焦亡,并观察到外周血中T细胞增加和肿瘤浸润淋巴细胞(TILs)增多,表明免疫系统被激活。更重要的是,该策略不仅抑制了原发肿瘤,还显著延缓了继发肿瘤的生长,证明焦亡具有免疫记忆效应。此外,结合DNA甲基化抑制剂恢复肿瘤细胞中GSDME的表达,该技术可拓展至更多癌症类型。

最后,我们开发了一种生物正交"门控"双特异性抗体系统,实现了T细胞活性的精准时空调控,为肿瘤免疫治疗提供了更安全的策略。传统T细胞接合器(T cell engager, TCE)疗法虽能有效激活T细胞杀伤肿瘤,但常因系统性免疫激活导致严重的细胞因子释放综合征(CRS)等副作用。为解决这一问题,我们设计条件性激活能力的“门控”型TCE:通过遗传编码将TCOY插入抗CD3抗体(aCD3)的关键结合位点(Y59),并连接HER2靶向模块,构建了ZHER2-aCD3-Y59TCOY双特异性抗体。实验表明该修饰几乎完全阻断了TCE与CD3的结合,而加入Me2Tz后可高效恢复其活性(荧光恢复率>90%)。体外实验证实,该"门控"TCE能选择性结合HER2高表达的MDA-MB-231肿瘤细胞,但不会激活外周血单核细胞(PBMCs),仅在化学解锁后才引发IL-2/IFN-γ释放和T细胞介导的肿瘤杀伤。

通过活体生物正交剪切技术恢复T细胞活性,精确激活肿瘤部位的T细胞,有效避免了系统性细胞因子释放综合征及其引发的毒性反应,提高了治疗安全性,为在实体瘤的免疫治疗中,将免疫原性低的“冷”肿瘤转变为高免疫原性的“热”肿瘤提供了新的途径。
总的来说,本研究开发的CAGE-Proxvivo技术平台,通过结合机器学习与生物正交化学反应,实现了活体水平蛋白质功能的精准时空调控。该技术能够在活体环境中原位激活目标蛋白,精确调控蛋白质相互作用网络,从而诱导特定细胞表型变化,如肿瘤细胞焦亡,进而增强抗肿瘤免疫应答。这一突破性进展将生物正交反应的应用范围拓展至任意蛋白质的活体调控领域,不仅为动态生物学研究提供了有力工具,也为发展精准可控的治疗策略开辟了新途径。
北京大学前沿交叉学科研究院北大-清华生命科学联合中心毕业生汪欣博士(现就职深圳湾实验室)、北京大学化学与分子工程学院专职副研究员刘源博士为论文共同第一作者,陈鹏教授和王初教授为本文的共同通讯作者。本工作获得科技部、基金委、北京分子科学国家研究中心、北京大学AI4S专项和新基石基金会的支持。
本文作者:LY
原文链接:
https://www.cell.com/cell/abstract/S0092-8674(25)00517-3
DOI: 10.1016/j.cell.2025.05.006
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢