理解和设计生物分子是推动药物发现、合成生物学和酶工程发展的核心。近年来,人工智能在生物分子预测与设计方面取得了革命性突破,大幅提升了准确性与效率。然而,尽管已有显著进展,一个关键挑战仍然存在:如何用自然语言有效地理解生物分子,并根据人类意图对其进行设计。这暴露出当前AI计算能力与研究人员实际应用需求之间的差距。
为解决上述问题,浙江大学陈华钧、张强、丁科炎等人合作,于2025年7月21日,在Nature Machine Intelligence发表研究论文Advancing biomolecular understanding and design following human instructions。

文章提出了InstructBioMol,一种专为生物分子任务构建的大型语言模型,能够实现自然语言与分子、蛋白质之间的全面任意对齐。该模型能够以多模态生物分子为输入,使研究人员能够用自然语言表达设计目标,并输出满足精准生物学需求的生物分子。实验结果显示,该模型能够根据人类指令理解并设计生物分子。具体来说,所生成药物分子的结合亲和力提升了10%,设计的酶在酶-底物对预测中显著超过当前公认的性能阈值,显示出其在真实生物分子研究中具有变革性潜力。
介绍
尽管人工智能已极大推动了生物分子研究,但当前的AI工具仍难以有效对齐复杂的分子信息与研究人员通过自然语言表达的设计意图,这限制了其在真实科研场景中的广泛应用。要充分释放AI在生物分子科学中的潜力,迫切需要一种能够无缝连接生物分子数据与人类意图的系统。这样的系统应允许研究人员通过自然语言表达设计目标,并获得满足精确生物学需求的分子输出。实现AI与人类专业知识和直觉的深度对齐,对于推动那些依赖创造力和领域经验的研究方向至关重要。
然而,构建这样一个系统面临三项关键挑战:
(1) 专业知识不足:通用LLM对生物分子领域理解有限,难以准确解析专业术语和语义。
(2) 多模态数据整合困难:生物分子本质上是多模态的,例如小分子可通过序列、二维图结构或三维结构表示,这对以文本为主的LLMs提出了独特挑战。
(3) 人类意图难以对齐:通用LLM难以直接将人类自然语言中的意图映射到具体的生物分子任务,往往需要借助精心设计的、领域特定的指令进行引导,从而实现对任务的深度理解与精确执行。
尽管近年来已有多项尝试致力于通过大规模指令数据来定制LLM以适应生物分子任务,但仍面临两大主要局限:
(1) 多数模型仅实现自然语言与分子或蛋白质其中之一的对齐,缺乏自然语言、分子与蛋白质三者之间的任意对任意对齐能力。
(2) 在处理多模态生物分子数据方面能力有限,未能实现将这些数据与自然语言有效对齐。
针对上述问题,本研究提出了InstructBioMol,一种专为生物分子任务构建的大型语言模型。InstructBioMol实现了自然语言、分子和蛋白质三者之间的任意对任意对齐,并引入基于motif引导的多模态特征提取模块,有效整合序列、二维拓扑结构和三维几何信息。同时,该模型可作为研究助手,支持如药物发现与酶设计等实际科研应用。InstructBioMol基于一亿级别的生物分子任务指令数据集进行训练,具备出色的指令理解与执行能力。实验结果显示,InstructBioMol在分子与蛋白质任务的理解与设计方面整体表现提升了12%。在为特定靶点蛋白设计药物分子的任务中,其生成的分子在结合亲和力上提升了10%;而在酶设计任务中,其设计的酶在酶-底物预测得分(ESP得分)上达到了70.4,成为唯一超过ESP评分开发者推荐阈值60.0的方法。
InstructBioMol
InstructBioMol的整体框架如图1a所示。它是一个统一的多模态大语言模型,可同时处理自然语言、分子和蛋白质。该模型的输入可以是自然语言,也可以是多模态的分子或蛋白质,输出则可以是自然语言、分子或蛋白质的文本形式。
为了处理分子与蛋白质的多模态数据,研究人员开发了一个基于motif引导的多模态特征提取模块(图1b),其采用Transformer编码器–解码器结构。该模块对分子部分编码其二维图结构和三维结构,对蛋白质部分则编码其一级序列和三维结构。通过使用预训练的轻量化编码器,模型能够将输入数据编码为相应的表示向量,并输入到Transformer编码器中。在Transformer解码器中,模型使用motif prompt提取器挖掘motif中的生物学知识,该信息用于指导多模态特征的融合,融合后的特征最终被整合进语言模型中。
模型训练如图1c所示,使用了两个主要数据集:持续预训练数据集与指令微调数据集。前者包括从科学文献中获取的分子、蛋白质和自然语言文本,后者则是自然语言与分子/蛋白质之间的数据配对。训练分为两个阶段:首先进行持续预训练,增强模型在生物分子科学领域的领域知识;然后进行指令微调,实现自然语言、分子与蛋白质之间的任意对齐。指令微调过程采用分阶段策略:从大规模粗粒度数据学习,再到精细高质量数据学习,逐步提升模型性能。
最终,如图1d所示,InstructBioMol实现了自然语言、分子和蛋白质三者之间的任意对齐,能够胜任广泛的生物分子任务,包括:按照人类意图发现靶向蛋白的小分子药物、设计特定底物的酶等实际挑战。
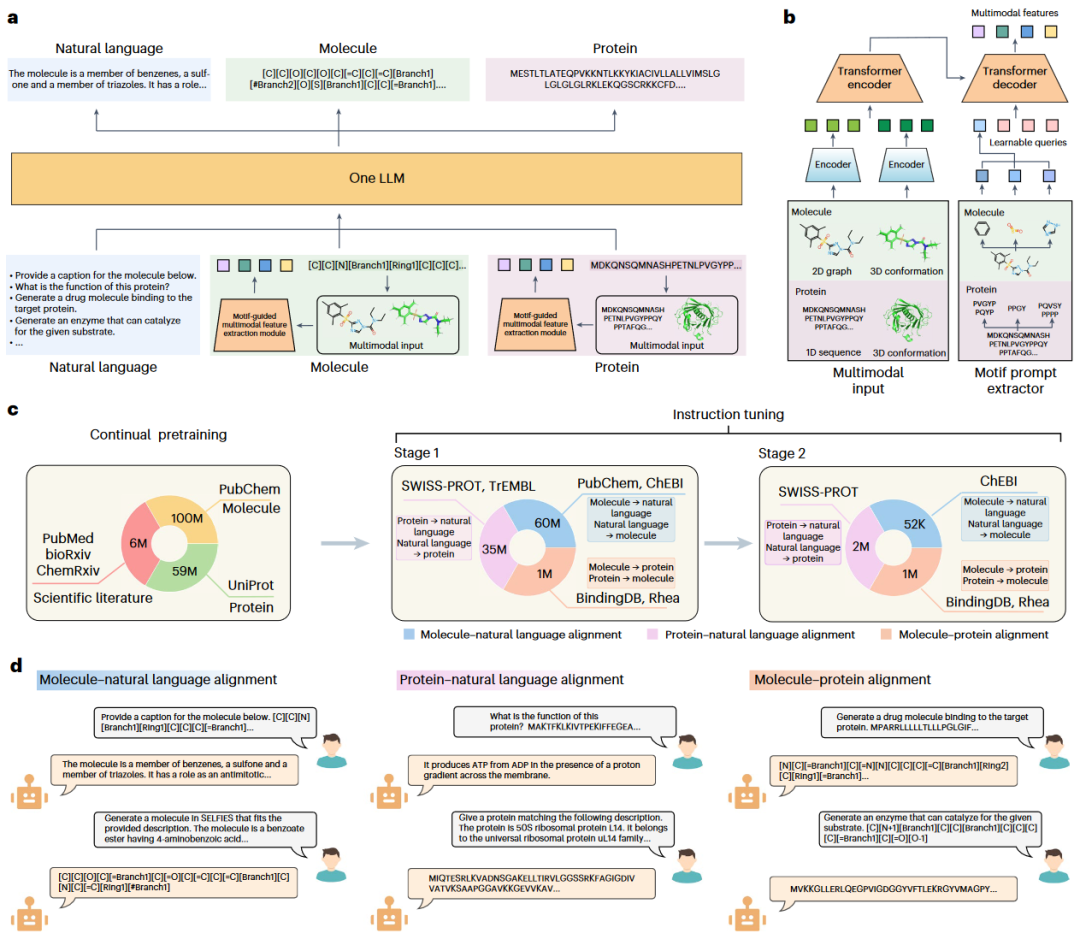
图1 InstructBioMol总体架构
分子设计
实验设置
通过两个任务评估模型在理解和设计分子方面的能力:(1)分子描述任务,即为一个分子生成文本描述;(2)基于描述的分子生成任务,即根据给定文本描述生成一个分子。本实验评估了两类基线模型:(1)通用语言模型:GPT-3.5(zero-shot),以及GPT-3.5(10-shot MolReGPT)和GPT-4(10-shot MolReGPT)。(2)分子任务增强语言模型:ChemDFM、InstructMol、MolT5、BioT5和BioT5+。
实验结果
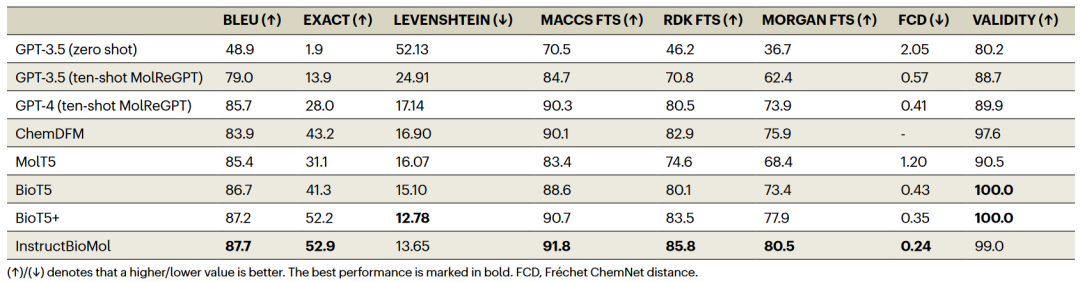
表1和表2分别列出了分子描述任务和分子生成任务的量化评估结果。由于对领域指令数据集进行了微调,分子专用模型明显优于通用语言模型,这表明通用模型缺乏化学领域知识,而这可通过指令微调来弥补。特别地,InstructBioMol在几乎所有评估指标上表现最佳。
表1 分子描述任务性能比较
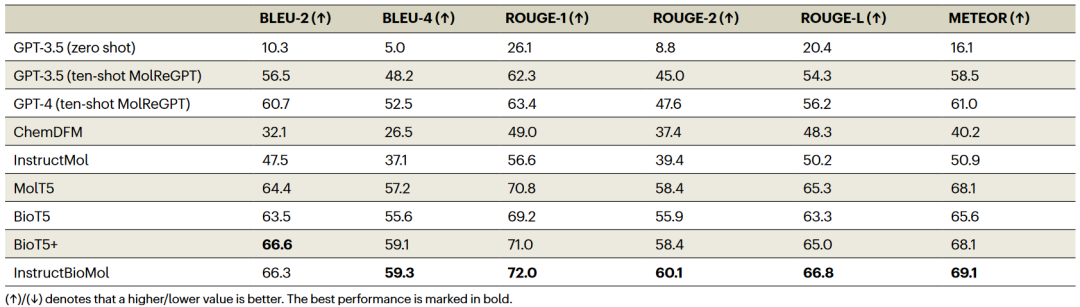
表2 基于描述的分子生成任务性能比较
蛋白设计
实验设置
通过以下两个任务评估该模型在理解与设计蛋白质方面的能力:(1) 回答有关蛋白质属性的问题,包括蛋白质家族、亚细胞定位、官方名称和功能等;(2) 根据文本描述生成蛋白质序列。在实验中,对比了以下基线方法:(1) 通用语言模型GPT-3.5:GPT-3.5(zero shot)、GPT-3.5(five-shot random)和GPT-3.5(five-shot similarity);(2) 蛋白质领域增强语言模型:Mol-Instructions、InstructProtein、BioT5+、ProtT3、BioMedGPT、ProteinDT以及ProteinDT+motif。
实验结果
关于蛋白质属性问答与基于描述的蛋白质生成的定量结果分别展示于图2a和图2b。由实验结果可知,InstructBioMol在两个任务中均表现出最优性能。其次,面向领域的指令对齐效果显著。在蛋白质属性问答任务中,InstructBioMol明显优于GPT-3.5(zero shot);在蛋白质生成任务中,其在序列一致性上提升了17.8%,比对性能提升了27.7%。
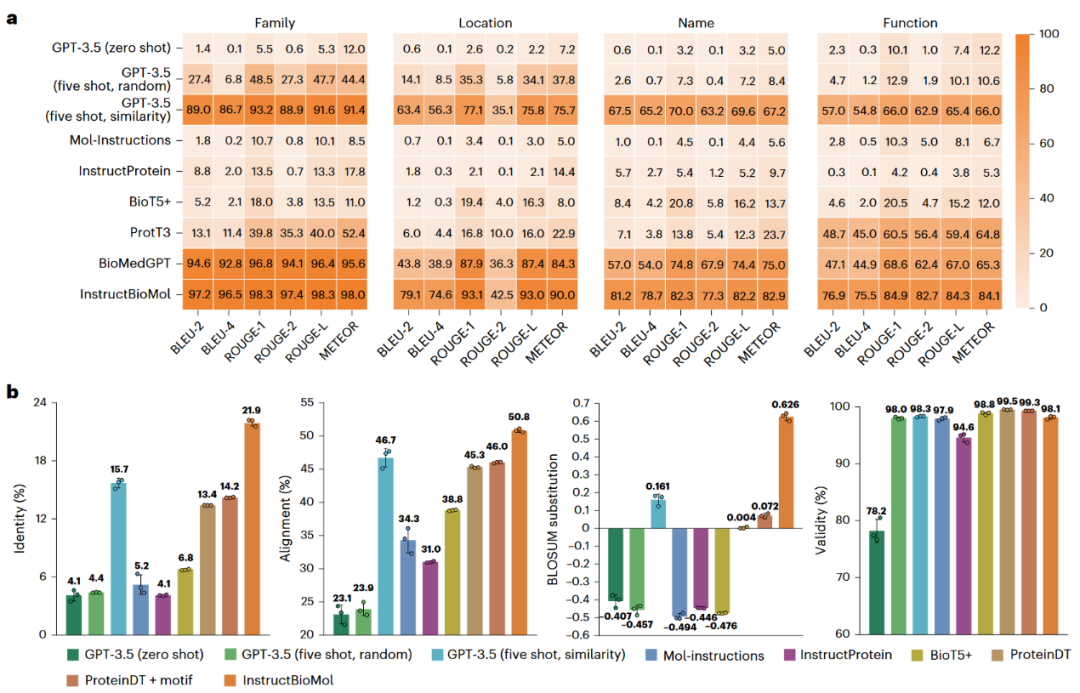
图2 蛋白质理解和设计基准的模型性能
基于靶点蛋白的药物发现
实验设置
InstructBioMol可依据人类意图为靶点蛋白设计候选药物,从而显著减少药物开发所需的时间与成本。为进行对比,引入三种GPT-3.5的变体(zero-shot、five-shot random和five-shot similarit),以及Pocket2Mol、AR-SBDD、TargetDiff和DrugGPT作为基线方法,同时也将测试集中的真实分子作为参考基线。在测试集中选取了100个靶点蛋白,每个靶点生成100个候选分子。
实验结果
图3a展示了实验结果,InstructBioMol在三个关键维度上表现优异:结合亲和力、分子性质和整体评估表现。表明InstructBioMol在生成具备高亲和力和良好药物性质的小分子方面具有更强的能力,且其生成有效率高达99.9%。图3b展示了top-1、top-5、top-10及全部生成分子的平均Vina打分结果。可以明显看出,InstructBioMol在所有设定下均优于其他方法。此外,InstructBioMol是唯一一个其生成分子的平均得分超过参考值的方法,这表明其生成分子的质量可与数据集中的真实分子媲美。InstructBioMol还被证明是在大多数靶点蛋白上生成最佳Vina打分分子的最有效方法。图3c展示了所有生成分子的Vina打分分布情况。分布结果显示,InstructBioMol生成分子的平均打分更低、方差更小,进一步验证了其整体生成质量优于其他方法。
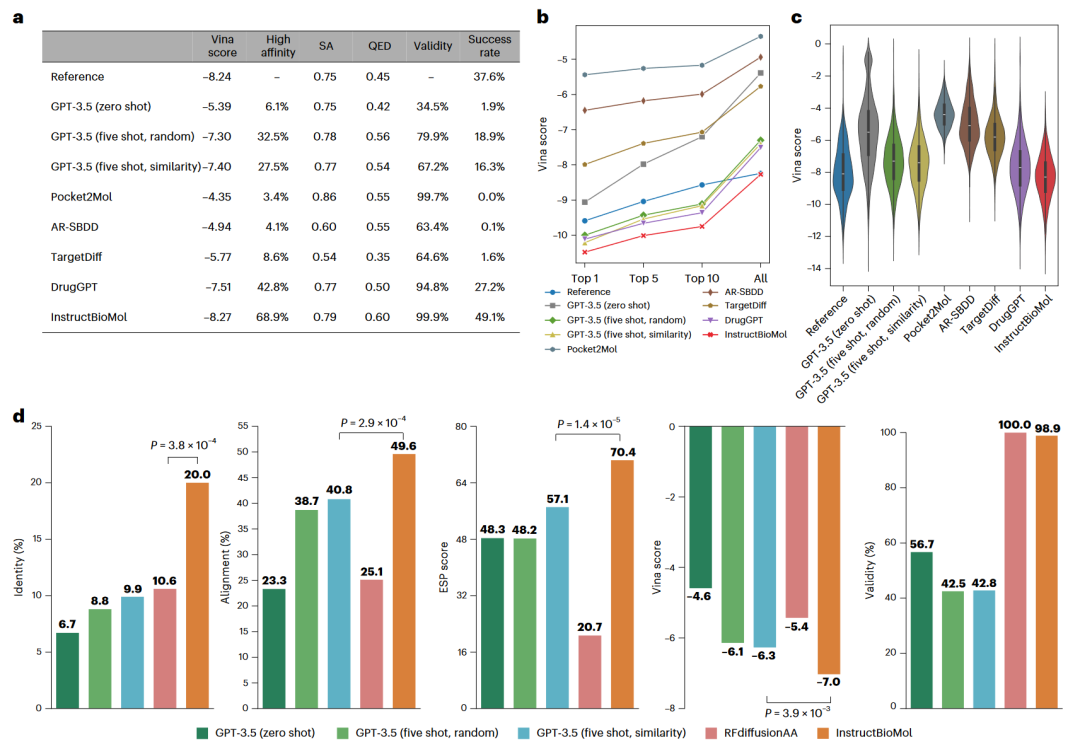
图3 药物发现和酶设计的性能比较
基于底物的靶向酶设计
实验设置
酶通过与特定底物结合并对其进行作用,从而提高底物的转化效率。InstructBioMol能够辅助研究人员针对特定底物设计蛋白酶,从而推动高效酶设计的进展。为了建立实验验证,构建了一个包含100个底物的测试集,并为每个目标底物生成100个酶蛋白。作为对比,我们采用GPT-3.5的三个变体(zero-shot、five-shot-random和 five-shot-similarity)以及RFdiffusionAA5作为基线模型。
实验结果
图3d展示了所生成的蛋白酶在多个评估指标上的性能表现。InstructBioMol在与真实结构的相似性、与底物的相互作用能力及生成有效性等方面均表现出最优性能。值得注意的是,InstructBioMol获得了70.4的ESP得分,是唯一一个超越ESP开发者建议的酶–底物相互作用阈值(60.0)的方法,表明其设计的酶能够以高亲和力结合相应底物。这些结果表明InstructBioMol在生成高效且具特异性的蛋白酶方面具有潜力,可为生物催化等领域提供更有效的解决方案。
基于描述的蛋白质与分子协同设计任务
实验设置
进一步探索该统一模型的优势,通过输入文本描述,同时生成蛋白质和结合配体。划分了训练集和测试集,测试集中包含100条目标蛋白质描述。在评估阶段,对于每条描述,生成100个蛋白质–分子对。本任务中,在InstructBioMol上采用LoRA微调策略。基线模型共分为三组:(1)GPT-3.5,包括GPT-3.5(five-shot random)与 GPT-3.5(five-shot similarity)两个变体;(2)微调后的LLM,Llama7、Mol-Instructions-molecule、Mol-Instructions-protein、BioT5和BioT5+,所有模型均在训练集上进行了微调;(3)顺序集成方法:GPT-3.5(five-shot similarity)+ DrugGPT,结合了从自然语言生成蛋白质与从蛋白质生成分子的两种最具竞争力的技术,首先利用GPT-3.5(five-shot similarity)根据描述生成蛋白质,再使用DrugGPT根据蛋白质生成分子。
实验结果
如图4所示,InstructBioMol在所有评估指标上均优于基线方法。在蛋白质生成方面,其对齐度提升了6.8%;在分子生成方面,Vina打分与合成可达性均表现最优。整体成功率达到48.3%,比基线高出15.2%。此外,还分析了每条描述的top-n(–Alignment×Vina score)结果,该指标越高表示生成的蛋白质与分子质量越优。结果表明,即使在top-20范围内,InstructBioMol的表现仍略优于最强基线的top-1分数,进一步验证了其在协同设计任务中的有效性。
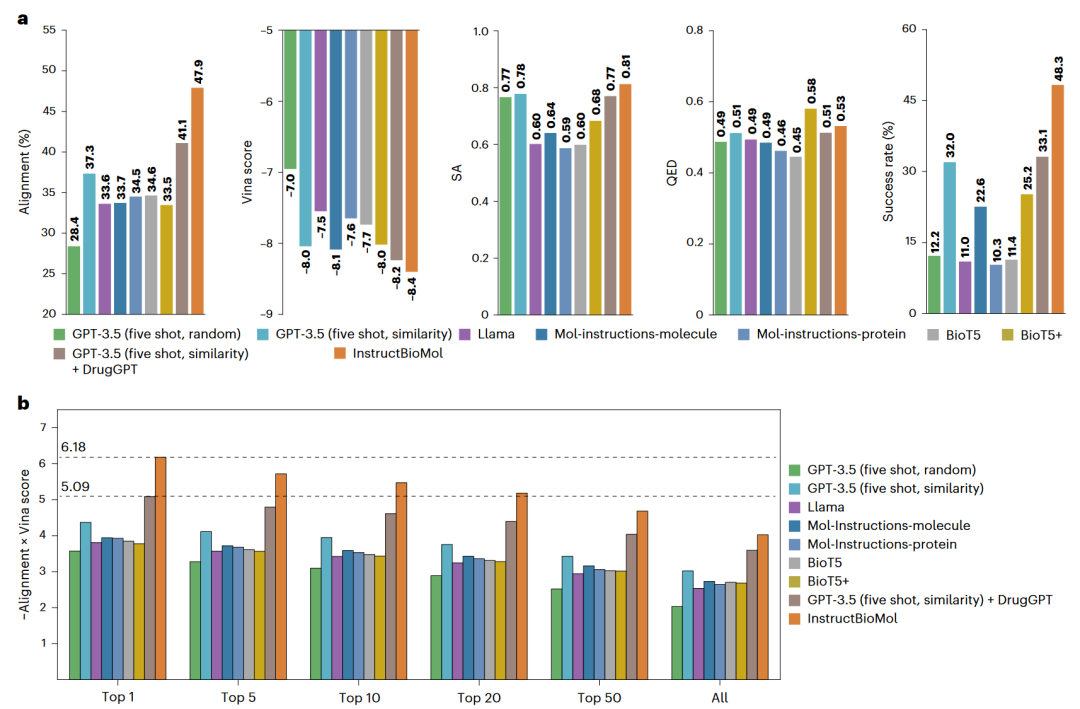
图4 基于描述的蛋白质-分子对生成
此外,观察到基于微调LLM的基线表现整体不佳。虽然Mol-Instructions-molecule、Mol-Instructions-protein与InstructBioMol均基于Llama模型训练,但它们的性能差异显著:Mol-Instructions-molecule仅在文本-分子数据上训练,在分子相关指标上表现良好,但在蛋白质生成方面存在明显不足;Mol-Instructions-protein则仅在文本-蛋白质数据上训练,具备较强的蛋白质生成能力和高对齐度,但在分子相关指标上表现较差。相比之下,InstructBioMol展现出明显的性能优势。这一性能差异清晰地表明,专注于单一模态的模型在同时处理自然语言、分子与蛋白质等多模态任务时存在明显劣势。而InstructBioMol具备自然语言、分子与蛋白质三者之间的任意模态对齐能力,使其能更有效地在不同数据类型之间学习,从而更好地捕捉跨模态的语义关系。
讨论
在本研究中,作者提出InstructBioMol,一个能够遵循人类指令以理解和设计生物分子的多模态大语言模型。为了解决通用LLM无法处理多模态生物分子数据的局限性,作者设计了一个基于motif引导的多模态特征提取模块。在训练过程中,采用了持续预训练和指令微调的训练范式,基于大量的预训练与指令数据进行训练。通过系统性的指令微调,InstructBioMol成为了首个可实现自然语言、分子与蛋白质之间任意双向对齐的模型。实验表明,这种对齐能力在多个涉及自然语言、分子和蛋白质的任务中均表现出色。不仅可以根据人类意图理解和设计分子或蛋白质,还能为靶点蛋白设计类药分子,或为底物设计酶催化剂,显示出作为科研助手的潜力,可为研究人员提供洞见与启发,在药物与酶设计中具有实用价值。
但仍存在以下局限:当前InstructBioMol不支持DNA/RNA等更多类型生物分子,尚未覆盖所有生物分子任务。此外,存在伦理与生物安全风险,需加强人类价值对齐。InstructBioMol的核心价值在于其使用LLM处理生物分子数据的能力,展示了通用智能在多任务统一建模方面的潜力。随着计算资源的提升、训练数据的丰富与伦理对齐机制的增强,InstructBioMol有望不断进化,支持更广泛的任务,推动科学研究中通用人工智能的发展。
参考链接:
https://doi.org/10.1038/s42256-025-01064-0
--------- End ---------
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢