DRUGONE
预训练的大语言模型所蕴含的知识正用于解决各类科学难题,并在液体处理机器人的协助下推动自动化实验室系统的发展。研究人员提出了PrimeGen,一个由大语言模型驱动的多智能体系统,旨在加速靶向下一代测序中的引物设计工作。PrimeGen 以 GPT-4o 为核心控制器,与实验人员进行任务规划与拆解的对话,协调多个专门代理执行特定子任务。这些任务包括:搜索代理用于数据库中检索基因目标;引物代理在多种场景下设计引物序列;实验协议代理通过增强生成和提示工程生成可执行的机器人脚本;实验代理结合视觉语言模型,能检测并报告实验异常。通过实验验证,PrimeGen 可设计高达955个扩增子,确保高扩增均一性并最小化二聚体形成,展示了由通用基础模型协调的协作代理在生物医学研究中的智能化潜力。

自动化实验室正在化学与生物领域迅速发展,能高效探索庞大的参数空间,降低成本,提升实验安全性,同时提高实验重复性与吞吐量。尽管传统自动化系统依赖人工来解读实验结果,但大语言模型凭借其在语言理解、生成与决策方面的能力,正逐步被引入实验自动化。结合检索增强生成(RAG)等技术,大语言模型的响应质量显著提升,并在协议合成、遗传变异合成及微生物筛选中展现出独立任务执行能力。相比于宏基因组或全基因组测序,靶向测序更具成本效益与灵敏性,适用于病毒追踪、病原检测、致病突变识别等。然而,针对不同靶标序列设计高通量PCR引物的复杂性促使研究人员开发更智能的自动化方案。
结果
PrimeGen 架构
PrimeGen 被设计为实验人员的共设计者,以 GPT-4o 为核心,负责理解实验目标、规划任务并分配给特定代理,如搜索代理、引物代理、协议代理与实验代理。每个代理使用模板化提示,独立与用户对话、获取执行子任务所需细节并协调实验执行,同时具备自我反思与优化能力。
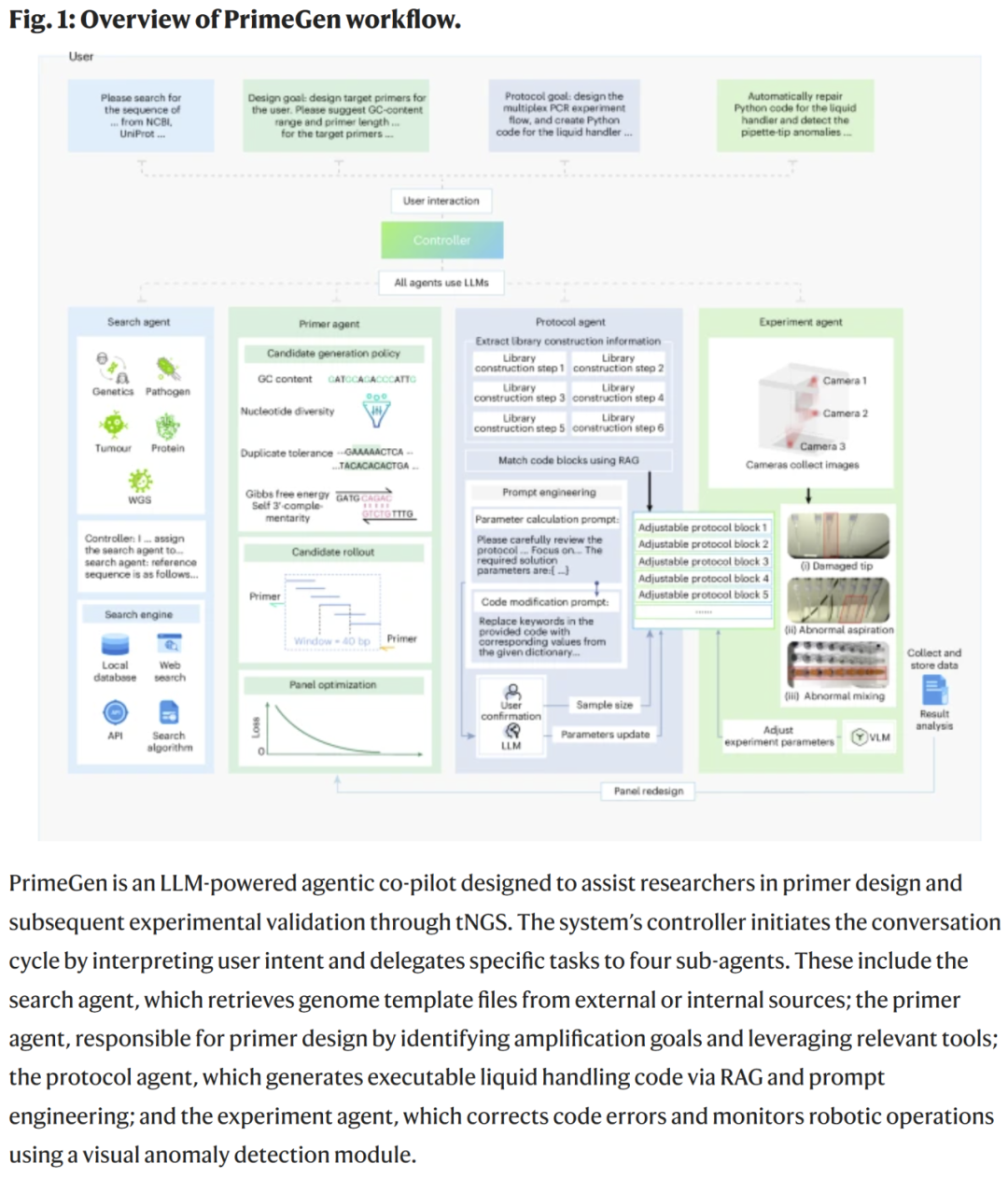
搜索代理
该代理可帮助用户识别PCR模板的潜在靶区,适用于五种常见应用场景:病毒/细菌WGS平铺PCR、癌症或遗传病基因分析、病原检测、SNP关联耐药性分析、酶突变检测等。代理通过调用数据库(如OMIM、ClinVar、NCBI等),提取参考基因组与注释信息。对于SNP相关任务,采用聚类方法确定靶区。搜索流程中可能出现三种情况:用户提供明确目标、目标不确定需多轮对话确认、或直接提供目标文件。若搜索失败,则触发ReAct机制进行推理与行动调整。

引物代理
采用滑动窗口与候选展开策略,在目标区域内搜索合格引物。设计考虑自互补性、Gibbs自由能、GC含量、多样性容忍度等因素。在确保靶区覆盖与扩增均一的同时,还集成SADDLE算法优化多重引物设计,使用损失函数衡量二聚体形成与比对得分。引物筛选通过四种优化方法比较:贪心算法、AdaLead、遗传算法、LLM优化器。结果显示在高维任务中LLM优化器表现更佳。
实验验证
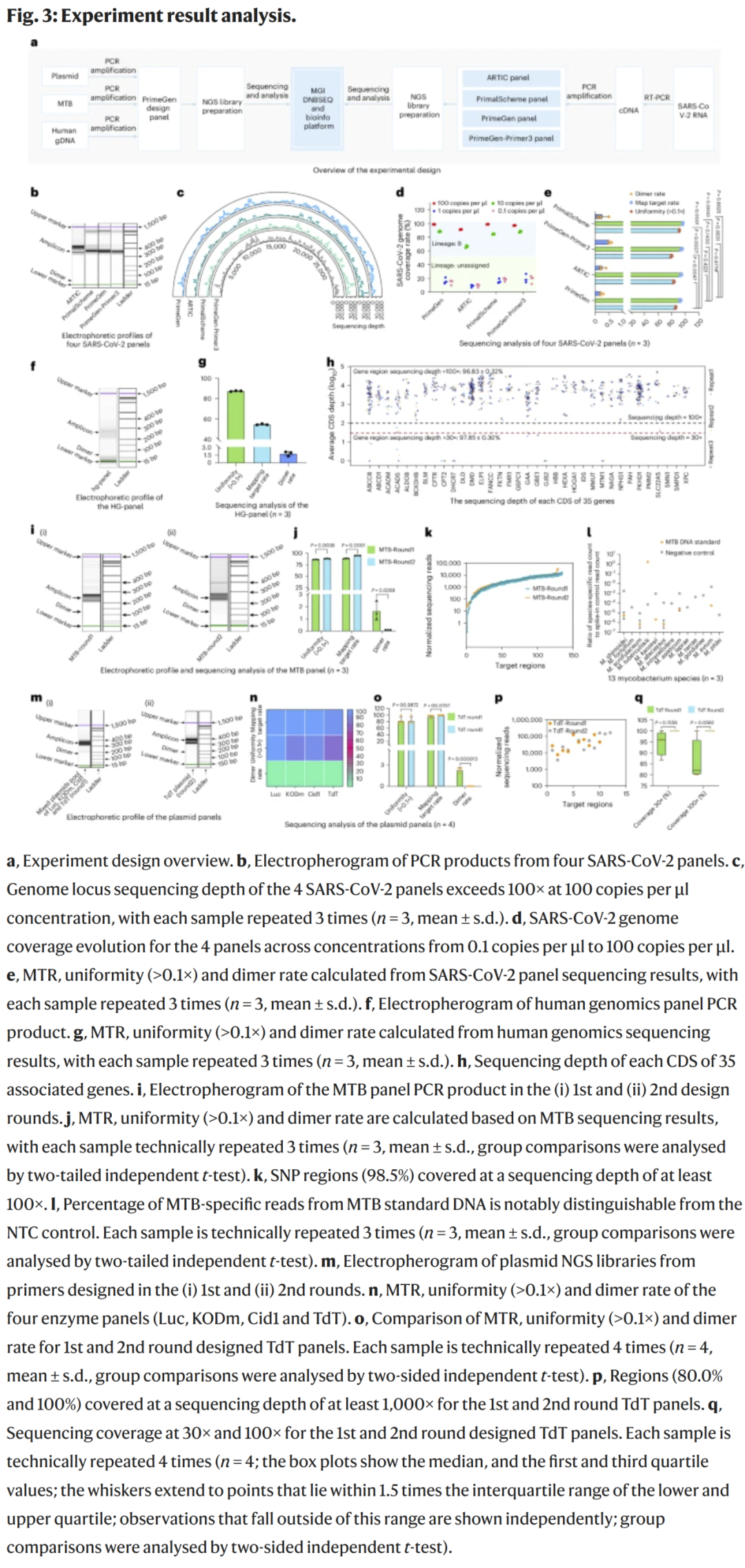
PrimeGen 在四类实验中展示优异表现:
SARS-CoV-2全基因组测序:设计131个扩增子,与ARTIC、Primer3等方法相比,在低浓度下展现更高的覆盖率与均一性;
携带者筛查(ECS):设计覆盖35种严重遗传病的955个扩增子,验证结果显示97.85%编码区深度超过30×;
结核分枝杆菌(MTB)耐药突变检测:目标SNP区域覆盖超过96%,并能区分13种分枝杆菌;
酶突变质粒测序:设计4个关键酶的测序引物,在重设计中将覆盖率从95%提升至100%。
协议代理
PrimeGen 的协议代理可将引物设计结果转换为可执行的实验步骤代码,适配液体处理机器人。其使用28个可调实验模块(APBs),通过RAG机制从PDF实验手册中识别步骤并匹配代码块,进一步通过大语言模型计算实验参数如试剂体积、PCR循环数等,并支持自定义重写。与多种大模型对比,GPT-4o在序列搜索、面板优化与代码修改方面均展现领先性能。

实验代理
该代理结合视觉语言模型(VLM)实现实时监控与异常检测。通过三个摄像头从不同角度监控操作流程,检测吸头损坏、液体处理异常等问题。如发生三次抓取失败或磁珠混合异常,系统将调整参数并重试,最多尝试三次再提示人工干预。VLM通过两阶段微调完成训练,在异常检测任务中,Qwen2-vl达成87%的准确率。

讨论
研究人员提出的PrimeGen展示了LLM驱动的多智能体框架在引物设计与实验自动化中的强大潜力。该系统通过自我反思、API集成、计划执行等机制,有效组织各子任务代理协同工作,推动科学实验流程智能化。然而,该系统仍面临挑战,如复杂任务的自主规划能力不足、搜索代理在复杂基因组目标识别方面存在局限、协议解析仍依赖于人工设计的提示。未来可通过引入自我批判机制、对抗式大模型、结构化数据接口等手段进一步增强其自治能力。
研究人员相信,通过遵守伦理原则、确保系统稳健部署,LLM驱动的智能体系统将在推动科学发现与创新中发挥关键作用。
整理 | DrugOne团队
参考资料
Wang, Y., Hou, Y., Yang, L. et al. Accelerating primer design for amplicon sequencing using large language model-powered agents. Nat. Biomed. Eng (2025).
https://doi.org/10.1038/s41551-025-01455-z
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢