RNA 语言模型通过利用大规模序列数据,在多种下游任务中取得了优异的性能。然而,RNA 的功能本质上由其层次结构决定,将结构信息整合到预训练中至关重要。现有方法往往依赖于噪声较大的结构注释或引入任务特定的偏差,限制了模型的泛化能力。在此,我们引入 structRFM,这是一种结构引导的 RNA 基础模型,通过一种新颖的配对匹配操作将碱基配对相互作用整合到掩码语言建模中,在数百万条 RNA 序列和二级结构数据上进行预训练。结构引导掩码和核苷酸级掩码通过动态掩码比例进一步得到平衡。structRFM 学习序列和结构数据的联合知识,产生多样化的表示,包括分类级、序列级和成对矩阵特征,支持广泛的下游适配。structRFM 在十五种生物语言模型中的零样本同源性分类中位居顶尖模型之列,并在二级结构预测方面设立了新的基准。 structRFM 进一步衍生出 Zfold,该模型能够实现稳健可靠的 tertiary 结构预测,在估计 3D 结构及其相应提取的 2D 结构方面持续改进,与 AlphaFold3 相比,在 RNA Puzzles 数据集上实现了 19% 的性能提升。在内部核糖体进入位点识别等功能任务中,structRFM 在 F1 分数上实现了惊人的 49% 性能提升。这些结果表明了结构引导预训练的有效性,并突出了在计算生物学中开发多模态 RNA 语言模型的 promising 方向。为了支持更广泛的科学界,我们已将包含 2100 万条序列-结构的数据集和预训练的 structRFM 模型完全开源,以促进生物学中多模态基础模型的开发。

作者:Heqin Zhu, Ruifeng Li, Feng Zhang, Fenghe Tang, Tong Ye, Xin Li, Yunjie Gu, Peng Xiong, S. Kevin Zhou
单位:中国科学技术大学、浙江大学、江苏省多模态数字孪生技术重点实验室
日期:2025年8月6日
论文:doi.org/10.1101/2025.08.06.668731
代码:github.com/heqin-zhu/structRFM
📌 背景
RNA在细胞生命活动中发挥着重要作用,例如转录调控、翻译调控、剪接等过程。其功能不仅取决于核苷酸序列,更取决于其层级的二级和三级结构。然而,现有RNA语言模型多基于大规模序列数据进行训练,未充分利用结构信息,导致对结构相关功能的预测能力有限。 已有方法尝试引入结构特征,但往往依赖噪声较大的结构注释数据,或在训练过程中引入任务特定的监督信号,从而牺牲了模型的泛化性和通用性。这一局限性在RNA结构预测、结构导向的功能推断等领域尤为突出。
基于此,本文提出了structRFM,一种完全开源的结构引导 RNA 基础模型,基于结构引导掩码语言建模(SgMLM)策略进行预训练,通过整合序列和结构层面的掩码,隐式地编码碱基配对互作,联合建模结构和序列依赖。structRFM有效利用结构先验,同时保持任务无关的预训练方式,实现对包括零样本预测、二级、三级结构预测及多项功能推断在内的广泛下游任务的强泛化能力 (图 1a)。
🛠 方法
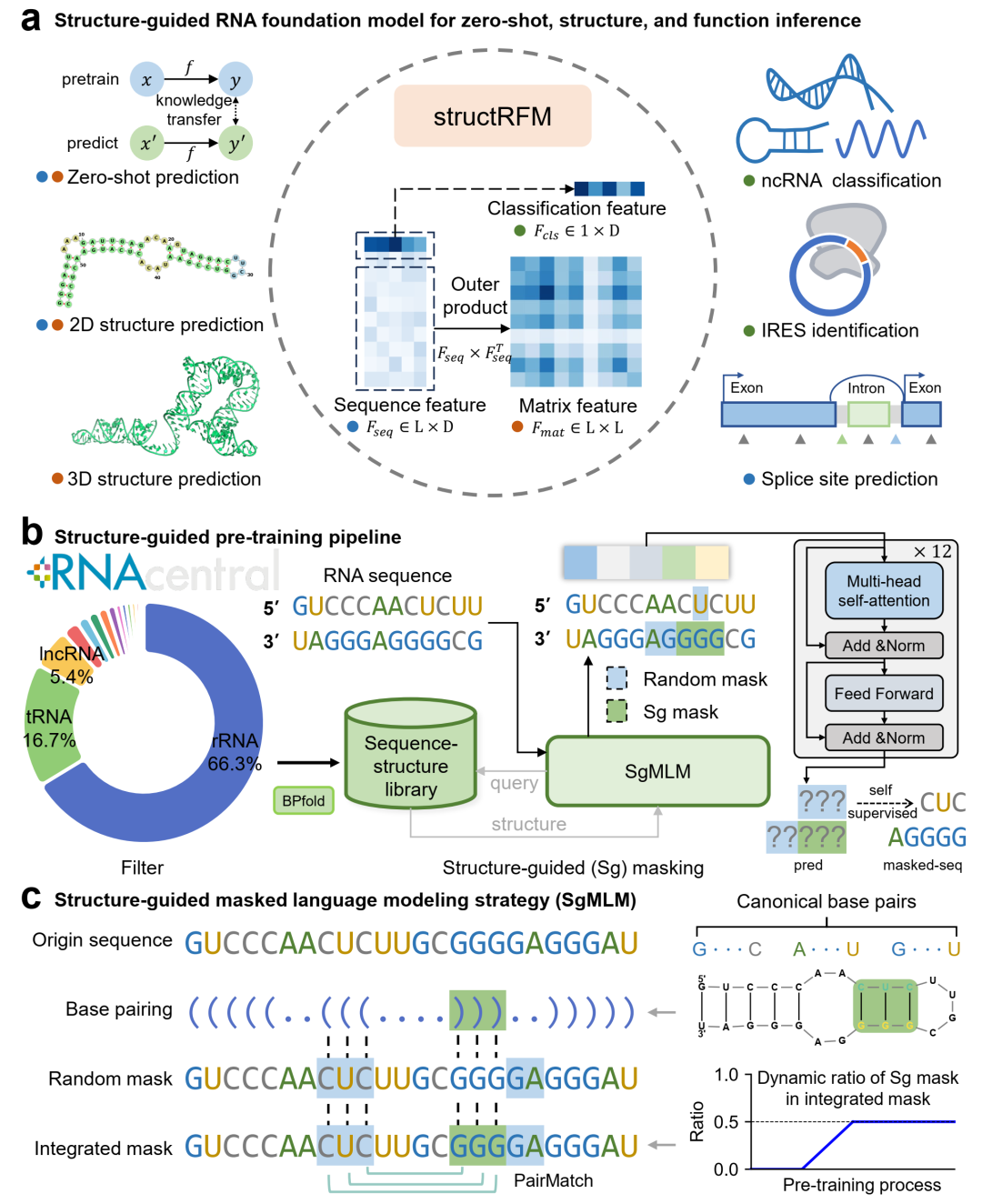
图 1 structRFM 整体框架
核心设计:
2100万条 RNA 序列–二级结构配对数据;
结构引导掩码语言建模(SgMLM):通过创新的 配对匹配机制(PairMatch),优先掩码二级结构中碱基配对位点,引导模型直接建模配对模式;
动态掩码比例策略:训练初期降低结构掩码比例,后期逐渐提高,平衡序列与结构信息的学习;
多层次表征:同时输出分类级、序列级、配对矩阵级特征,支持多种下游任务。
SgMLM 结构引导预训练策略(图 1c),包含两大核心组成:结构引导掩码和动态掩码比例策略。结构引导掩码选择性地掩盖局部结构上下文中的经典碱基对对应的输入 token,促使模型根据邻近的环区恢复碱基配对,从而隐式引导 structRFM 捕捉 RNA 的序列模式及层级结构规律,无需依赖特定任务目标,挖掘多样的核苷酸间关系,促进序列和结构知识向下游结构及功能预测任务的迁移。动态掩码比例策略则在预训练过程中逐步提升结构引导掩码在整体掩码中的比例,使模型焦点从序列级别转向结构信息表示,最终在核苷酸和结构掩码之间达到平衡。
📊 实验结果
表格 1整体实验结果
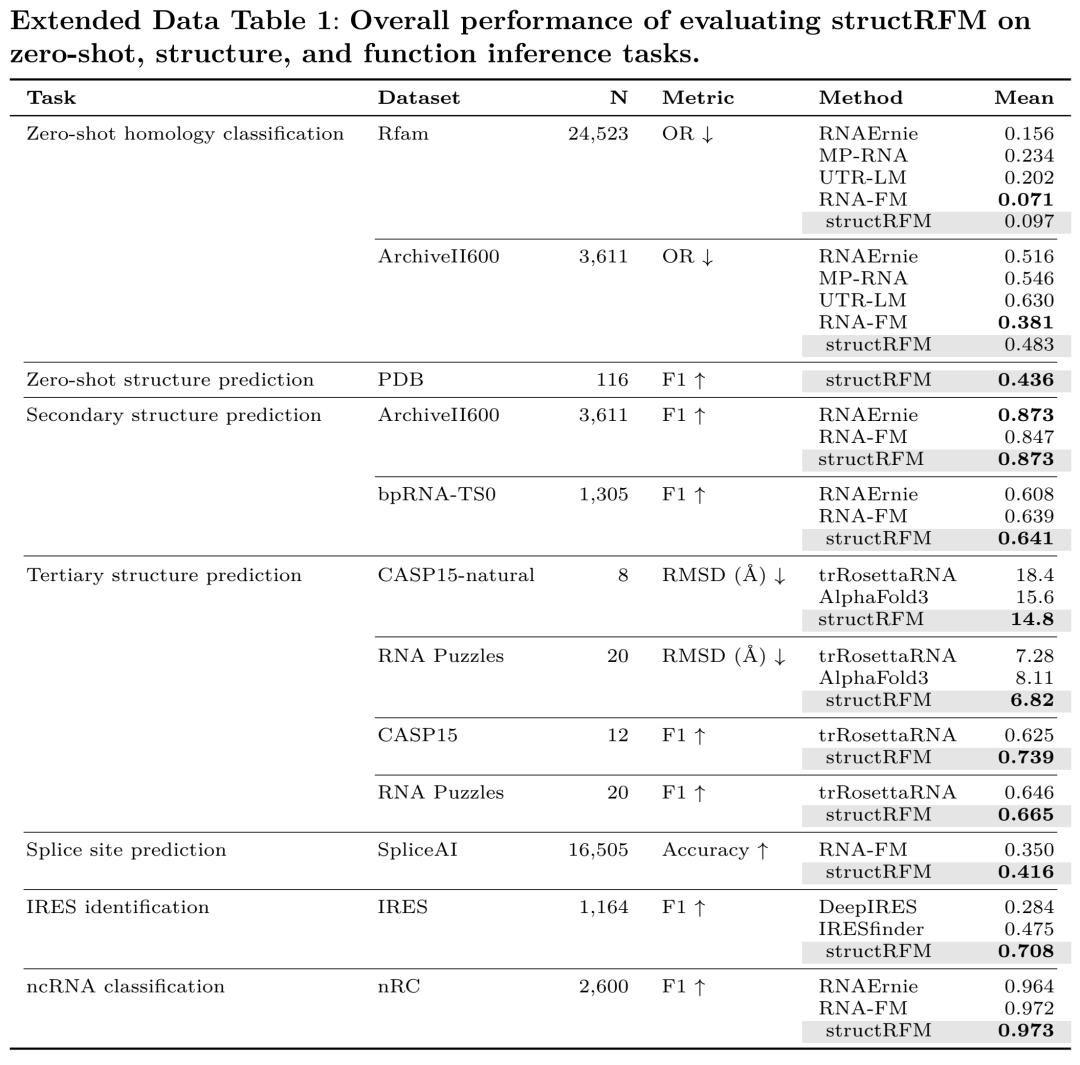
零样本同源分类:15个生物语言模型中排名领先;
二级结构预测:ArchiveII600 F1=0.873,bpRNA-TS0 F1=0.641,均为当前最佳;
三级结构预测(Zfold):在 RNA Puzzles 数据集上 RMSD 比 AlphaFold3 提升 19%;
功能预测:IRES 识别任务 F1 比 IRESfinder 高 49%;
在剪切位点预测、ncRNA 分类等任务中表现同样领先。
1.零样本测试
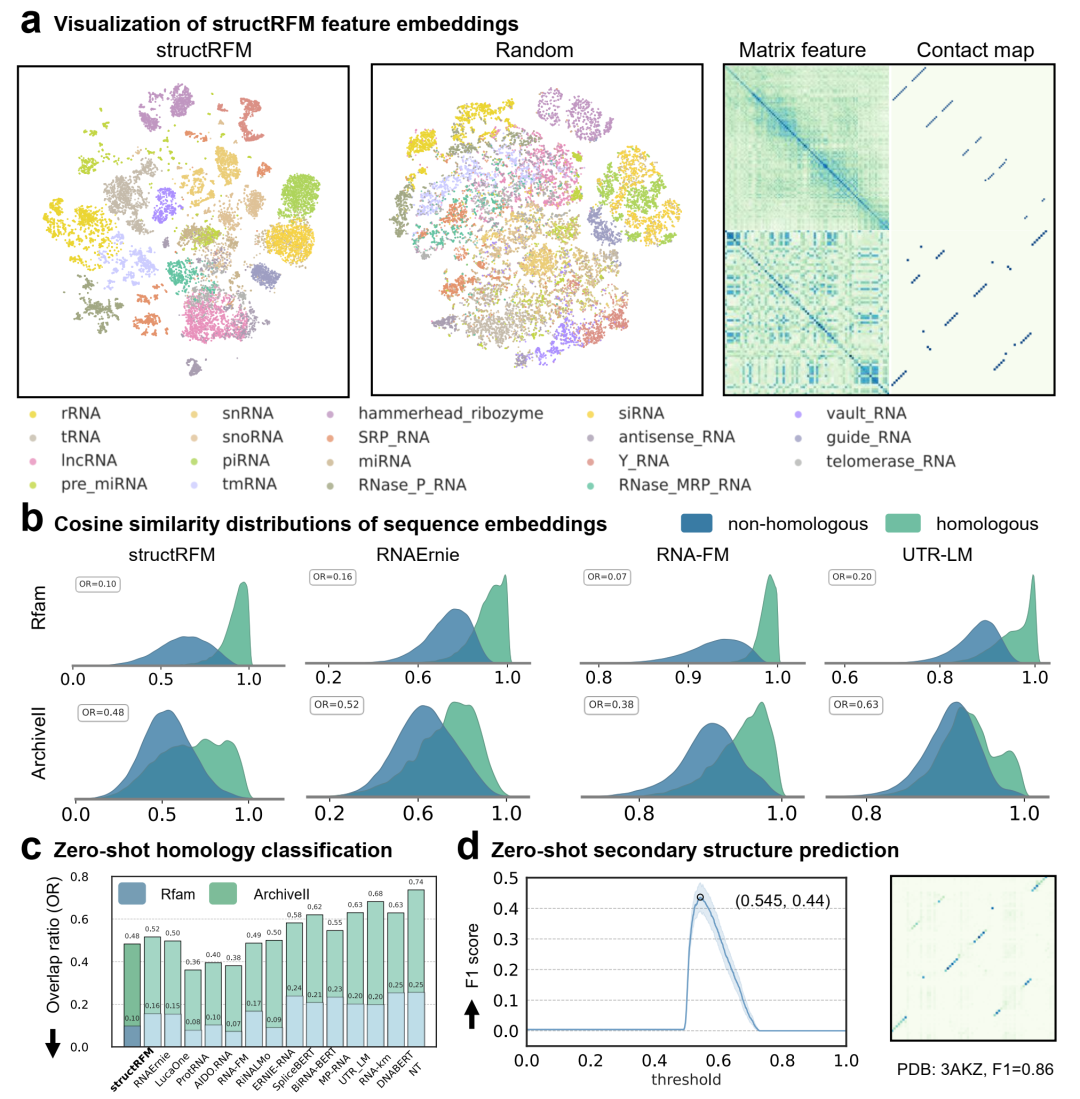
图 2 零样本测试
a 使用 t-SNE 在 RNAcentral 子集(23,994 条 RNA)上可视化 structRFM 与随机基线模型的 RNA 嵌入,显示structRFM 能清晰区分 RNA 家族。并展示了 structRFM 提取的矩阵特征与对应接触图的两个示例。b 在 Rfam(24,523 条 RNA)和 ArchiveII600(3,611 条RNA)数据集上,不同模型同源和非同源 RNA 序列对的余弦相似度分布,及 c两个分布的重叠率(OR),展示 structRFM 出色的同源分类能力。各方法和数据集的均值标注。d structRFM 的零样本二级结构预测,展示阈值从 0 到 1(步长0.001)间的 F1 分数及示例热图。
2.结构预测
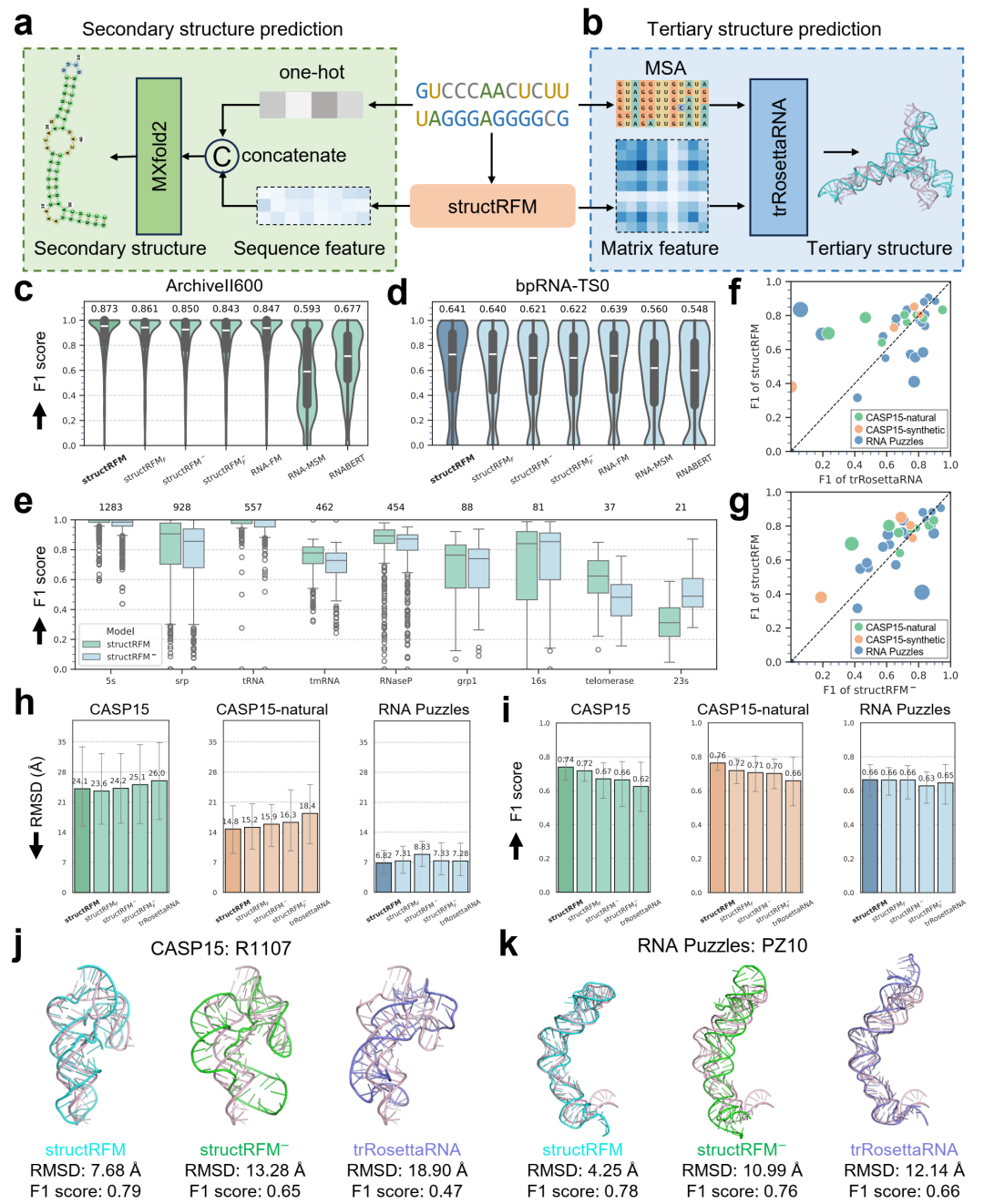
图 3 structRFM 用于结构预测
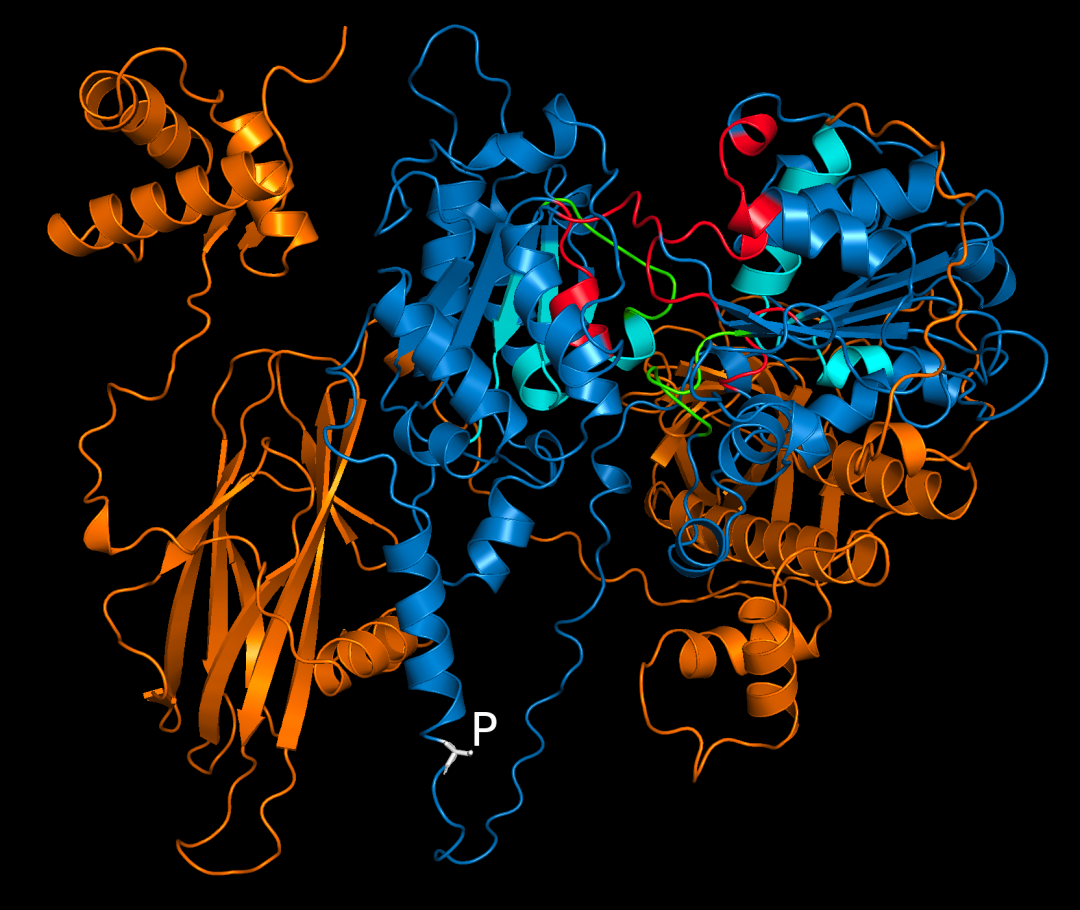
a, b 二级和三级结构预测的神经网络架构。c, d structRFM 及其他模型在 ArchiveII600(3,611 条 RNA)和 bpRNA-TS0(1,305条 RNA)数据集上的二级结构预测 F1 分数。小提琴图中白线为中位数,均值显示于上方。e structRFM 与 structRFM− 在 ArchiveII600 九个家族上的 F1 分数对比。各家族样本数显示于图顶端。f, g 在 CASP15(12 条 RNA)和 RNA Puzzles(20 条 RNA)数据集上,基于预测三级结构提取的二级结构头对头比较。圆圈大小依差值大小而定。h, i CASP15、CASP15-natural 和 RNA Puzzles 三级结构预测的 RMSD 和 F1 分数比较,灰色误差线为 95% 置信区间。j, k CASP15 和RNA Puzzles 上预测三级结构示例,原生结构以粉色展示。
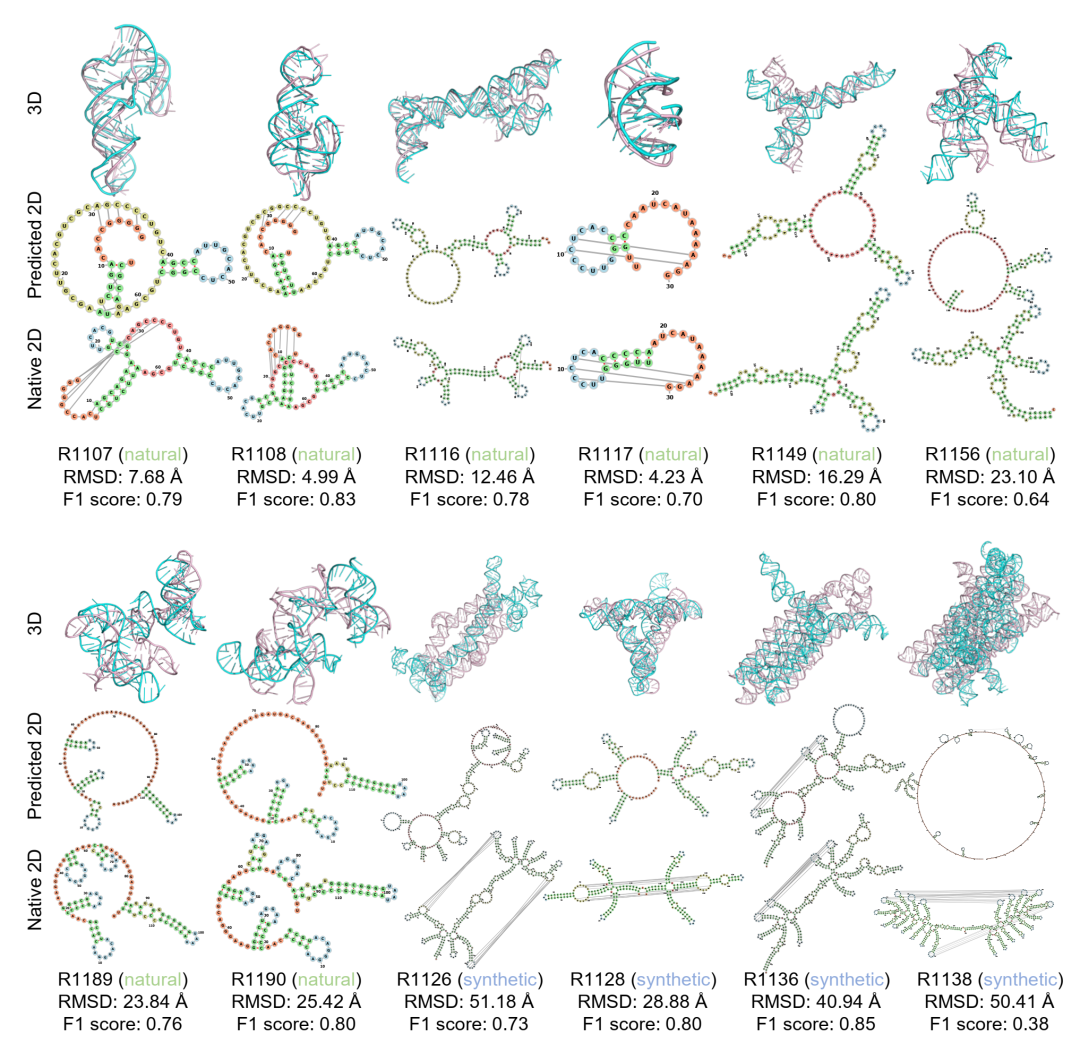
图 4结构预测可视化
structRFM预测结构与 CASP15 数据集原生结构的可视化。列出三级结构 RMSD 及由三级结构提取的二级结构 F1 分数。二级结构中,茎结构为绿色,多重环为红色,内环为黄色,发夹环为蓝色,5’ 和 3’ 非配对区为橙色。总体来看,structRFM 在自然靶标上的表现优于含密集长程交互的合成靶标,但有时未能预测局部短茎(绿色),导致大而错误的多重环(红色)。
3.功能预测
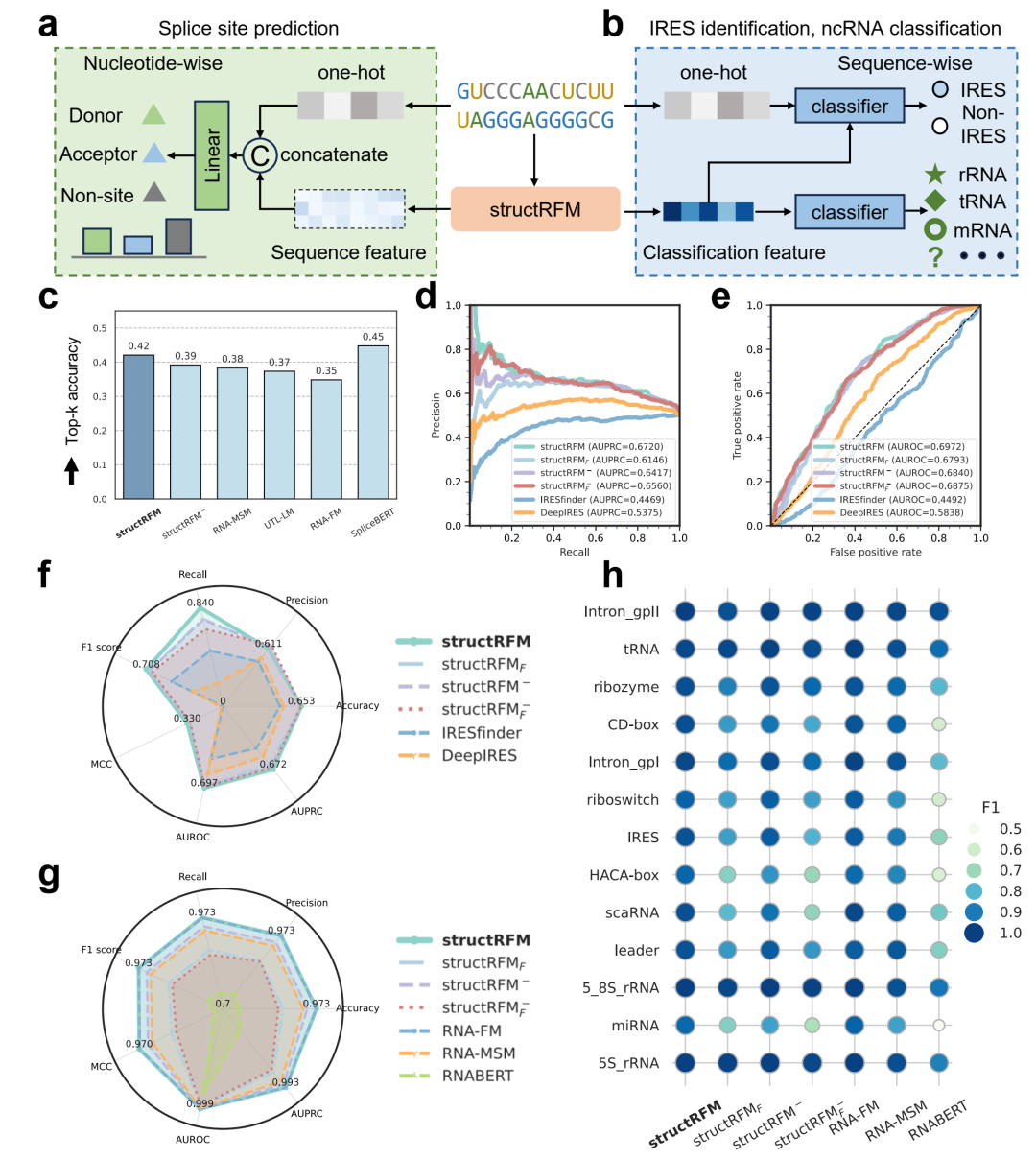
图 5 structRFM 用于功能预测
a, b 用于剪接位点预测、IRES 识别和 RNA序列分类的神经网络结构示意图。c structRFM、其变体及 SpliceBERT 在剪接位点预测任务(16,505 条 RNA)上的 top-k 准确率比较。d, e IRES 识别(1,164 条RNA)的精确率-召回率曲线(PR 曲线)和受试者工作特征曲线(ROC 曲线)。f,g IRES 识别(1,164 条 RNA)及 nRC 数据集(2,600 条 RNA)上的 ncRNA 分类任务七项指标雷达图,展示 structRFM 性能。h nRC 数据集 13 类 ncRNA 上七个模型的 F1 分数比较。structRFM 和 RNA-FM 达到最新的最优性能,明显领先其他模型。
⚠ 局限性
structRFM 存在下面的局限性
最大输入长度为 512 nt。因此,为了处理长序列,简单而有效的方法是冻结编码器主体部分,重新微调一个合适的位置编码。否则需要在长链数据上重新进行预训练。不过,对于序列层面而非碱基层面的任务,可以将长链 RNA 分段处理,然后再汇总。
结构标注来自 BPfold,比较单一,未来可引入热力学和非经典配对信息等信息,集成多种可信的结构信息输入,提高结构信息的准确性和模型的鲁棒性。
动态掩码比例策略可针对不同任务优化。
💡 创新点
1.首次将结构引导掩码策略规模化应用于 RNA 基础模型自监督预训练;
2. 构建迄今最大规模的 RNA 序列–结构配对数据集;
3. 模型、数据、代码全部开源,推动多模态生物基础模型发展;
4. 多任务性能领先,实现跨域泛化。
总结
structRFM 作为一个完全开源的 RNA 基础模型,在下游任务适应上展现出高效与多样性。更重要的是,本文提出的 SgMLM 预训练策略及收集的序列-结构数据集具有良好的扩展性,可无缝整合进其他预训练框架,标志着在赋能RNA 基础模型多模态结构知识、推动更广泛生物学研究方面迈出了重要一步。



内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢